Запрос «Вирус» перенаправляется сюда; см. также другие значения.
| Вирусы | |||||||
|---|---|---|---|---|---|---|---|
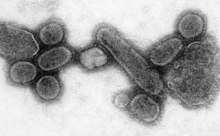
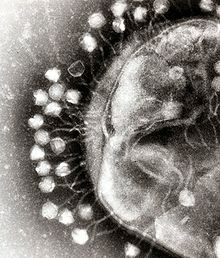
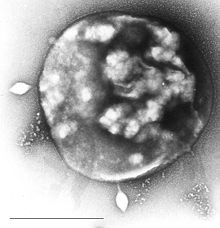
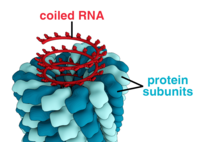
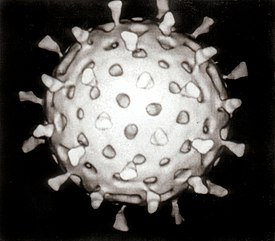 Ротавирус, компьютерная реконструкция на основе данных электронной криомикроскопии |
|||||||
| Научная классификация | |||||||
|
Группа: Вирусы |
|||||||
| Международное научное название | |||||||
| Viruses [комм. 1] | |||||||
| Реалмы[комм. 2] | |||||||
систематика 1 класса (Naldaviricetes) и 15 семейств вирусов не определена (как и 2 семейств вироидов и 2 семейств сателлитов), они не входят в описанные реалмы[1] |
|||||||
|
Ви́рус (лат. virus) — неклеточный инфекционный агент, который может воспроизводиться только внутри клеток[комм. 3]. Вирусы поражают все типы организмов, от растений и животных до бактерий и архей[2] (вирусы бактерий обычно называют бактериофагами). Обнаружены также вирусы, способные реплицироваться только в присутствии других вирусов (вирусы-сателлиты).
Со времени публикации в 1892 году статьи Дмитрия Ивановского, описывающей небактериальный патоген растений табака[3][4], и открытия в 1898 году Мартином Бейеринком вируса табачной мозаики[5] были детально описаны более 6 тысяч видов вирусов[6], хотя предполагают, что их существует более ста миллионов[7]. Вирусы обнаружены почти в каждой экосистеме на Земле, они являются самой многочисленной биологической формой[8][9]. Изучением вирусов занимается наука вирусология, раздел микробиологии.
Геномы вирусов могут быть представлены как ДНК, так и РНК, причем в обоих случаях как одноцепочечной, так и двуцепочечной. Некоторые вирусы способны к обратной транскрипции. Кроме того, у некоторых РНК-содержащих вирусов в репликации участвует рибозим, что сближает их с вироидами. Однако все вирусы, в отличие от вироидов, образуют белковые капсиды, в которые заключён их генетический материал.
У животных вирусные инфекции вызывают иммунный ответ, который чаще всего приводит к уничтожению болезнетворного вируса. Иммунный ответ также можно вызвать вакцинами, дающими активный приобретённый иммунитет против конкретной вирусной инфекции. Однако некоторым вирусам, в том числе вирусу иммунодефицита человека и возбудителям вирусных гепатитов, удаётся избежать иммунного ответа, вызывая хроническую болезнь. Антибиотики не действуют на вирусы, однако было разработано несколько противовирусных препаратов.
Термин «вирус» является общим для обозначения инфекционного агента, способного паразитировать внутри клеток. Для обозначения единичной стабильной вирусной частицы, покинувшей клетку и способной инфицировать другие клетки того же типа, применяется термин «вирион»[10].
Этимология названия[править | править код]
Слово «вирус» образовано от лат. virus — «болезнетворный яд», «слизь»[11]. Для обозначения инфекционного заболевания оно впервые было применено в 1728 году, в 1790-х годах использовалось для обозначения агента, способного вызывать инфекционное заболевание[12]. Для обозначения субмикроскопического инфекционного агента латинское слово virus начало использоваться с работы голландского микробиолога Мартина Бейеринка, опубликованной в 1898 году, однако на тот момент вирус был ошибочно принят за жидкость, которая проходила через фильтр, не пропускающий бактерии[13]. Сам вирус был открыт в 1892 году Дмитрием Ивановским[4]. В подобных случаях инфекционного агента стали называть «фильтрующимся вирусом», чтобы отличать его от бактерий. Хотя таких фильтрующихся вирусов к 1920-м годам было известно уже достаточно, их природа была всё ещё непонятна, а в 1930-х годах этот термин перестали использовать в пользу более простого слова «вирус» для обозначения небактериальных агентов. В конце 1930-х годов впервые удалось визуализировать вирусы с помощью электронного микроскопа и их природа, наконец, стала понятна[14]. Первое использование производного от virus термина «вирион» для обозначения отдельных вирусных частиц датируется 1959 годом[15].
История исследований[править | править код]
По мере накопления данных об инфекционных заболеваниях различных организмов стало очевидно, что далеко не все из них вызываются патогенами, известными на тот момент — бактериями, протистами или микроскопическими грибками. В частности, Луи Пастер не смог найти агент, вызывающий бешенство, и предполагал, что этот патоген слишком мал, чтобы увидеть его в микроскоп[16]. В 1884 году французский микробиолог Шарль Шамберлан изобрёл фильтр (сейчас известный как фильтр Шамберлана или фильтр Шамберлана—Пастера), поры которого меньше бактерий. С помощью этого фильтра можно полностью удалить бактерии из раствора[17]. В 1892 году русский биолог Дмитрий Ивановский использовал его для изучения вида, сейчас известного как вирус табачной мозаики. Его эксперименты показали, что экстракт перетёртых листьев заражённых растений табака сохраняет инфекционные свойства и после фильтрации. Ивановский предположил, что инфекция может вызываться токсином, выделяемым бактериями, однако он не развил эту идею[18]. В то время считалось, что любой инфекционный агент можно выделить на фильтре и выращивать в питательной среде — таков один из постулатов микробной теории болезней[5]. Кроме того, Ивановский в оптическом микроскопе наблюдал в заражённых клетках растений кристаллоподобные тела, которые в современном понимании являлись скоплениями вирусов, впоследствии они были названы «кристаллами Ивановского»[19]. В 1898 году голландский микробиолог Мартин Бейеринк повторил эксперименты Ивановского и пришёл к выводу, что прошедший сквозь фильтр инфекционный материал есть не что иное, как новая форма инфекционных агентов[20]. Он подметил, что агент размножался только в делящихся клетках, однако его опыты не выявили того, что он представляет собой частицы. Бейеринк назвал его Contagium vivum fluidum (дословно лат. растворимый живой микроб) и вновь ввёл в употребление слово «вирус»[18]. Он утверждал, что по своей природе вирус жидкий. Эта теория впоследствии была опровергнута Уэнделлом Стэнли, доказавшим, что вирусы представляют собой частицы[18]. В том же году Фридрих Лёффлер и Пауль Фрош обнаружили первый вирус животных — возбудитель ящура (афтовирус), пропустив его через схожий фильтр[21].
В начале XX века английский бактериолог Фредерик Туорт открыл группу вирусов, инфицирующих бактерии (сейчас они известны как бактериофаги[22] или просто фаги), а франко-канадский микробиолог Феликс д’Эрелль описал вирусы, которые при добавлении к бактериям на агаре образуют вокруг себя пространство с мёртвыми бактериями. Д’Эрелль сделал точные разведения суспензии этих вирусов и установил наибольшее разведение (наименьшую концентрацию вирусов), при котором не все бактерии погибают, но тем не менее образуются отдельные области с мёртвыми клетками. Подсчитав число таких областей и учитывая коэффициент разведения, он определил число вирусных частиц в первоначальной суспензии[23]. Фаги были объявлены потенциальным средством от болезней, таких как тиф и холера, однако это было забыто в связи с открытием уникальных свойств пенициллина. Изучение фагов дало информацию о явлении «включения» и «выключения» генов, а также позволило использовать их для введения в бактериальный геном чужеродных генов.
К концу XIX века было известно, что вирусы обладают инфекционными свойствами, способны проходить через фильтры и нуждаются в живом хозяине для размножения. В то время вирусы в исследовательских целях культивировали только в растениях и животных. В 1906 году Росс Грэнвилл Гаррисон изобрёл метод выращивания тканей в лимфе, и в 1913 году Штейнард, Израэли и Ламберт использовали этот метод при выращивании вируса осповакцины на фрагментах ткани роговицы морских свинок[24]. В 1928 году Г. Б. Мэйтланд и М. К. Мэйтланд вырастили вирус осповакцины на суспензии из измельчённых куриных почек. Этот метод не применялся широко до конца 1950-х годов, когда в больших масштабах стали выращивать полиовирус для производства вакцины[25].
Другое крупное достижение принадлежит американскому патологу Эрнесту Уильяму Гудпасчеру; в 1939 г он вырастил вирус гриппа и несколько других вирусов в оплодотворённых куриных яйцах[26]. В 1949 году Джон Франклин Эндерс, Томас Уэллер и Фредерик Роббинс вырастили полиовирус на культуре клеток зародыша человека. Это был первый вирус, выращенный не на тканях животных или яйцах. Эта работа дала возможность Джонасу Солку создать эффективную полиовакцину (вакцину против полиомиелита)[27].
Первые изображения вирусов были получены после изобретения электронного микроскопа немецкими инженерами Эрнстом Руской и Максом Кноллем[28]. В 1935 году американский биохимик и вирусолог Уэнделл Мередит Стэнли тщательно изучил вирус табачной мозаики и обнаружил, что он по большей части состоит из белка[29]. Спустя короткое время этот вирус был разделён на белковую и РНК-составляющую[30]. Вирус табачной мозаики был кристаллизован первым среди вирусов, что позволило многое узнать о его структуре. Первая рентгенограмма кристаллизованного вируса была получена Берналем и Фэнкухеном в конце 1930-х годов. На основании полученных ею изображений Розалинд Франклин в 1955 году определила полную структуру вируса[31]. В том же году Хайнц Френкель-Конрат и Робли Уилльямс показали, что очищенная РНК вируса табачной мозаики и белок оболочки способны к самосборке в функциональный вирус. Это позволило им предположить, что подобный механизм лежит в основе сборки вирусов внутри клеток-хозяев[32].
Вторая половина XX века стала периодом расцвета вирусологии. В то время было открыто свыше 2000 видов вирусов животных, растений и бактерий[33]. В 1957 году были открыты лошадиный артеривирус и возбудитель вирусной диареи коров (пестивирус). В 1963 году Барух Бламберг открыл вирус гепатита B[34], а в 1965 году Хоуард Темин описал первый ретровирус. В 1970 году Темин и Дейвид Балтимор независимо друг от друга описали обратную транскриптазу, ключевой фермент, с помощью которого ретровирусы синтезируют ДНК-копии своих РНК[35]. В 1983 году группа учёных во главе с Люком Монтанье из Института Пастера во Франции впервые выделила ретровирус, известный сейчас как ВИЧ[36].
В 2002 году в Нью-Йоркском университете был создан первый синтетический вирус (вирус полиомиелита)[37].
Происхождение[править | править код]
Появление вирусов на эволюционном древе жизни неясно: некоторые из них могли образоваться из плазмид, небольших молекул ДНК, способных передаваться от одной клетки к другой, в то время как другие могли произойти от бактерий. В эволюции вирусы являются важным звеном горизонтального переноса генов, обуславливающего генетическое разнообразие[38]. Некоторые учёные считают вирусы особой формой жизни, так как они имеют генетический материал, способны создавать себе подобные вирусы, и эволюционируют путем естественного отбора. Однако у вирусов отсутствуют важные характеристики (такие как клеточное строение и собственный обмен веществ), без которых их нельзя отнести к живому. Так как они обладают некоторыми, но не всеми свойствами живого, вирусы описываются как «организмы на краю жизни».
Вирусы найдены везде, где есть жизнь, и, вероятно, вирусы существуют с момента появления первых живых клеток[39]. Происхождение вирусов неясно, поскольку они не оставляют каких бы то ни было ископаемых остатков, а их родственные связи можно изучать только методами молекулярной филогенетики[40].
Гипотезы о происхождении вирусов[править | править код]
Существует три основные гипотезы происхождения вирусов: регрессивная гипотеза, гипотеза клеточного происхождения и гипотеза коэволюции[41][42].
Регрессивная гипотеза[править | править код]
Согласно этой гипотезе, вирусы когда-то были мелкими клетками, паразитирующими в более крупных клетках. С течением времени эти клетки предположительно утратили гены, которые были «лишними» при паразитическом образе жизни. Эта гипотеза основывается на наблюдении, что некоторые бактерии, а именно риккетсии и хламидии, представляют собой клеточные организмы, которые тем не менее подобно вирусам могут размножаться только внутри другой клетки. Эту гипотезу также называют гипотезой дегенерации[43][44] или гипотезой редукции[45].
Гипотеза клеточного происхождения[править | править код]
Некоторые вирусы могли появиться из фрагментов ДНК или РНК, которые «высвободились» из генома более крупного организма. Такие фрагменты могут происходить от плазмид (молекул ДНК, способных передаваться от клетки к клетке) или от транспозонов (молекул ДНК, реплицирующихся и перемещающихся с места на место внутри генома)[46]. Транспозоны, которые раньше называли «прыгающими генами», являются примерами мобильных генетических элементов, возможно, от них могли произойти некоторые вирусы. Транспозоны были открыты Барбарой Мак-Клинток в 1950 году в кукурузе[47]. Эту гипотезу также называют гипотезой кочевания[5][48] или гипотезой побега[45].
Гипотеза коэволюции[править | править код]
Эта гипотеза предполагает, что вирусы возникли из сложных комплексов белков и нуклеиновых кислот в то же время, что и первые на Земле живые клетки, и зависят от клеточной жизни вот уже миллиарды лет. Помимо вирусов, существуют и другие неклеточные формы жизни. Например, вироиды — это молекулы РНК, которые не рассматриваются как вирусы, потому что у них нет белковой оболочки. Тем не менее, ряд характеристик сближает их с некоторыми вирусами, а потому их относят к субвирусным частицам[49]. Вироиды являются важными патогенами растений[50]. Они не кодируют собственные белки, однако взаимодействуют с клеткой-хозяином и используют её для осуществления репликации своей РНК[51]. Вирус гепатита D имеет РНК-геном, схожий с геномом вироидов, однако сам не способен синтезировать белок оболочки. Для формирования вирусных частиц он использует белок капсида вируса гепатита B и может размножаться только в клетках, заражённых этим вирусом. Таким образом, вирус гепатита D является дефектным вирусом[52]. Вирофаг Спутник схожим образом зависит от мимивируса, поражающего простейшее Acanthamoeba castellanii[53]. Эти вирусы зависят от присутствия в клетке-хозяине другого вируса и называются вирусами-сателлитами. Подобные вирусы демонстрируют, как может выглядеть промежуточное звено между вирусами и вироидами[54][55].
Каждая из этих гипотез имеет свои слабые места: регрессивная гипотеза не объясняет, почему даже мельчайшие клеточные паразиты никак не походят на вирусы. Гипотеза побега не даёт объяснения появлению капсида и других компонентов вирусной частицы. Гипотеза коэволюции противоречит определению вирусов как неклеточных частиц, зависимых от клеток-хозяев[45].
Тем не менее, в настоящее время многие специалисты признают вирусы древними организмами, появившимися, предположительно, ещё до разделения клеточной жизни на три домена[56]. Это подтверждается тем, что некоторые вирусные белки не обнаруживают гомологии с белками бактерий, архей и эукариот, что свидетельствует о сравнительно давнем обособлении этой группы. В остальном же достоверно объяснить происхождение вирусов на основании трёх закрепившихся классических гипотез не удаётся, что делает необходимыми пересмотр и доработку этих гипотез[56].
Мир РНК[править | править код]
Гипотеза мира РНК[57] и компьютерный анализ последовательностей вирусной ДНК и ДНК хозяина дают лучшее понимание эволюционных взаимоотношений между различными группами вирусов и могут помочь определить предков современных вирусов. До настоящего времени такие исследования пока не прояснили, какая из трёх основных гипотез верна[57]. Однако представляется маловероятным, чтобы все современные вирусы имели общего предка, и, возможно, в прошлом вирусы независимо возникали несколько раз по одному или нескольким механизмам, так как между различными группами вирусов имеются значительные различия в организации генетического материала[58].
Препарат головного мозга коровы, поражённой коровьим бешенством. В сером веществе образуются микроскопические полости, которые придают ткани вид губки
Прионы[править | править код]
Прионы — это инфекционные белковые молекулы, не содержащие ДНК или РНК[59]. Они вызывают такие заболевания, как почесуха овец[60], губчатая энцефалопатия крупного рогатого скота и хроническая слабость (англ. chronic wasting disease) у оленей. К прионным болезням человека относят куру, болезнь Крейтцфельдта — Якоба и синдром Герстмана — Штраусслера — Шейнкера[61]. Прионы способны стимулировать образование собственных копий. Прионный белок способен существовать в двух изоформах: нормальной (PrPC) и прионной (PrPSc). Прионная форма, взаимодействуя с нормальным белком, способствует его превращению в прионную форму. Хотя прионы фундаментально отличаются от вирусов и вироидов, их открытие даёт больше оснований поверить в то, что вирусы могли произойти от самовоспроизводящихся молекул[62].
Биология[править | править код]
Вирусы как форма жизни[править | править код]
Пока вирус находится во внеклеточной среде или в процессе заражения клетки, он существует в виде независимой частицы. Вирусные частицы (вирионы) состоят из двух или трёх компонентов: генетического материала в виде ДНК или РНК (некоторые, например мимивирусы, имеют оба типа молекул); белковой оболочки (капсида), защищающей эти молекулы, и, в некоторых случаях, — дополнительных липидных оболочек. Наличие капсида отличает вирусы от вирусоподобных инфекционных нуклеиновых кислот — вироидов. В зависимости от того, каким типом нуклеиновой кислоты представлен генетический материал, выделяют ДНК-содержащие вирусы и РНК-содержащие вирусы; на этом принципе основана классификация вирусов по Балтимору. Ранее к вирусам также ошибочно относили прионы, однако впоследствии оказалось, что эти возбудители представляют собой особые инфекционные белки и не содержат нуклеиновых кислот. Форма вирусов варьирует от простой спиральной и икосаэдрической до более сложных структур. Размеры среднего вируса составляют около одной сотой размеров средней бактерии. Большинство вирусов слишком малы, чтобы быть отчётливо различимыми под световым микроскопом.
Вирусы являются облигатными паразитами, так как не способны размножаться вне клетки. Вне клетки вирусные частицы не проявляют признаки живого и ведут себя как частицы биополимеров. От живых паразитарных организмов вирусы отличаются полным отсутствием основного и энергетического обмена и отсутствием сложнейшего элемента живых систем — аппарата трансляции (синтеза белка), степень сложности которого превышает таковую самих вирусов.
Согласно одному из определений вирусы представляют собой форму жизни, согласно другому вирусы являются комплексами органических молекул, взаимодействующими с живыми организмами. Вирусы характеризуют как «организмы на границе живого»[21]. Вирусы похожи на живые организмы в том, что они имеют свой набор генов и эволюционируют путём естественного отбора[63], а также в том, что способны размножаться, создавая собственные копии путём самосборки. Вирусы имеют генетический материал, однако лишены клеточного строения, а именно эту черту обычно рассматривают как фундаментальное свойство живой материи. У вирусов нет собственного обмена веществ, и для синтеза собственных молекул им необходима клетка-хозяин. По этой причине они не способны размножаться вне клетки[64]. В то же время такие бактерии, как риккетсии и хламидии, несмотря на то, что не могут размножаться вне клеток хозяина, считаются живыми организмами[65][66]. Общепризнанные формы жизни размножаются делением клетки, в то время как вирусные частицы самопроизвольно собираются в инфицированной клетке. От роста кристаллов размножение вирусов отличается тем, что вирусы наследуют мутации и находятся под давлением естественного отбора. Самосборка вирусных частиц в клетке даёт дополнительное подтверждение гипотезы, что жизнь могла зародиться в виде самособирающихся органических молекул[2]. Опубликованные в 2013 году данные о том, что некоторые бактериофаги обладают собственной иммунной системой, способной к адаптации[67], являются дополнительным доводом в пользу определения вируса как формы жизни.
Структура[править | править код]
Строение икосаэдрических вирионов:
А. Вирус, не имеющий липидной оболочки (например, пикорнавирус).
B. Оболочечный вирус (например, герпесвирус).
Цифрами обозначены: (1) капсид, (2) геномная нуклеиновая кислота, (3) капсомер, (4) нуклеокапсид, (5) вирион, (6) липидная оболочка, (7) мембранные белки оболочки
Палочковидный вирион вируса табачной мозаики. Цифрами обозначены: (1) РНК-геном вируса, (2) капсомер, состоящий всего из одного протомера, (3) зрелый участок капсида
Строение вириона неикосаэдрического оболочечного вируса на примере ВИЧ. Цифрами обозначены: (1) РНК-геном вируса, (2) нуклеокапсид, (3) капсид, (4) белковый матрикс, подстилающий (5) липидную мембрану, (6) gp120 — гликопротеин, с помощью которого происходит связывание вируса с клеточной мембраной, (7) gp41 — гликопротеин.
Цифрами 8—11 обозначены белки, входящие в состав вириона и необходимые вирусу на ранних стадиях инфекции: (8) — интеграза, (9) — обратная транскриптаза, (10) — Vif, Vpr, Nef и p7, (11) — протеаза
Вирусы демонстрируют огромное разнообразие форм и размеров. Как правило, вирусы значительно мельче бактерий. Большинство изученных вирусов имеют диаметр в пределах от 20 до 300 нм. Некоторые филовирусы имеют длину до 1400 нм, но их диаметр составляет лишь 80 нм[68]. В 2013 году самым крупным из известных вирусов считался Pandoravirus размерами 1 × 0,5 мкм, однако в 2014 году из многолетней мерзлоты из Сибири был описан Pithovirus, достигающий 1,5 мкм в длину и 0,5 мкм в диаметре. В настоящий момент он считается крупнейшим из известных вирусов[69]. Большинство вирионов невозможно увидеть в световой микроскоп, поэтому используют электронные — как сканирующие, так и просвечивающие[70]. Чтобы вирусы резко выделялись на окружающем фоне, применяют электронно-плотные «красители». Они представляют собой растворы солей тяжёлых металлов, таких как вольфрам, которые рассеивают электроны на покрытой ими поверхности. Однако обработка такими веществами ухудшает видимость мелких деталей. В случае негативного контрастирования «окрашивается» только фон[71].
Зрелая вирусная частица, известная как вирион, состоит из нуклеиновой кислоты, покрытой защитной белковой оболочкой — капсидом. Капсид складывается из одинаковых белковых субъединиц, называемых капсомерами[72]. Вирусы могут также иметь липидную оболочку поверх капсида (суперкапсид), образованную из мембраны клетки-хозяина. Капсид состоит из белков, кодируемых вирусным геномом, а его форма лежит в основе классификации вирусов по морфологическому признаку[73][74]. Сложноорганизованные вирусы, кроме того, кодируют специальные белки, помогающие в сборке капсида. Комплексы белков и нуклеиновых кислот известны как нуклеопротеины, а комплекс белков вирусного капсида с вирусной нуклеиновой кислотой называется нуклеокапсидом. Форму капсида и вириона в целом можно механически (физически) исследовать при помощи сканирующего атомно-силового микроскопа[75][76].
Капсид[править | править код]
Классифицируют четыре морфологических типа капсидов вирусов: спиральный, икосаэдрический, продолговатый и комплексный.
Спиральный[править | править код]
Эти капсиды состоят из одного типа капсомеров, уложенных по спирали вокруг центральной оси. В центре этой структуры может находиться центральная полость или канал. Такая организация капсомеров приводит к формированию палочковидных и нитевидных вирионов: они могут быть короткими и очень плотными или длинными и очень гибкими. Генетический материал, как правило, представлен одноцепочечной РНК (в некоторых случаях одноцепочечной ДНК) и удерживается в белковой спирали ионными взаимодействиями между отрицательными зарядами на нуклеиновых кислотах и положительными зарядами на белках. В целом, длина спирального капсида зависит от длины окружённой им нуклеиновой кислоты, а диаметр определяется размером и расположением капсомеров. Примером спирального вируса может служить вирус табачной мозаики[77].
Вирус с капсомером типа гексон. Эта иллюстрация показывает, что вирусный капсид может быть выстроен из множественных копий всего лишь двух белков
Икосаэдрический[править | править код]
Большинство вирусов животных имеют икосаэдрическую или почти шарообразную форму с икосаэдрической симметрией. Правильный икосаэдр является оптимальной формой для закрытого капсида, сложенного из одинаковых субъединиц. Минимальное необходимое число одинаковых капсомеров — 12, каждый капсомер состоит из пяти идентичных субъединиц. Многие вирусы, такие как ротавирус, имеют более двенадцати капсомеров и выглядят круглыми, но сохраняют икосаэдрическую симметрию. Капсомеры, находящиеся в вершинах, окружены пятью другими капсомерами и называются пентонами. Капсомеры треугольных граней имеют 6 соседей-капсомеров и называются гексонами[78]. Гексоны, по существу, являются плоскими, а пентоны, образующие 12 вершин, — изогнутыми. Один и тот же белок может выступать субъединицей и пентомеров, и гексамеров, или же они могут состоять из различных белков.
Продолговатый[править | править код]
Продолговатыми называют икосаэдрические капсиды, вытянутые вдоль оси симметрии пятого порядка. Такая форма характерна для головок бактериофагов[79].
Комплексный[править | править код]
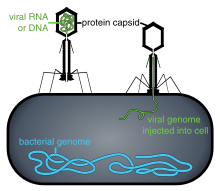
Форма этих капсидов ни чисто спиральная, ни чисто икосаэдрическая. Они могут нести дополнительные наружные структуры, такие как белковые хвосты или сложные наружные стенки. Некоторые бактериофаги, такие как фаг Т4, имеют комплексный капсид, состоящий из икосаэдрической головки, соединённой со спиральным хвостом, который может иметь шестигранное основание с отходящими от него хвостовыми белковыми нитями. Этот хвост действует наподобие молекулярного шприца, прикрепляясь к клетке-хозяину и затем впрыскивая в неё генетический материал вируса[80].
Оболочка[править | править код]
Некоторые вирусы окружают себя дополнительной оболочкой из модифицированной клеточной мембраны (плазматической или внутренней, такой как ядерная мембрана или мембрана эндоплазматического ретикулума). Этот дополнительный билипидный слой называется суперкапсидом, а шиповидные выступы на нём — пепломерами. Липидная оболочка вируса испещрена белками, кодируемыми вирусными геномом и геномом хозяина; сама же мембрана, а также любые её углеводные компоненты происходят целиком из клетки-хозяина. Таким образом формируют свою оболочку вирус гриппа и ВИЧ. Инфекционность большинства вирусов, имеющих оболочку, зависит именно от этой оболочки[81].
Поксвирусы представляют собой крупные сложноорганизованные вирусы с необычной морфологией. Генетический материал вируса связан с белками в центральной дисковидной структуре, известной как нуклеоид. Нуклеоид окружён мембраной и двумя боковыми телами неизвестной функции. Вирус имеет наружную оболочку с большим количеством белков на её поверхности. Весь вирион слегка плейоморфен (то есть способен изменять форму и размер в зависимости от условий) и может принимать форму от овальной до блоковидной[82]. Мимивирус является одним из крупнейших описанных вирусов и имеет икосаэдрический капсид диаметром 400—500 нм. Белковые филаменты, отходящие от поверхности вириона, достигают 100 нм в длину[83][84]. В 2011 году исследователи обнаружили ещё более крупный вирус на океаническом дне вблизи побережья Чили. Вирус, которому было дано временное название Megavirus chilensis, можно увидеть даже в обычный оптический микроскоп[85].
Геном[править | править код]
| Свойства | Параметры |
|---|---|
| Нуклеиновая кислота |
|
| Форма |
|
| Количество цепей |
|
| Полярность |
|
Реассортимент может стать причиной появления нового высокопатогенного штамма человеческого гриппа
Вирусы демонстрируют огромное количество вариантов организации генома; в этом смысле они более разнообразны, чем растения, животные, археи и бактерии. Существуют миллионы различных типов вирусов[7], но только примерно 5000 из них были детально описаны[6]. Генетический материал вируса может быть представлен либо ДНК, либо РНК, соответственно, вирусы подразделяют на ДНК-содержащие и РНК-содержащие. Подавляющее большинство вирусов являются РНК-содержащими. Вирусы растений чаще всего содержат одноцепочечную РНК, а бактериофаги, как правило, обладают двухцепочечными ДНК[86].
Вирусный геном может быть кольцевым, как у полиомавирусов, или линейным, как у аденовирусов. Форма генома не зависит от типа нуклеиновой кислоты. У многих РНК-содержащих вирусов и некоторых ДНК-содержащих вирусов геном часто представлен несколькими молекулами (частями), в связи с чем он называется сегментированным. У РНК-содержащих вирусов каждый сегмент часто кодирует только один белок, и обычно эти сегменты упаковываются в один капсид. Однако присутствие всех сегментов не всегда обязательно для инфекционности вируса, как это демонстрируют вирус мозаики костра[en] и некоторые другие вирусы растений[68].
Независимо от типа нуклеиновой кислоты вирусные геномы, как правило, относятся к одному из двух видов: либо одноцепочечному, либо двухцепочечному. Двухцепочечный геном включает пару комплементарных цепей нуклеиновой кислоты, а одноцепочечный — только одну цепь. И только у некоторых семейств (например, Hepadnaviridae) геном включает в себя как одноцепочечные, так и двухцепочечные участки[86].
Для большинства РНК-содержащих вирусов и некоторых вирусов с одноцепочечной ДНК определяют полярность нуклеиновой кислоты в зависимости от того, комплементарна ли она вирусной мРНК. Молекула РНК с положительной полярностью (плюс-цепь) имеет ту же последовательность нуклеотидов, что и мРНК, поэтому, по крайней мере, какая-то её часть может незамедлительно начать транслироваться клеткой-хозяином. РНК с отрицательной полярностью (минус-цепь) комплементарна мРНК, поэтому до начала трансляции на ней должна быть синтезирована положительная РНК при помощи фермента РНК-зависимой-РНК-полимеразы. Названия цепей ДНК для вирусов, содержащих одноцепочечную ДНК, сходны с таковыми для РНК: кодирующая цепь комплементарна мРНК (-), а некодирующая является её копией (+)[86]. Однако геномы нескольких типов ДНК- и РНК-содержащих вирусов представлены молекулами, имеющими различную полярность, то есть транскрипции может подвергаться любая цепь. Таковы, например, геминивирусы — вирусы растений, содержащие одноцепочечную ДНК, — и аренавирусы — вирусы животных с одноцепочечной РНК[87].
Размер генома широко варьирует у различных видов. Самым маленьким одноцепочечным ДНК-геномом обладает цирковирус из семейства Circoviridae: его геном кодирует лишь два белка и содержит всего 2000 нуклеотидов. Один из самых крупных геномов обнаружен у мимивируса: он содержит свыше 1,2 млн пар оснований и кодирует более тысячи белков[88]. Как правило, РНК-содержащие вирусы имеют меньший геном, чем ДНК-содержащие — размер их генома ограничен из-за большей вероятности ошибок во время репликации[40]. При большем размере генома ошибки, произошедшие во время его репликации, сделали бы вирус нежизнеспособным или неконкурентоспособным. Чтобы преодолеть это ограничение, РНК-вирусы часто имеют сегментированный геном — это уменьшает вероятность того, что ошибка в одном из сегментов окажется фатальной для всего генома. Напротив, ДНК-содержащие вирусы обычно имеют более крупные геномы благодаря большей точности их репликативных ферментов[89]. Однако вирусы, содержащие одноцепочечные ДНК, являются исключением из этого правила — скорость накопления мутаций в их геномах приближается к таковой для вирусов, содержащих одноцепочечные РНК[90].
Генетические изменения происходят у вирусов по разным механизмам. В их число входят случайные замены отдельных оснований в РНК или ДНК. В большинстве случаев эти точечные мутации являются «молчащими» — они не изменяют структуру белков, кодируемых мутантными генами, но иногда в результате таких изменений вирус может приобрести эволюционные преимущества, такие как устойчивость к противовирусным препаратам[91]. Дрейф антигенов имеет место тогда, когда в геноме вируса происходят масштабные изменения. Это может быть результатом рекомбинации или реассортимента. Когда это случается с вирусом гриппа, результатом может стать пандемия[92]. РНК-вирусы часто существуют как квазивиды или смесь вирусов одного вида, но с чуть различающимися нуклеотидными последовательностями генома. Такие квазивиды являются главной мишенью для естественного отбора[93].
Сегментированный геном даёт эволюционные преимущества: различные штаммы вируса с сегментированным геномом могут обмениваться генами и производить потомков с уникальными характеристиками. Это явление называется реассортиментом[94].
Генетическая рекомбинация — это процесс внесения разрыва в молекулу нуклеиновой кислоты с последующим «сшиванием» её с другими молекулами нуклеиновой кислоты. Рекомбинация может происходить между геномами двух вирусов, когда они заражают клетку одновременно. Исследования эволюции вирусов показали, что у изученных видов рекомбинация широко распространена[95]. Рекомбинация характерна как для РНК-, так и для ДНК-содержащих вирусов[96][97].
Жизненный цикл[править | править код]
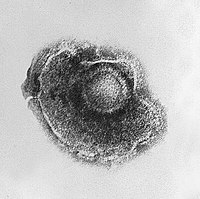
Типичный жизненный цикл вируса на примере вируса гепатита C
Вирусы не размножаются клеточным делением, поскольку не имеют клеточного строения. Вместо этого они используют ресурсы клетки-хозяина для образования множественных копий самих себя, и их сборка происходит внутри клетки.
Условно жизненный цикл вируса можно разбить на несколько взаимоперекрывающихся этапов (обычно выделяют 6 этапов[98]):
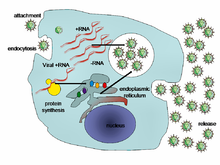
- Прикрепление представляет собой образование специфичной связи между белками вирусного капсида и рецепторами на поверхности клетки-хозяина. Это специфичное связывание определяет круг хозяев вируса. Например, ВИЧ поражает только определённый тип человеческих лейкоцитов. Это связано с тем, что оболочечный гликопротеин вируса gp120 специфично связывается с молекулой CD4 — хемокиновым рецептором, который обычно встречается на поверхности CD4+ T-лимфоцитов. Этот механизм обеспечивает инфицирование вирусом только тех клеток, которые способны осуществить его репликацию. Связывание с рецептором может вызвать конформационные изменения белка оболочки (или белка капсида в случае безоболочечного вируса), что в свою очередь служит сигналом к слиянию вирусной и клеточной мембран и проникновению вируса в клетку.
- Проникновение в клетку. На следующем этапе вирусу необходимо доставить внутрь клетки свой генетический материал. Некоторые вирусы также переносят внутрь клетки собственные белки, необходимые для её реализации (особенно это характерно для вирусов, содержащих негативные РНК). Различные вирусы для проникновения в клетку используют разные стратегии: например, пикорнавирусы впрыскивают свою РНК через плазматическую мембрану, а вирионы ортомиксовирусов захватываются клеткой в ходе эндоцитоза и попадают в кислую среду лизосом, где происходит депротеинизация вирусной частицы, после чего РНК в комплексе с вирусными белками преодолевает лизосомальную мембрану и попадает в цитоплазму. Вирусы также различают по тому, где происходит их репликация: часть вирусов (например, те же пикорнавирусы) размножается в цитоплазме клетки, а часть (например, ортомиксовирусы) в её ядре. Процесс инфицирования вирусами клеток грибов и растений отличается от инфицирования клеток животных. Растения имеют прочную клеточную стенку, состоящую из целлюлозы, а грибы — из хитина, так что большинство вирусов могут проникнуть в них только после повреждения клеточной стенки[99]. Однако почти все вирусы растений (включая вирус табачной мозаики) могут перемещаться из клетки в клетку в форме одноцепочечных нуклеопротеиновых комплексов через плазмодесмы[100]. Бактерии, как и растения, имеют крепкую клеточную стенку, которую вирусу, чтобы попасть внутрь, приходится повредить. Но в связи с тем, что клеточная стенка бактерий намного тоньше таковой у растений, некоторые вирусы выработали механизм впрыскивания генома в бактериальную клетку через толщу клеточной стенки, при котором капсид остаётся снаружи[101].
- Лишение оболочек представляет собой процесс потери капсида. Это достигается при помощи вирусных ферментов или ферментов клетки-хозяина, а может быть и результатом простой диссоциации. В конечном счёте вирусная геномная нуклеиновая кислота освобождается.
- Репликация вирусов подразумевает, прежде всего, репликацию генома. Репликация вируса включает синтез мРНК ранних генов вируса (с исключениями для вирусов, содержащих положительную РНК), синтез вирусных белков, возможно, сборку сложных белков и репликацию вирусного генома, которая запускается после активации ранних или регуляторных генов. Вслед за этим может последовать (у комплексных вирусов с крупными геномами) ещё один или несколько кругов дополнительного синтеза мРНК: «поздняя» экспрессия генов приводит к синтезу структурных или вирионных белков.
Отпочковывание вируса от клетки-хозяина
- Вслед за этим происходит сборка вирусных частиц, позже происходят некоторые модификации белков. У вирусов, таких как ВИЧ, такая модификация (иногда называемая созреванием) происходит после выхода вируса из клетки-хозяина[102].
- Выход из клетки. Вирусы могут покинуть клетку после лизиса, процесса, в ходе которого клетка погибает из-за разрыва мембраны и клеточной стенки, если такая есть. Эта особенность есть у многих бактериальных и некоторых животных вирусов. Некоторые вирусы подвергаются лизогенному циклу, где вирусный геном включается путём генетической рекомбинации в специальное место хромосомы клетки-хозяйки. Тогда вирусный геном называется провирусом, или, в случае бактериофага, профагом[103]. Когда клетка делится, вирусный геном также удваивается. В пределах клетки вирус в основном не проявляет себя; однако в некоторый момент провирус или профаг может вызвать активацию вируса, который может вызвать лизис клеток-хозяев[104].
Активно размножающийся вирус не всегда убивает клетку-хозяина. Оболочечные вирусы, в том числе ВИЧ, обычно отделяются от клетки путём отпочковывания. В ходе этого процесса вирус обзаводится своей оболочкой, которая представляет собой модифицированный фрагмент клеточной мембраны хозяина или другой внутренней мембраны[105]. Таким образом, клетка может продолжать жить и продуцировать вирус.
Особенности жизненного цикла различных групп[править | править код]
Генетический материал внутри вирусных частиц и способ его репликации значительно отличается у различных вирусов.
- ДНК-содержащие вирусы. Репликация генома у большинства ДНК-содержащих вирусов происходит в клеточном ядре. Если клетка имеет соответствующий рецептор на своей поверхности, эти вирусы проникают в клетку либо путём непосредственного слияния с клеточной мембраной (напр. герпесвирусы), либо — что бывает чаще — путём рецептор-зависимого эндоцитоза. Большинство ДНК-содержащих вирусов полностью полагаются на синтетический аппарат клетки-хозяина для производства их ДНК и РНК, а также последующего процессинга РНК. Однако вирусы с крупными геномами (например, поксвирусы) могут сами кодировать большую часть необходимых для этого белков. Геному вируса эукариот необходимо преодолеть ядерную оболочку для того, чтобы получить доступ к ферментам, синтезирующим ДНК и РНК, в случае же бактерифагов ему достаточно просто проникнуть в клетку[106][107].
- РНК-содержащие вирусы. Репликация таких вирусов обычно происходит в цитоплазме. РНК-содержащие вирусы можно подразделить на 4 группы в зависимости от способа их репликации. Механизм репликации определяется тем, является ли геном вируса одноцепочечным или двухцепочечным, вторым важным фактором в случае одноцепочечного генома является его полярность (может ли он непосредственно служить матрицей для синтеза белка рибосомами). Все РНК-вирусы используют собственную РНК-репликазу для копирования своих геномов[108].
- Вирусы, использующие обратную транскрипцию. Эти вирусы содержат одноцепочечную РНК (Retroviridae, Metaviridae, Pseudoviridae) или двухцепочечную ДНК (Caulimoviridae и Hepadnaviridae). РНК-содержащие вирусы, способные к обратной транскрипции (ретровирусы, например, ВИЧ), используют ДНК-копию генома как промежуточную молекулу при репликации, а содержащие ДНК (параретровирусы, например, вирус гепатита B) — РНК[109]. В обоих случаях используется обратная транскриптаза, или РНК-зависимая ДНК-полимераза. Ретровирусы встраивают ДНК, образующуюся в процессе обратной транскрипции, в геном хозяина, такое состояние вируса называется провирусом. Параретровирусы же этого не делают, хотя встроенные копии их генома могут давать начало инфекционным вирусам, особенно у растений[110]. Вирусы, использующие обратную транскрипцию, восприимчивы к противовирусным препаратам, ингибирующим обратную транскриптазу, в том числе к зидовудину и ламивудину.
Действие на клетки[править | править код]
Диапазон структурных и биохимических эффектов, оказываемых вирусом на инфицированную клетку, очень широк[111]. Они называются цитопатическими эффектами[112]. Большинство вирусных инфекций приводят к гибели клеток-хозяев. Причинами гибели могут быть лизис клетки, изменения клеточной мембраны и апоптоз[113]. Часто причиной гибели клетки является подавление её нормальной активности белками вируса, не все из которых входят в состав вирусной частицы[114].
Некоторые вирусы не вызывают никаких видимых изменений в поражённой клетке. Клетки, в которых вирус находится в латентном состоянии и неактивен, имеют мало признаков инфекции и нормально функционируют[115]. Это является причиной хронических инфекций, и вирус при них может никак себя не проявлять многие месяцы или годы. Так часто бывает, например, с вирусом герпеса[116][117]. Некоторые вирусы, например вирус Эпштейна — Барр, могут вызывать быстрое размножение клеток без появления злокачественности[118], в то время как другие, такие как папилломавирусы, могут вызвать рак[119].
Диапазон хозяев[править | править код]
Вирусы, несомненно, являются самыми многочисленными биологическими объектами на Земле, и по этому показателю они превосходят все организмы, вместе взятые[120]. Они поражают все формы клеточных организмов, включая животных, растения, бактерии и грибы[6]. Тем не менее различные типы вирусов могут поражать только ограниченный круг хозяев, многие вирусы видоспецифичны. Некоторые, как, например, вирус оспы, могут поражать только один вид — людей[121], в подобных случаях говорят, что вирус имеет узкий диапазон хозяев. Напротив, вирус бешенства может поражать различные виды млекопитающих, то есть он имеет широкий диапазон хозяев[122]. Вирусы растений безвредны для животных, а большинство вирусов животных безопасны для человека[123]. Диапазон хозяев некоторых бактериофагов ограничивается одним штаммом бактерий, и они могут использоваться для определения штаммов, вызывающих вспышки инфекционных заболеваний методом фагового типирования[124].
Распространение[править | править код]
Вирусы распространяются многими способами: вирусы растений часто передаются от растения к растению насекомыми, питающимися растительными соками, к примеру, тлями; вирусы животных могут распространяться кровососущими насекомыми, такие организмы известны как переносчики. Вирус гриппа распространяется воздушно-капельным путём при кашле и чихании. Норовирус и ротавирус, обычно вызывающие вирусные гастроэнтериты, передаются фекально-оральным путём при контакте с заражённой пищей или водой. ВИЧ является одним из нескольких вирусов, передающихся половым путём и при переливании заражённой крови. Каждый вирус имеет определённую специфичность к хозяевам, определяющуюся типами клеток, которые он может инфицировать. Круг хозяев может быть узок или, если вирус поражает многие виды, широк[125].
Классификация[править | править код]
В таксономии живой природы вирусы выделяют в отдельный таксон, образующий в классификации Systema Naturae 2000 вместе с доменами Bacteria, Archaea и Eukaryota корневой таксон Biota[126]. В течение XX века в систематике выдвигались предложения о создании выделенного таксона для неклеточных форм жизни (Aphanobionta Novak, 1930[127]; надцарство Acytota Jeffrey, 1971[128]; Acellularia[129]), однако такие предложения не были кодифицированы[130].
Главной задачей классификации является описание разнообразия вирусов и группировка их на основании общих свойств. В 1962 году Андре Львов, Роберт Хорн[en] и Пауль Турнье были первыми, кто разработал основные принципы классификации вирусов на основании Линнеевской иерархической системы[131]. Основными таксонами в этой системе являются отдел, класс, порядок, семейство, род и вид. Вирусы были разделены на группы по их (а не их хозяев) общим свойствам и типу нуклеиновых кислоты в геноме[132]. Классификация вирусов по Балтимору является, по существу, дополнением к более традиционной классификации[133].
Систематику и таксономию вирусов в настоящий момент кодифицирует и поддерживает Международный комитет по таксономии вирусов (International Committee on Taxonomy of Viruses, ICTV), поддерживающий также и таксономическую базу (The Universal Virus Database, ICTVdB).
Развитие вирусной метагеномики привело к идентификации множества новых РНК-вирусов, которые помогли воссоздать эволюционную историю РНК-содержащих вирусов[133]. Не существует ни одного гена, который был бы общим для всех вирусов, что однозначно свидетельствует в пользу полифилетического происхождения вирусов. Тем не менее, существует набор из приблизительно 20 ключевых генов, кодирующих белки, которые участвуют в репликации вирусов и образовании вирионов. На основании сходства по этим генам в 2018 году было предложено разделение всех вирусов на таксоны высшего ранга — реалмы[комм. 2][134]. По состоянию на 2021 год вирусы подразделяются на шесть реалмов[134].
Классификация ICTV[править | править код]
Международный комитет по таксономии вирусов разработал современную классификацию вирусов и выделил основные свойства вирусов, имеющие больший вес для классификации с сохранением единообразия семейств.
Была разработана объединённая таксономия (универсальная система для классификации вирусов). Седьмой отчёт ICTV закрепил для первых пор понятие о виде вируса как о низшем таксоне в иерархии вирусов[135][комм. 4]. Однако к настоящему моменту была изучена лишь небольшая часть от общего разнообразия вирусов, анализ образцов вирусов из человеческого организма выявил, что около 20 % последовательностей вирусных нуклеиновых кислот ещё не было рассмотрено ранее, а образцы из окружающей среды, например, морской воды и океанского дна, показали, что подавляющее большинство последовательностей являются совершенно новыми[136].
Допустимыми таксономическими единицами являются следующие ранги[137], которым соответствуют определённые суффиксы в научных названиях таксонов[138]:
- Реалм (-viria) и субреалм (-vira)
- Царство (-virae) и подцарство (-virites)
- Тип (-viricota) и подтип (-viricotina)
- Класс (-viricetes) и подкласс (-viricetidae)
- Порядок (-virales) и подпорядок (-virineae)
- Семейство (-viridae) и подсемейство (-virinae)
- Род (-virus) и подрод (-virus)
- Вид (-virus)
- Род (-virus) и подрод (-virus)
- Семейство (-viridae) и подсемейство (-virinae)
- Порядок (-virales) и подпорядок (-virineae)
- Класс (-viricetes) и подкласс (-viricetidae)
- Тип (-viricota) и подтип (-viricotina)
- Царство (-virae) и подцарство (-virites)
Классификация ICTV не регулирует подвиды, штаммы и изоляты[139].
По состоянию на апрель 2021 года выделяют 6 реалмов, 10 царств, 17 типов, 2 подтипа, 39 классов, 59 порядков, 8 подпорядков, 189 семейств, 136 подсемейств, 2224 рода, 70 подродов и 9110 видов вирусов, вироидов и сателлитов[1]. Ещё свыше 3000 вирусов не классифицировано[140].
Как отмечалось ранее, реалмы были выделены на основании сравнений последовательностей приблизительно двадцати ключевых генов, которые кодируют белки, участвующие в репликации вирусов и образовании вирионов. Реалм Riboviria включает вирусы, кодирующие РНК-зависимую РНК-полимеразу; к нему относятся вирусы III, IV, V, VI и VII групп классификации по Балтимору. К реалму Riboviria относятся практически все вирусы, имеющие РНК-геном, за исключением вируса гепатита дельта и родственных ему вирусов, которые выделяются в реалм Ribozyviria. Вирусы реалма Ribozyviria похожи на вироиды и имеют рибозим, участвующий в созревании вирусной РНК, однако, в отличие от вироидов, они кодируют нуклеокапсидный белок. Вирусы, геномы которых представлены одноцепочечной ДНК, выделяют в реалм Monodnaviria. У большинства членов этого реалма имеется общий знаковый белок — эндонуклеаза, задействованная в репликации по типу катящегося кольца. Многие представители Monodnaviria также имеют капсидный белок, содержащий укладку jelly roll. Также к реалму Monodnaviria относят два семейства вирусов с геномами в виде двуцепочечной ДНК, Polyomaviridae и Papillomaviridae, которые произошли от вирусов с геномами в виде одноцепочечной ДНК, вероятно, вирусов семейства Parvoviridae. К реалму Varidnaviria относятся все вирусы с геномами в виде двуцепочечной ДНК, которые имеют основной капсидный белок (англ. major capsid protein) с укладкой jelly roll[141]. Реалм Duplodnaviria также включает вирусы с геномами в виде двуцепочечной ДНК, однако в их основных капсидных белках присутствует укладка HK97[142]. В реалм Adnaviria выделены вирусы, у которых геном в составе вириона представлен двуцепочечной ДНК в A-форме[134].
Классификация по Балтимору[править | править код]
Группы вирусов по Балтимору. Условные обозначения: оц — одноцепочечная, дц — двуцепочечная, РТ — ретровирус или ретроидный вирус
Лауреат Нобелевской премии биолог Дейвид Балтимор разработал классификацию вирусов, названную его именем[35][143]. Классификация ICTV в настоящее время объединяется с классификацией по Балтимору, составляя современную систему классификации вирусов[144][145].
Классификация вирусов по Балтимору основывается на механизме образования мРНК. Вирусы синтезируют мРНК из собственного генома для образования белков и репликации своей нуклеиновой кислоты, однако каждое семейство вирусов имеет собственный механизм этого процесса. Вирусные геномы могут быть одноцепоченые (оц) или двухцепочечные (дц), ДНК- или РНК-содержащие, могут использовать или не использовать обратную транскриптазу. Кроме того, одноцепочечные РНК-вирусы в составе своего генома могут иметь положительную (+) или отрицательную (-) цепь РНК.
Эта система включает в себя семь основных групп[143][146]:
- (I) Вирусы, содержащие двуцепочечную ДНК и не имеющие РНК-стадии (например, герпесвирусы, поксвирусы, паповавирусы, мимивирус).
- (II) Вирусы, содержащие одноцепочечную молекулу ДНК (например, парвовирусы). В этом случае ДНК всегда положительной полярности.
- (III) Вирусы, содержащие двуцепочечную РНК (например, ротавирусы).
- (IV) Вирусы, содержащие одноцепочечную молекулу РНК положительной полярности (например, пикорнавирусы, флавивирусы).
- (V) Вирусы, содержащие одноцепочечную молекулу РНК негативной или двойной полярности (например, ортомиксовирусы, филовирусы).
- (VI) Вирусы, содержащие одноцепочечную молекулу РНК положительной полярности и имеющие в своем жизненном цикле стадию синтеза ДНК на матрице РНК, ретровирусы (например, ВИЧ).
- (VII) Вирусы, содержащие частично двуцепочечную, частично одноцепочечную ДНК[147][148] и имеющие в своём жизненном цикле стадию синтеза ДНК на матрице РНК, ретроидные вирусы (например, вирус гепатита B)[149].
Дальнейшее деление производится на основе таких признаков, как структура генома (наличие сегментов, кольцевая или линейная молекула), генетическое сходство с другими вирусами, наличие липидной оболочки, таксономическая принадлежность организма-хозяина и других.
Эволюция[править | править код]
Не существует гена, который бы присутствовал у всех вирусов, поэтому вирусы в целом являются полифилетической группой. Развитие вирусной метагеномики привело к идентификации множества новых РНК-вирусов, которые помогли воссоздать эволюционную историю вирусов[133]. Тем не менее, существует набор из приблизительно 20 ключевых генов, кодирующих белки, которые участвуют в репликации вирусов и образовании вирионов. На основании сходства по этим генам в 2018 году было предложено разделение всех вирусов на таксоны высшего ранга — реалмы. Представители реалма Riboviria, который считается монофилетическим, могли произойти от древнего простого генетического элемента, который имел РНК-зависимую РНК-полимеразу. Вирусы, геномы которых представлены двуцепочечной РНК, скорее всего, произошли как минимум дважды от разных групп вирусов с одноцепочечной РНК положительной полярности. Группы IV (геном — одноцепочечная РНК положительной полярности) и V (геном — одноцепочечная РНК отрицательной полярности) в классификации по Балтимору являются монофилетическими, а группа III, к которой относятся вирусы с геномами в виде двуцепочечной РНК, — полифилетической. Однако вирус гепатита дельта и другие члены реалма Ribozyviria формально относятся к группе V, хотя фундаментально отличаются от других РНК-содержащих вирусов наличием рибозима, участвующего в репликации вирусной РНК, и потребности в вирусе-хозяине (вирусы-сателлиты), без которого их размножение невозможно. Если включать Ribozyviria в группу V, то её также следует считать полифилетической[134].
Вирусы, геномы которых представлены одноцепочечной ДНК, образуют группу II в классификации по Балтимору и реалм Monodnaviria. Несмотря на то что все они имеют эндонуклеазу, задействованную в репликации по типу катящегося кольца, эта группа вирусов является полифилетической и возникала в ходе эволюции несколько раз как объединение в одном генетическом элементе гена указанной эндонуклеазы, заимствованной у плазмид, и гена капсидного белка у различных вирусов IV группы в ходе рекомбинации. Эволюционный путь вирусов с геномом в виде двуцепочечной ДНК также непрост. Они подразделяются на три неродственных реалма: Duplodnaviria, Varidnaviria и Adnaviria. Вирусы с геномом в виде двуцепочечной ДНК в ходе эволюции возникали по меньшей мере четыре раза от независимых предков[134].
Роль в заболеваниях человека[править | править код]
Примерами наиболее известных вирусных заболеваний человека могут служить простуда (она может иметь и бактериальную этиологию), грипп, ветряная оспа и простой герпес. Многие серьёзные болезни, например, геморрагическая лихорадка Эбола, СПИД, птичий грипп и тяжёлый острый респираторный синдром тоже вызываются вирусами. Относительная способность вируса вызывать заболевание характеризуется термином вирулентность. Некоторые заболевания исследуются на наличие вирусов в числе вызывающих агентов, например, возможна связь между человеческим герпесвирусом 6 типа и нейрологическими заболеваниями, такими как рассеянный склероз и синдром хронической усталости[150], а борнавирус, возбудитель нейрологических заболеваний у лошадей, возможно, вызывает и психические расстройства у людей[151].
Вирусы различаются механизмами воздействия на организм хозяина, которые сильно зависят от вида. На клеточном уровне этот механизм предполагает лизис клетки, то есть её смерть. У многоклеточных организмов при гибели большого числа клеток начинает страдать организм в целом. Хотя вирусы подрывают нормальный гомеостаз, приводя к заболеванию, они могут существовать внутри организма и относительно безвредно. Некоторые вирусы (например, вирус простого герпеса первого типа) могут пребывать внутри тела человека в состоянии покоя, которое называется латентностью[152]. Оно характерно для вирусов герпеса, в том числе вируса Эпштейна — Барр, вызывающего инфекционный мононуклеоз, а также вируса, вызывающего ветрянку и опоясывающий лишай. Большинство людей переболело по крайней мере одним из этих типов вируса герпеса[153]. Однако такие латентные вирусы могут и принести пользу, поскольку присутствие этих вирусов может вызвать иммунный ответ против бактериальных патогенов, например, чумной палочки (Yersinia pestis)[154].
Некоторые вирусы могут вызывать пожизненные или хронические инфекции, при которых вирус продолжает размножаться в теле хозяина, несмотря на его защитные механизмы[155]. Так происходит, например, при инфекциях, вызванных вирусами гепатита B и C. Хронически больные люди (носители), таким образом, выступают в роли резервуара инфекции[156]. Если в популяции доля вирусоносителей высока, то такое состояние характеризуется как эпидемия[157].
Эпидемиология[править | править код]
Вирусная эпидемиология является частью медицинской науки, изучающей передачу и контроль вирусных инфекций среди людей. Передача вирусов может осуществляться вертикально, то есть от матери к ребёнку, или горизонтально, то есть от человека к человеку. Примерами вертикальной передачи могут служить вирус гепатита B и ВИЧ, при которых малыш рождается уже заражённым[158]. Другим, более редким, примером служит вирус ветрянки и опоясывающего лишая, который, хотя и вызывает относительно слабые инфекции среди взрослых людей, может оказаться смертельным для эмбрионов и новорождённых малышей[159].
Горизонтальная передача является наиболее распространённым механизмом распространения вируса в популяции. Передача может осуществляться: при передаче жидкостей организма при половом акте, например, у ВИЧ; через кровь при переливании заражённой крови или пользовании грязным шприцом, например, у вируса гепатита C; передаче слюны губами, например, у вируса Эпштейна — Барр; проглатывании заражённой воды или пищи, например, у норовируса; при вдыхании воздуха, в котором находятся вирионы, например, вирус гриппа; насекомыми, например, комарами, повреждающими кожу хозяина, например, лихорадка денге. Скорость передачи вирусной инфекции зависит от нескольких факторов, к которым относят плотность популяции, количество чувствительных людей (то есть не имеющих иммунитета)[160], качество здравоохранения и погоду[161].
Эпидемиология используется, чтобы приостановить распространение инфекции в популяции во время вспышки вирусного заболевания[162]. Предпринимаются контрольные меры, основанные на знании того, как распространяется вирус. Важно найти источник (или источники) вспышки и идентифицировать вирус. Когда вирус определён, бывает возможным остановить инфекцию при помощи вакцин. Если вакцины недоступны, могут быть эффективными санация и дезинфекция. Часто заражённых людей изолируют от остального общества, то есть вирус помещается в карантин[163]. Чтобы взять под контроль вспышки ящура в Великобритании в 2001 году, были зарезаны тысячи коров[164]. У большинства инфекций человека и животных есть инкубационный период, в течение которого не проявляется никаких симптомов инфекции[165]. Инкубационный период вирусных заболеваний может длиться от нескольких дней до недель[166]. Часто перекрывающийся с ним, но в основном следующий после инкубационного периода — период передачи инфекции, когда заражённый человек или животное является заразным и может заразить других людей или животных[166]. Этот период также известен для многих инфекций, и знание длины обоих периодов является важным для контролирования вспышек[167]. Если вспышка приводит к необычно высокому числу случаев заболевания в популяции или регионе, то она называется эпидемией. Если вспышки имеют широкое распространение, то говорят о пандемии[168].
Эпидемии и пандемии[править | править код]
Численность коренного населения Америки была сильно уменьшена заразными заболеваниями, в частности, оспой, завезёнными в Америку европейскими колонизаторами. По некоторым оценкам, иноземными болезнями после прибытия Колумба в Америку, было убито около 70 % от всего коренного населения. Урон, нанесённый этими болезнями аборигенам, помог европейцам вытеснить и покорить их[169].
Пандемия — это эпидемия всемирного масштаба. Эпидемия испанского гриппа 1918 года, продолжавшаяся до 1919 года, относится к 5 категории пандемий вируса гриппа. Она была вызвана чрезвычайно агрессивным и смертоносным вирусом гриппа A. Его жертвами часто становились здоровые взрослые люди, в отличие от большинства вспышек гриппа, которые поражали в основном детей и подростков, людей старшего поколения и других ослабленных людей[170]. По старым оценкам, испанский грипп унёс 40—50 млн жизней[171], а по современным оценкам эта цифра приближается к 100 млн, то есть 5 % населения Земли в то время[172].
Большинство исследователей полагают, что ВИЧ появился в Африке южнее Сахары в течение XX столетия[173]. Сейчас эпидемия СПИД имеет масштаб пандемии. По оценкам, сейчас 38,6 миллионов людей на земле заражено ВИЧ[174]. По оценкам Объединённой программы Организации Объединённых Наций по ВИЧ/СПИД и Всемирной Организации Здравоохранения, от СПИДа (последней стадии ВИЧ-инфекции) умерло более 25 миллионов человек с момента регистрации первого случая заболевания 5 июня 1981 года, что делает его одной из наиболее разрушительных эпидемий за всю документированную историю[175]. В 2007 году было зарегистрировано 2,7 млн случаев заражения ВИЧ и 2 млн смертей от связанных с ВИЧ заболеваний[176].
Несколько высоколетальных вирусных патогенов относятся к семейству филовирусов (Filoviridae). Филовирусы представляют собой филаментовидные вирусы, которые вызывают геморрагическую лихорадку, к ним также относят возбудителя геморрагической лихорадки Эбола и марбургский вирус. Марбургский вирус привлёк широкое внимание прессы в апреле 2005 года из-за вспышки в Анголе. Продолжавшаяся с октября 2004 года и вплоть до 2005 года, эта вспышка вошла в историю как наиболее ужасная эпидемия любой геморрагической лихорадки[177].
Злокачественные опухоли[править | править код]
Вирусы могут вызывать злокачественные новообразования (в частности, гепатоцеллюлярную карциному или саркому Капоши) у человека и других видов, хотя они возникают лишь у небольшой части инфицированных. Опухолеродные вирусы относятся к различным семействам; они включают и РНК-, и ДНК-содержащие вирусы, поэтому единого типа «онковирус» не существует (устаревший термин, первоначально применявшийся для быстро трансформирующихся ретровирусов). Развитие рака определяется множеством факторов, такими как иммунитет хозяина[178] и его мутации[179]. К вирусам, способным вызывать рак у человека, относят некоторых представителей папилломавируса человека, вируса гепатита B и C, вируса Эпштейна — Барр, герпесвируса саркомы Капоши и человеческого T-лимфотропного вируса. Совсем недавно открытым вирусом рака человека является полиомавирус (полиомавирус клеток Меркеля), который в большинстве случаев вызывает редкую форму рака кожи, называемого карциномой клеток Меркеля[180]. Вирусы гепатита могут вызвать хроническую вирусную инфекцию, которая приводит к раку печени[181][182]. Заражение человеческим T-лимфотрофным вирусом может привести к тропическому спастическому параперезу и зрелой лейкемии Т-клеток[183]. Человеческие папилломавирусы могут вызывать рак шейки матки, кожи, ануса и полового члена[184]. Из герпесвирусов герпесвирус саркомы Капоши вызывает саркому Капоши и лимфому полости тела, вирус Эпштейна — Барр — лимфому Беркитта, лимфогранулематоз, нарушения B-лимфопролиферации и назофарингеальную карциному[185]. Полиомавирус клеток Меркеля близок к вирусу SV40 и полиомавирусам мышей, которые более 50 лет использовались как животные модели для изучения вирусного рака[186].
Защитная реакция хозяина[править | править код]
Первой защитной линией организма против вируса является врождённый иммунитет. Он включает клетки и другие механизмы, обеспечивающие неспецифическую защиту. Это значит, что клетки врождённого иммунитета распознают и реагируют на патогены общими способами, одинаково по отношению ко всем патогенам, но, в отличие от приобретённого иммунитета, врождённый иммунитет не даёт продолжительной и надёжной защиты хозяину[187].
Важным врождённым способом защиты организма эукариот против вирусов является РНК-интерференция[188]. Стратегия репликации многих вирусов предполагает наличие стадии двуцепочечной РНК. Для борьбы с подобными вирусами, клетка имеет систему неспецифической деградации одно- и двуцепочечных РНК. Когда подобный вирус попадает в клетку и освобождает геномную РНК в цитоплазму, белковый комплекс Dicer связывает и разрывает вирусную РНК на короткие фрагменты. Активируется биохимический путь, называемый RISC, который разрушает вирусную РНК, и препятствует размножению вируса. Ротавирусам удаётся избежать РНК-интерференции, сохраняя часть капсида даже внутри клетки и выпуская новообразованные мРНК через поры во внутреннем капсиде. Геномная двуцепочечная РНК же остаётся внутри него[189][190].
Когда система приобретённого иммунитета у позвоночных сталкивается с вирусом, она образует специфические антитела, присоединяющиеся к вирусу и часто делающие его неопасным. Это называется гуморальным иммунитетом. Наиболее важными являются два типа антител. Первый, называемый IgM, обладает высокой эффективностью в нейтрализации вирусов, но образуется клетками иммунной системы лишь в течение нескольких недель. Синтез второго — IgG — продолжается неопределённо долго. Присутствие IgM в крови хозяина говорит о наличии острой инфекции, в то время IgG свидетельствует об инфекции, перенесённой в прошлом[191]. Именно количество IgG измеряется при тестах на иммунитет[192]. Антитела могут продолжать оставаться эффективным защитным механизмом даже тогда, когда вирусу удаётся проникнуть в клетку. Клеточный белок TRIM21 может прикреплять антитела к поверхности вирусных частиц. Это вызывает последующее разрушение вирусной частицы ферментами клеточной протеосомной системы[193].
Второй защитный механизм позвоночных против вирусов называется клеточным иммунитетом и включает иммунные клетки, известные как T-лимфоциты. Клетки тела постоянно несут короткие фрагменты собственных белков на своих поверхностях, и, если Т-лимфоциты распознают здесь подозрительные вирусные фрагменты, клетка-хозяин разрушается клетками, называемыми Т-киллерами, и начинается образование специфичных к вирусу T-лимфоцитов. Такие клетки, как макрофаги, специализируются на презентации антигенов[194]. Важной защитной реакцией хозяина является продукция интерферона. Интерферон — это гормон, образуемый организмом в ответ на присутствие вируса. Его роль в иммунитете — комплексная, в конце концов он останавливает вирус, прекращая образование новых вирусов поражёнными клетками, убивая их и их близких соседей[195].
Не против всех вирусов образуется такой защитный иммунный ответ. ВИЧ удаётся избежать иммунного ответа, постоянно меняя последовательность аминокислот поверхностных белков вириона. Такие устойчивые вирусы уходят от иммунной системы, изолируясь от иммунных клеток, блокируя презентацию антигенов, благодаря устойчивости к цитокинам, уклоняясь от естественных киллеров, останавливая апоптоз клеток-хозяев, а также за счёт антигенной изменчивости[196]. Другие вирусы, называемые нейротропными вирусами, распространяются среди нервных клеток, то есть там, где иммунная система не в состоянии добраться до них из-за ГЭБ.
Профилактика и лечение[править | править код]
Так как вирусы используют для размножения естественные метаболические пути клеток-хозяев, их сложно уничтожить без применения препаратов, токсичных для самих клеток-хозяев. Наиболее эффективными медицинскими мерами против вирусных инфекций являются вакцинации, создающие иммунитет к инфекции, и противовирусные препараты, избирательно ингибирующие репликацию вирусов.
Вакцины[править | править код]
Вакцинация представляет собой дешёвый и эффективный способ предотвращения вирусных инфекций. Вакцины для предотвращения вирусных инфекций применялись ещё задолго до открытия самих вирусов. Их применение вызвано тяжёлым перенесением и смертностью от вирусных инфекций, таких как полиомиелит, корь, свинка и краснуха, поэтому лучше сделать прививку, чем переболеть[197]. С помощью вакцинации оспа была искоренена[198]. С помощью вакцин можно предотвратить более 30 вирусных инфекций у человека[199], а ещё больше вакцин используется для предотвращения вирусных заболеваний животных[200]. Вакцины могут включать ослабленные и убитые вирусы, а также вирусные белки (антигены)[201]. Живые вакцины содержат ослабленные формы вирусов, которые не вызывают болезни, но тем не менее вызывают иммунный ответ. Такие вирусы называются аттенуированными. Живые вакцины могут быть опасны для людей со слабым иммунитетом (то есть имеющих иммунодефицит), так как даже ослабленный вирус у них может вызвать исходное заболевание[202]. Для производства т. н. субъединичных вакцин используются биотехнологии и генная инженерия. В таких вакцинах используются только капсидные белки вирусов. Примером такой вакцины может служить вакцина против вируса гепатита B[203]. Субъединичные вакцины безвредны для людей с иммунодефицитом, так как они не могут вызвать заболевание[204]. Вакцина против вируса жёлтой лихорадки, содержащая ослабленный штамм 17D, пожалуй, является наиболее эффективной и безопасной из когда-либо созданных вакцин[205].
Противовирусные препараты[править | править код]
Противовирусные препараты часто представляют собой аналоги нуклеозидов. Они встраиваются в геном вируса в ходе репликации, и на этом жизненный цикл вируса останавливается, поскольку новосинтезированная ДНК неактивна. Это вызвано тем, что у аналогов отсутствуют гидроксильные группы, которые вместе с атомами фосфора соединяются и формируют жёсткий «остов» молекулы ДНК. Это называется цепной терминацией ДНК[206]. Примеры аналогов нуклеозидов — ацикловир, применяющийся против инфекций, вызванных простым вирусом герпеса, и ламивудин (против ВИЧ и вируса гепатита B). Ацикловир — один из старейших и наиболее часто назначаемых противовирусных препаратов[207]. Другие используемые противовирусные препараты имеют мишенью различные стадии жизненного цикла вирусов. Вирусу иммунодефицита человека, чтобы стать полностью заразным, необходим протеолитический фермент, известный как ВИЧ-1 протеаза. На основании этого разработан большой класс препаратов, называемых протеазными ингибиторами, инактивирующими этот фермент.
Гепатит C вызывается РНК-содержащим вирусом. У 80 % заражённых людей инфекция имеет хронический характер, и без лечения они останутся заражёнными до конца своих дней. Однако сейчас используется эффективное лекарство, состоящее из нуклеозидного аналога рибавирина, комбинированного с интерфероном[208]. Для лечения хронических носителей гепатита B было разработано схожее лечение с использованием ламивудина[209].
Вирусные заболевания у различных организмов[править | править код]
Вирусы поражают всю клеточную жизнь, но, несмотря на повсеместное распространение вирусов, каждый вид клеточных организмов имеет свой ряд поражающих вирусов, часто поражающих только этот вид[210]. Некоторые вирусы, называемые сателлитами, могут размножаться только в клетках, уже заражённых другим вирусом[53].
Вирусы животных[править | править код]
У животных вирусные инфекции вызывают иммунный ответ, который чаще всего приводит к уничтожению болезнетворного вируса. Иммунный ответ также можно вызвать вакцинами, дающими активный приобретённый иммунитет против конкретной вирусной инфекции. Однако некоторым вирусам, в том числе вирусу иммунодефицита человека и возбудителям вирусных гепатитов, удаётся ускользнуть от иммунного ответа, вызывая хроническую болезнь. Антибиотики не действуют на вирусы, однако было разработано несколько противовирусных препаратов (см. выше).
Вирусы являются важными патогенами домашнего скота. Вирусами вызываются такие заболевания, как ящур и «синий язык» (англ. bluetongue)[211]. Домашние животные, например, кошки, собаки и лошади, если их не вакцинировать, являются чувствительными к серьёзным вирусным болезням. Собачий парвовирус — это маленький ДНК-содержащий вирус, часто оказывается смертельным для щенков[212]. Однако большинство вирусов безвредно сосуществуют со своими хозяевами, не подавая никаких признаков или симптомов болезни[5].
Вирусы беспозвоночных[править | править код]
На долю беспозвоночных приходится около 80 % всех известных видов животных, поэтому нет ничего удивительного в том, что они скрывают в себе огромное множество вирусов различных типов. Наиболее изучены вирусы, поражающие насекомых, но даже здесь доступная по ним информация носит фрагментарный характер. Впрочем, в последнее время были описаны вирусные заболевания и у других беспозвоночных. Эти вирусы остаются малоизученными, и некоторые сообщения об открытии следует принимать с осторожностью, пока вирусная природа этих болезней не будет окончательно доказана. Кроме того, необходимо также проверить инфективность изолированных вирусов по отношению к неинфицированным хозяевам того же вида, у кого эти вирусы были обнаружены[213].
В настоящее время выделено отдельное семейство вирусов, поражающих главным образом членистоногих, в особенности — насекомых, обитающих в водных и влажных средах: иридовирусы (Iridoviridae, от англ. Invertebrate iridescent viruses — «радужные вирусы беспозвоночных»; такой цвет наблюдается у образцов поражённых насекомых). Они представляют собой икосаэдрические частицы 120—180 нм в диаметре, содержащие внутреннюю липидную мембрану и геном в виде двуцепочечной ДНК, содержащей 130—210 тыс. пар нуклеотидов[214].
Другие вирусы, поражающие насекомых: семейство Baculoviridae, подсемейство Entomopoxvirinae семейства Poxviridae, род Densovirus семейства Parvoviridae, некоторые вирусы семейств Rhabdoviridae, Reoviridae, Picornaviridae[215].
Как и все беспозвоночные, медоносная пчела чувствительна ко многим вирусным инфекциям[216].
Вирусы растений[править | править код]
Перцы, поражённые вирусом пятнистости
Существует много типов вирусов растений. Часто они вызывают снижение урожайности, принося большие убытки сельскому хозяйству, поэтому контроль таких вирусов очень важен с экономической точки зрения[217]. Вирусы растений часто распространяются от растения к растению организмами, известными как переносчики. Обычно ими выступают насекомые, но ими могут быть также грибы, черви-нематоды и одноклеточные организмы. Если контроль вируса растений признаётся экономически выгодным, например, в случае многолетних фруктовых деревьев, усилия направляются на устранение переносчиков или альтернативных хозяев, к примеру, сорняков[218]. Вирусы растений не могут поражать человека и других животных, так как они могут размножаться лишь в живых растительных клетках[219].
Растения имеют сложные и эффективные механизмы защиты от вирусов. Наиболее эффективным механизмом является наличие так называемого гена устойчивости (R от англ. resistance — «устойчивость»). Каждый R-ген отвечает за устойчивость к отдельному вирусу и вызывает гибель клеток, соседних с поражённой, что невооружённым глазом видно как большое пятно. Это останавливает развитие болезни вследствие остановки распространения вируса[220]. Другим эффективным методом является РНК-интерференция[221]. Будучи поражёнными вирусом, растения часто начинают вырабатывать природные противовирусные вещества, такие как салициловая кислота, оксид азота NO и активные формы кислорода[222].
Вирусы растений и созданные на их основе вирусоподобные частицы (VLPs) нашли применение в биотехнологиях и нанотехнологиях. Капсиды большинства вирусов растений имеют простую и устойчивую структуру, и вирусные частицы могут производиться в огромных количествах как поражённым растением, так и различными гетерологичными системами. Вирусы растений могут изменяться химически и генетически, заключая в оболочку инородные частицы, а также способны встраиваться в надмолекулярные структуры, что делает возможным их применение в биотехнологиях[223].
Для повышения достоверности результатов диагностики вирусологического статуса растений необходимо использовать как минимум два метода, причем, желательно, высокочувствительные — ИФА и ПЦР. Выявляемость вирусов повышается за счет использования гидроксипроизводного бензойной кислоты (ГПБК) в качестве эффективного антиоксиданта, учёта биологических особенностей культур и условий окружающей среды[224].
Вирусы грибов[править | править код]
Вирусы грибов называются миковирусами. В настоящий момент вирусы выделены у 73 видов из 57 родов, относящихся к 5 классам[225], но, предположительно, в безвредном состоянии вирусы существуют у большинства грибов. В общем эти вирусы представляют собой круглые частицы 30—45 нм диаметром, состоящие из множества субъединиц единственного белка, сложенных вокруг генома, представленного двуцепочечной РНК. Как правило, вирусы грибов относительно безвредны. Некоторые грибные штаммы могут поражаться многими вирусами, но большинство миковирусов тесно связаны со своим единственным хозяином, от которого передаются его потомкам. Классификацией вирусов грибов сейчас занимается специально созданный комитет в составе ICTV[225]. В данный момент он признаёт 3 семейства вирусов грибов, а наиболее изученные миковирусы относятся к семейству Totiviridae[226]
Установлено, что антивирусная активность пенициллиновых грибов вызвана индукцией интерферона двухцепочечной РНК от вирусов, поражающих грибы[225].
Если же вирус, попадая в гриб, проявляет свою вирулентность, то реакция гриба на это может быть различной: снижение или повышение вирулентности у патогенных видов, дегенерация мицелия и плодовых тел, изменение окраски, подавление спороношения. Некапсидированные вирусные РНК передаются через анастомозы независимо от митохондрий.
Вирусные заболевания могут наносить ущерб грибоводческим предприятиям, например, вызывать побурение плодовых тел шампиньона, изменение окраски у зимнего опёнка, что снижает их коммерческую ценность. Вирусы, вызывающие гиповирулентность грибов-патогенов, могут использоваться для борьбы с заболеваниями растений[227][228].
Вирусы протистов[править | править код]
К вирусам протистов относят вирусы, поражающие одноклеточных эукариот, не включённых в царство животные, растения или грибы. Некоторые из известных на данный момент вирусов протистов[229]:
| Название вируса (род) | Систематическое положение (семейство) |
Поражаемый протист |
|---|---|---|
| Dinornavirus | Alvernaviridae | Heterocapsa circularisquama |
| Endornavirus | Endornaviridae | Phytophthora |
| Labyrnavirus | Labyrnaviridae | Aurantiochytrium |
| Marnavirus | Marnaviridae | Heterosigma akashiwo |
| Marseillevirus | Marseilleviridae | Amoeba |
| Mimivirus | Mimiviridae | Acanthamoeba polyphaga |
| Chlorovirus | Phycodnaviridae | Paramecium bursaria |
| Coccolithovirus | Phycodnaviridae | Emiliania huxleyi |
| Prasinovirus | Phycodnaviridae | Micromonas pusilla |
| Prymnesiovirus | Phycodnaviridae | Chrysochromulina brevifilum |
| Raphidovirus | Phycodnaviridae | Heterosigma akashiwo |
| Cryspovirus | Partitiviridae | Cryptosporidium parvum |
| Hemivirus | Pseudoviridae | Volvox carteri |
| Pseudovirus | Pseudoviridae | Physarum polycephalum |
| Mimoreovirus | Reoviridae | Micromonas pusilla |
| Giardiavirus | Totiviridae | Giardia lamblia |
| Leishmaniavirus | Totiviridae | Leishmania |
| Trichomonasvirus | Totiviridae | Trichomonas vaginalis |
| Bacilladnavirus | Не определено | Chaetoceros salsugineum
Rhizosolenia setigera |
| Dinodnavirus | Не определено | Heterocapsa circularisquama |
| Rhizidiovirus | Не определено | Rhizidiomyces |
Многие вирусы простейших имеют необычно большие размеры. Например, геном Marseillevirus, впервые выделенный из амёбы, имеет геном размером 368 КБ, а Mamavirus, поражающий протиста Acanthamoeba, по размеру превосходит даже мимивирус (а его капсид достигает около 500 нм в диаметре) и некоторые бактерии. Также в число гигантских вирусов входит вирус, поражающий широко распространённого морского протиста Cafeteria roenbergensis (англ. Cafeteria roenbergensis virus, CroV)[230].
Вирусы бактерий[править | править код]
Бактериофаги представляют собой широко распространённую и разнообразную группу вирусов, достигающую большей численности в водных средах обитания — в океанах этих вирусов более чем в 10 раз больше, чем бактерий[231], достигая численности в 250 млн. вирусов на миллилитр морской воды[232]. Эти вирусы поражают специфичные для каждой группы бактерии, связываясь с клеточными рецепторами на поверхности клетки и затем проникая внутрь неё. В течение короткого промежутка времени (иногда считанных минут) бактериальная полимераза начинает транслировать вирусную мРНК в белки. Эти белки или входят в состав вирионов, собираемых внутри клетки, или являются вспомогательными белками, помогающими сборке новых вирионов, или вызывают лизис клетки. Вирусные ферменты вызывают разрушение клеточной мембраны, и, в случае фага Т4, всего лишь через 20 минут после проникновения в клетку на свет появляются свыше трёх сотен бактериофагов[233].
Главным механизмом защиты бактериальных клеток от бактериофагов является образование ферментов, разрушающих чужеродную ДНК. Эти ферменты, называемые эндонуклеазами рестрикции, «разрезают» вирусную ДНК, впрыснутую внутрь клетки[234]. Бактерии также используют систему, называемую CRISPR, которая хранит информацию о геномах вирусов, с которыми бактерия сталкивалась ранее, и это позволяет клетке блокировать репликацию вируса с помощью интерференции РНК[235][236]. Эта система обеспечивает приобретённый иммунитет бактериальной клетки.
Бактериофаги могут выполнять и полезную для бактерий функцию, так, именно бактериофаг, заражающий дифтерийные палочки, кодирует ген их токсина, нужного этим бактериям и столь опасного для человека[237]:45.
Вирусы архей[править | править код]
Некоторые вирусы размножаются внутри архей: это двуцепочечные ДНК-содержащие вирусы с необычной, подчас уникальной формой[8][243]. Наиболее детально они изучены у термофильных архей, в частности, порядков Sulfolobales и Thermoproteales[244]. Мерами защиты против этих вирусов могут быть РНК-интерференция от повторяющихся последовательностей ДНК в геномах архей, родственных генам вирусов[245][246].
Вирусы вирусов[править | править код]
При изучении вирусных фабрик мимивируса было обнаружено, что на них собираются небольшие вирионы и другого вируса, который был назван Спутником[247]. Спутник, по всей видимости, сам не способен заражать клетки амёб (которые служат хозяевами мимивируса) и размножаться в них, но может делать это совместно с мама- или мимивирусом, что классифицирует его как вирус-сателлит. Спутник стал первым известным вирусом-сателлитом, содержащим двухцепочечную ДНК и размножающимся в эукариотических клетках. Однако авторы работы предлагают рассматривать его не просто как сателлит, а как вирофаг (вирус вируса) по аналогии с бактериофагами (вирусами бактерий)[248][249][250]. Репликация как вирусов-сателлитов, так и вирофагов зависит от другого вируса и клетки-хозяина. Однако для репликативного цикла вирофагов характерны три уникальные особенности. 1) Отсутствует ядерная фаза репликации. 2) Репликация вирофагов происходит в вирусных фабриках гигантских ДНК-содержащих вирусов-хозяев. 3) Вирофаги зависят от ферментов, синтезируемых вирусами-хозяевами, но не клетками-хозяевами. Таким образом, вирофаги считаются паразитами гигантских ДНК-содержащих вирусов, например, мимивирусов и фикоднавирусов[251][252]. При этом синтез капсидных белков вирофагов (как и синтез белков всех известных вирусов) полностью зависит от трансляционного аппарата клетки-хозяина[253]. Хотя строгого доказательства ещё нет, некоторые факты говорят в пользу того, что Спутник действительно является вирофагом. Например, в его геноме присутствуют регуляторные элементы, характерные для мимивируса и узнаваемые его транскрипционным аппаратом (последовательности, близкие к позднему промотору мимивируса, сигналы полиаденилирования). Кроме того, присутствие Спутника снижает продуктивность размножения мимивируса: лизис клетки-хозяина происходит с задержкой, и образуются дефектные вирионы мимивируса[247].
По данным на 2016 год, из культивируемых клеток было изолировано пять вирофагов. Ещё 18 вирофагов описано на основе данных метагеномного анализа (геномы двух из них почти полностью секвенированы)[254][255].
Роль вирусов в биосфере[править | править код]
Вирусы являются самой распространённой формой существования органической материи на планете по численности. Они играют важную роль в регуляции численности популяций некоторых видов живых организмов (например, вирус дикования с периодом в несколько лет сокращает численность песцов в несколько раз).
Иногда вирусы образуют с животными симбиоз[256][257]. Так, например, яд некоторых паразитических ос содержит структуры, называемые поли-ДНК-вирусами (Polydnavirus, PDV), имеющие вирусное происхождение.
Однако основная роль вирусов в биосфере связана с их деятельностью в водах океанов и морей.
Роль в водных экосистемах[править | править код]
Вирусы — это самая распространенная форма жизни в океане, их концентрация достигает 10 млн вирусов на 1 миллилитр поверхности моря[258]. Чайная ложка морской воды содержит около миллиона вирусов[259]. Они необходимы для регуляции пресноводных и морских экосистем[260]. Большая часть этих вирусов является бактериофагами, безвредными для растений и животных. Они поражают и разрушают бактерии в водном микробном сообществе, таким образом, участвуя в важном процессе круговорота углерода в морской среде. Органические молекулы, освободившиеся из бактериальных клеток благодаря вирусам, стимулируют рост новых бактерий и водорослей[261].
Микроорганизмы составляют более 90 % биомассы в море. По оценкам, каждый день вирусы убивают около 20 % этой биомассы, а количество вирусов в океанах в 15 раз превышает число бактерий и архей. Вирусы являются главными агентами, вызывающими быстрое прекращение цветения воды[262], убивающего другую жизнь в море[263], за счёт гибели вызывающих его водорослей. Численность вирусов убывает с удалением от берега и с увеличением глубины, поскольку там меньше организмов-хозяев[264].
Значение морских вирусов очень велико. Регулируя процесс фотосинтеза, они играют второстепенную роль в сокращении количества углекислого газа в атмосфере приблизительно на 3 гигатонны углерода в год[264].
Как и другие организмы, морские млекопитающие восприимчивы к вирусным инфекциям. В 1988 и 2002 годах тысячи обыкновенных тюленей были убиты парамиксовирусом Phocine distemper virus[265]. В популяциях морских млекопитающих циркулирует множество других вирусов, в том числе калицивирусы, герпесвирусы, аденовирусы и парвовирусы[264].
Роль в эволюции[править | править код]
Вирусы являются важным естественным средством переноса генов между различными видами, что вызывает генетическое разнообразие и направляет эволюцию[38]. Считается, что вирусы сыграли центральную роль в ранней эволюции, ещё до расхождения бактерий, архей и эукариот, во времена последнего универсального общего предка жизни на Земле[266]. Вирусы и по сей день остаются одним из крупнейших живых хранилищ неисследованного генетического разнообразия на Земле[264].
Вирусы имеют генетические связи с представителями флоры и фауны Земли. Согласно последним исследованиям[источник не указан 2591 день], геном человека более чем на 32 % состоит из вирусоподобных элементов, транспозонов и их остатков. С помощью вирусов может происходить так называемый горизонтальный перенос генов (ксенология), то есть передача генетической информации не от непосредственных родителей к своему потомству, а между двумя неродственными (или даже относящимися к разным видам) особями. Так, в геноме высших приматов существует ген, кодирующий белок синцитин, который, как считается, был привнесён ретровирусом.
Применение[править | править код]
В науках о жизни и медицине[править | править код]
Вирусы имеют важное значение для исследований в молекулярной и клеточной биологии, так как они представляют собой простые системы, которые можно использовать для управления и изучения функционирования клеток[267]. Изучение и использование вирусов дало ценную информацию о различных аспектах клеточной биологии[268]. К примеру, вирусы применялись в генетических исследованиях, и они помогли нам прийти к пониманию ключевых механизмов молекулярной генетики, таких как репликация ДНК, транскрипция, процессинг РНК, трансляция, транспорт белков.
Генетики часто используют вирусы как векторы для ввода генов в изучаемые клетки. Это позволяет заставить клетку производить чуждые вещества, а также изучить эффект от ввода нового гена в геном. Аналогично в виротерапии вирусы используют как векторы для лечения различных болезней, так как они избирательно действуют на клетки и ДНК. Это даёт надежды, что вирусы смогут помочь в борьбе с раком и найдут своё применение в генотерапии. Некоторое время восточноевропейские учёные прибегали к фаговой терапии как к альтернативе антибиотикам, и интерес к таким методам возрастает, поскольку в настоящее время у некоторых патогенных бактерий обнаружена высокая устойчивость к антибиотикам[269].
Биосинтез заражёнными клетками чужеродных белков лежит в основе некоторых современных промышленных способов получения белков, например, антигенов. Недавно промышленным способом были получены некоторые вирусные векторы и лекарственные белки, в настоящее время они проходят клинические и доклинические испытания[270].
В материаловедении и нанотехнологиях[править | править код]
Современные направления в нанотехнологиях обещают принести значительно более разностороннее применение вирусам. С точки зрения материаловедов, вирусы можно рассматривать как органические наночастицы. Их поверхность несёт специальные приспособления для преодоления биологических барьеров клетки-хозяина. Точно определены форма и размер вирусов, а также количество и природа функциональных групп на их поверхности. По существу, вирусы часто используют в материаловедении как «подмости» для ковалентно связанных поверхностных модификаций. Одно из примечательных качеств вирусов — то, что они специально «подогнаны» направленной эволюцией под клетки, выступающие хозяевами. Мощные методы, разработанные биологами, легли в основу инженерных приёмов в наноматериалах, открыв тем самым широкую сферу применения вирусов, выходящую далеко за пределы биологии и медицины[271].
Из-за своих размеров, формы и хорошо изученной химической структуры вирусы использовали как шаблоны для организации материалов на наноуровне. Примером такой недавней работы могут служить исследования, проведённые Исследовательской лабораторией Наваля в Вашингтоне (округ Колумбия) с использованием вируса мозаики коровьего гороха (англ. Cowpea Mosaic Virus (CPMV)) для усиления сигналов в сенсорах с ДНК-микрочипами. В данном случае вирусные частицы разделяли частицы флуоресцентных красителей, которые использовались для передачи сигнала, предотвращая, таким образом, скопление нефлуоресцентных димеров, выступающих как гасители сигнала[272]. Другим примером использования CPMV является применение его как наноразмерного образца для молекулярной электроники[273].
Искусственные вирусы[править | править код]
Многие вирусы могут быть получены de novo, то есть с нуля, а первый искусственный вирус был получен в 2002 году[37]. Несмотря на некоторые неправильные трактовки, при этом синтезируется не сам вирус как таковой, а его геномная ДНК (в случае ДНК-вирусов) или комплементарная копия ДНК его генома (в случае РНК-вирусов). У вирусов многих семейств искусственная ДНК или РНК (последняя получается путём обратной транскрипции синтетической комплементарной ДНК), будучи введённой в клетку, проявляет инфекционные свойства. Иными словами, они содержат всю необходимую информацию для образования новых вирусов. Эту технологию в настоящее время используют для разработки вакцин нового типа[274]. Возможность создавать искусственные вирусы имеет далеко идущие последствия, поскольку вирус не может вымереть, пока известна его геномная последовательность и имеются чувствительные к нему клетки. В наши дни полные геномные последовательности 2408 различных вирусов (в том числе оспы) находятся в публичном доступе в онлайн-базе данных, поддерживаемой Национальными институтами здравоохранения США[275].
Вирусы как оружие[править | править код]
Способность вирусов вызывать опустошительные эпидемии среди людей порождает беспокойство, что вирусы могут использоваться как биологическое оружие. Дополнительные опасения вызвало успешное воссоздание вредоносного вируса испанского гриппа в лаборатории[276]. Другим примером может служить вирус оспы. Он на всём протяжении истории опустошал множество стран вплоть до его окончательного искоренения. Официально образцы вируса оспы хранятся лишь в двух местах в мире — в двух лабораториях в России и США[277]. Опасения, что он может быть использован как оружие, не совсем беспочвенны[277]; вакцина против оспы иногда имеет тяжёлые побочные эффекты — в последние годы до официально объявленного искоренения вируса больше людей серьёзно заболели из-за вакцины, чем от вируса[278], поэтому вакцинация против оспы больше не практикуется повсеместно[279]. По этой причине большая часть современного населения Земли практически не имеет устойчивости к оспе[277].
В массовой культуре[править | править код]
В фильмах и других произведениях мир инфекционных заболеваний, в том числе вирусных, редко представляется достоверно. Исключая фильмы-биографии учёных и фильмы, повествующие о великих эпидемиях прошлого, в большинстве из них центральным событием является вспышка неизвестного болезнетворного агента, появление которого стало результатом акта биотерроризма, инцидента в лаборатории, или же он попал из космоса[280].
В литературе[править | править код]
Вирусная инфекция заложена в основу следующих произведений (список неполный):
- Кодзи Судзуки. «Звонок».
- Кир Булычёв. «Лиловый шар».
- Стивен Кинг. «Противостояние».
- Майкл Крайтон. «Штамм „Андромеда“»[280].
- Джек Лондон. «Алая чума».
- Дэн Браун. «Инферно».
В кинематографе[править | править код]
Вспышка необычной вирусной инфекции лежит в основе сюжета следующих художественных фильмов и сериалов[280]:
- «В город пришла беда» (1966)
- «Часы Пандоры»[en] (1996)
- «28 дней спустя» (англ. 28 Days Later) (2003)
- «28 недель спустя» (англ. 28 Weeks Later) (2007)
- Штамм «Андромеда». Этот фильм, снятый по одноимённой повести Майкла Крайтона, можно назвать наиболее точным в научном плане[280].
- «12 обезьян» (1995)
- «Обитель зла» (2002) и его продолжения.
- «Эпидемия» (1995)
- «Лиловый шар» (1987)
- «Носители» (2009)
- «Я — Легенда» (2007)
- «Заражение» (2011)
- «Карантин» (2008)
- «Карантин 2: Терминал» (2011)
- «Регенезис» (сериал, 2004—2008)
- «Выжить после» (сериал 2013)
- «Спираль» (сериал, 2014—2015)
- «Штамм» (сериал, 2014—2015)
- «Последний корабль» (сериал, 2014—2015)
- «Закрытая школа» (сериал, 2011—2012)
- «Война миров Z» (2013)
- «Инферно» (2016)
- «Эпидемия» (2019)
В мультипликации[править | править код]
В последние годы вирусы нередко становятся «героями» мультфильмов и мультсериалов, среди которых следует назвать, например, «Осмосис Джонс» (США, 2001), «Оззи и Дрикс» (США, 2002—2004 гг.) и «Вирус атакует» (Италия, 2011).
См. также[править | править код]
- Компьютерный вирус
- Медиавирус
- Вироид
- Вирусоподобные частицы
Примечания[править | править код]
- Комментарии
- ↑ На английском языке. В латинском языке вопрос о множественном числе данного слова является спорным. Слово лат. virus принадлежит редкой разновидности II склонения, словам среднего рода на -us: Nom.Acc.Voc. virus, Gen. viri, Dat.Abl. viro. Так же склоняются лат. vulgus и pelagus; в классической латыни множественное число зафиксировано только у последнего: pelage, форма древнегреческого происхождения, где η<εα.
- ↑ 1 2 На данный момент устоявшегося русскоязычного термина, соответствующего англ. realm в таксономии, нет.
- ↑ В различных источниках такое свойство вирусов, как инфекционность, имеет различное значение. Некоторые, например, Большая Советская энциклопедия, определяют вирусы как неклеточные организмы, обладающие свойством вызывать инфекционные болезни у клеточных организмов. Другие, например, Большой Энциклопедический словарь и Биологический энциклопедический словарь, не относят инфекционность к определяющим свойствам вирусов.
- ↑ Как отмечается там, «вид вирусов представляет собой политетический класс вирусов, которые вместе образуют единую линию поколений и занимают особенную экологическую нишу». «Политетический» класс — это таксономическая группа, члены которой имеют несколько общих свойств, хотя и не обязательно имеют все одинаковые свойства. Этим вид вирусов отличается от вышестоящих вирусных таксонов, которые являются «универсальными» классами и имеют набор свойств, обязательных для каждого их члена.
- Использованная литература и источники
- ↑ 1 2 Таксономия вирусов (англ.) на сайте Международного комитета по таксономии вирусов (ICTV). (Дата обращения: 14 мая 2021).
- ↑ 1 2 Koonin E. V., Senkevich T. G., Dolja V. V. The ancient Virus World and evolution of cells // Biol. Direct. — 2006. — Т. 1. — С. 29. — doi:10.1186/1745-6150-1-29. — PMID 16984643. — PMC 1594570.
- ↑ Дмитрий Гапон. «Фильтрующиеся вирусы» открытие в гранях времени // Наука и жизнь. — 2015. — № 6. — С. 38—50. Архивировано 29 июля 2017 года.
- ↑ 1 2 Дмитрий Гапон. «Фильтрующиеся вирусы» открытие в гранях времени // Наука и жизнь. — 2015. — № 7. — С. 31—41. Архивировано 30 июля 2017 года.
- ↑ 1 2 3 4 Dimmock, 2007, p. 4.
- ↑ 1 2 3 Dimmock, 2007, p. 49.
- ↑ 1 2 How many viruses on Earth? Дата обращения: 14 марта 2017. Архивировано 21 марта 2020 года.
- ↑ 1 2 Lawrence C. M., Menon S., Eilers B. J., et al. Structural and functional studies of archaeal viruses (англ.) // J. Biol. Chem. : journal. — 2009. — Vol. 284, no. 19. — P. 12599—12603. — doi:10.1074/jbc.R800078200. — PMID 19158076. — PMC 2675988. Архивировано 24 июля 2018 года.
- ↑ Edwards R. A., Rohwer F. Viral metagenomics // Nat. Rev. Microbiol. — 2005. — Т. 3, № 6. — С. 504—510. — doi:10.1038/nrmicro1163. — PMID 15886693.
- ↑ Casjens S. Desk Encyclopedia of General Virology / Mahy B. W. J. and Van Regenmortel M. H. V.. — Boston: Academic Press, 2010. — С. 167. — ISBN 0-12-375146-2.
- ↑ Русский перевод слова — согласно следующему изданию:
И. Х. Дворецкий. Латинско-русский словарь. Ок. 50000 слов. — Издание второе, переработанное и дополненное. — М.: Русский язык, 1976. — С. [1084] (стб. 1). — 1096 с. — 65 000 экз. Архивная копия от 19 января 2015 на Wayback Machine
virus, i n 1) слизь (cochlearum PM); слизистый сок (pastinanceae PM); семя животных V, PM; 2) ядовитое выделение, яд (serpentus V): ferro v. inest O стрела отравлена; 3) ядовитость, язвительность, жёлочность, едкость (acerbitatis C; linguae, mentis Sil); 4) отвратительный запах, зловоние (paludis Col; animae ursi pestilens v. PM); 5) острый вкус, острота (vini PM); едкость, горечь (sc. maris Lcr; ponti Man).
- ↑ Harper D. virus. The Online Etymology Dictionary (2011). Дата обращения: 23 декабря 2011. Архивировано 19 января 2013 года.
- ↑ Virus // Merriam-Webster.com Dictionary : [англ.] : [арх. 2 ноября 2021]. — Merriam-Webster.
- ↑ William C. Summers. Inventing Viruses (англ.) // Annual Review of Virology. — 2014. — 3 November (vol. 1, iss. 1). — P. 25–35. — ISSN 2327-056X. — doi:10.1146/annurev-virology-031413-085432. Архивировано 7 апреля 2020 года.
- ↑ Harper D. virion. The Online Etymology Dictionary (2011). Дата обращения: 24 декабря 2011. Архивировано 19 января 2013 года.
- ↑ Bordenave G. Louis Pasteur (1822—1895) // Microbes and Infection / Institut Pasteur. — 2003. — Т. 5, № 6. — С. 553—560. — doi:10.1016/S1286-4579(03)00075-3. — PMID 12758285.
- ↑ Shors, 2008, pp. 76—77.
- ↑ 1 2 3 Collier, 1998, p. 3.
- ↑ Гапон Д. «Фильтрующиеся вирусы». Открытие в гранях времени // Наука и жизнь. — 2015. — № 6. — С. 38—50.
- ↑ Dimmock, 2007, p. 4—5.
- ↑ 1 2 Fenner F. Desk Encyclopedia of General Virology / Mahy B. W. J. and Van Regenmortal M. H. V.. — 1. — Oxford, UK: Academic Press, 2009. — С. 15. — ISBN 0-12-375146-2.
- ↑ Shors, 2008, p. 589.
- ↑ D’Herelle F. On an invisible microbe antagonistic toward dysenteric bacilli: brief note by Mr. F. D’Herelle, presented by Mr. Roux (англ.) // Research in Microbiology : journal. — 2007. — Vol. 158, no. 7. — P. 553—554. — doi:10.1016/j.resmic.2007.07.005. — PMID 17855060.
- ↑ Steinhardt E., Israeli C., Lambert R. A. Studies on the cultivation of the virus of vaccinia (англ.) // J. Inf Dis. : journal. — 1913. — Vol. 13, no. 2. — P. 294—300. — doi:10.1093/infdis/13.2.294.
- ↑ Collier, 1998, p. 4.
- ↑ Goodpasture E. W., Woodruff A. M., Buddingh G. J. The cultivation of vaccine and other viruses in the chorioallantoic membrane of chick embryos (англ.) // Science : journal. — 1931. — Vol. 74, no. 1919. — P. 371—372. — doi:10.1126/science.74.1919.371. — PMID 17810781.
- ↑ Rosen, F. S. Isolation of poliovirus—John Enders and the Nobel Prize (англ.) // New England Journal of Medicine : journal. — 2004. — Vol. 351, no. 15. — P. 1481—1483. — doi:10.1056/NEJMp048202. — PMID 15470207.
- ↑ From Nobel Lectures, Physics 1981—1990, (1993) Editor-in-Charge Tore Frängsmyr, Editor Gösta Ekspång, World Scientific Publishing Co., Singapore.
- В 1887 году Бьюст увидел один из крупнейших вирусов, вирус коровьей оспы, в оптический микроскоп, предварительно окрасив его. В то время не было известно, что это вирус. (Buist J. B. Vaccinia and Variola: a study of their life history Churchill, London)
- ↑ Stanley W. M., Loring H. S. The isolation of crystalline tobacco mosaic virus protein from diseased tomato plants (англ.) // Science : journal. — 1936. — Vol. 83, no. 2143. — P. 85. — doi:10.1126/science.83.2143.85. — PMID 17756690.
- ↑ Stanley W. M., Lauffer M. A. Disintegration of tobacco mosaic virus in urea solutions (англ.) // Science : journal. — 1939. — Vol. 89, no. 2311. — P. 345—347. — doi:10.1126/science.89.2311.345. — PMID 17788438.
- ↑ Creager A. N., Morgan G. J. After the double helix: Rosalind Franklin’s research on Tobacco mosaic virus (англ.) // Isis : journal. — 2008. — Vol. 99, no. 2. — P. 239—272. — doi:10.1086/588626. — PMID 18702397. Архивировано 18 августа 2016 года.
- ↑ Dimmock, 2007, p. 12.
- ↑ Norrby E. Nobel Prizes and the emerging virus concept // Arch. Virol.. — 2008. — Т. 153, № 6. — С. 1109—1123. — doi:10.1007/s00705-008-0088-8. — PMID 18446425.
- ↑ Collier, 1998, p. 745.
- ↑ 1 2 Temin H. M., Baltimore D. RNA-directed DNA synthesis and RNA tumor viruses // Adv. Virus Res.. — 1972. — Т. 17. — С. 129—186. — doi:10.1016/S0065-3527(08)60749-6. — PMID 4348509. Архивировано 6 ноября 2015 года.
- ↑ Barré-Sinoussi, F. et al. Isolation of a T-lymphotropic retrovirus from a patient at risk for acquired immune deficiency syndrome (AIDS) (англ.) // Science : journal. — 1983. — Vol. 220, no. 4599. — P. 868—871. — doi:10.1126/science.6189183. — PMID 6189183. Архивировано 8 сентября 2015 года.
- ↑ 1 2 Cello J., Paul A. V., Wimmer E. Chemical synthesis of poliovirus cDNA: generation of infectious virus in the absence of natural template (англ.) // Science : journal. — 2002. — Vol. 297, no. 5583. — P. 1016—1018. — doi:10.1126/science.1072266. — PMID 12114528.
- ↑ 1 2 Canchaya C., Fournous G., Chibani-Chennoufi S., Dillmann M. L., Brüssow H. Phage as agents of lateral gene transfer // Curr. Opin. Microbiol.. — 2003. — Т. 6, № 4. — С. 417—424. — doi:10.1016/S1369-5274(03)00086-9. — PMID 12941415.
- ↑ Iyer L. M., Balaji S., Koonin E. V., Aravind L. Evolutionary genomics of nucleo-cytoplasmic large DNA viruses (англ.) // Virus Res. : journal. — 2006. — Vol. 117, no. 1. — P. 156—184. — doi:10.1016/j.virusres.2006.01.009. — PMID 16494962. Архивировано 10 августа 2017 года.
- ↑ 1 2 Sanjuán R., Nebot M. R., Chirico N., Mansky L. M., Belshaw R. Viral mutation rates // Journal of Virology. — 2010. — Октябрь (т. 84, № 19). — С. 9733—9748. — doi:10.1128/JVI.00694-10. — PMID 20660197. — PMC 2937809. Архивировано 8 августа 2014 года.
- ↑ Shors, 2008, pp. 14—16.
- ↑ Collier, 1998, pp. 11—21.
- ↑ Dimmock, 2007, p. 16.
- ↑ Collier, 1998, p. 11.
- ↑ 1 2 3 Mahy W. J. & Van Regenmortel M. H. V. (eds). Desk Encyclopedia of General Virology. — Oxford: Academic Press, 2009. — С. 24. — ISBN 0-12-375146-2.
- ↑ Shors, 2008, p. 574.
- ↑ McClintock, B. The origin and behavior of mutable loci in maize // Proc Natl Acad Sci U S A.. — 1950. — Т. 36, № 6. — С. 344—355. — doi:10.1073/pnas.36.6.344. — PMID 15430309. — PMC 1063197.
- ↑ Collier, 1998, pp. 11—12.
- ↑ Dimmock, 2007, p. 55.
- ↑ Shors, 2008, pp. 551—553.
- ↑ Tsagris E. M., de Alba A. E., Gozmanova M., Kalantidis K. Viroids // Cell. Microbiol.. — 2008. — Т. 10, № 11. — С. 2168. — doi:10.1111/j.1462-5822.2008.01231.x. — PMID 18764915.
- ↑ Shors, 2008, pp. 492—493.
- ↑ 1 2 La Scola B., Desnues C., Pagnier I., Robert C., Barrassi L., Fournous G., Merchat M., Suzan-Monti M., Forterre P., Koonin E., Raoult D. The virophage as a unique parasite of the giant mimivirus (англ.) // Nature : journal. — 2008. — Vol. 455, no. 7209. — P. 100—104. — doi:10.1038/nature07218. — PMID 18690211.
- ↑ Collier, 1998, p. 777.
- ↑ Dimmock, 2007, pp. 55—57.
- ↑ 1 2 Mahy W. J. & Van Regenmortel M. H. V. (eds). Desk Encyclopedia of General Virology. — Oxford: Academic Press, 2009. — С. 28. — ISBN 0-12-375146-2.
- ↑ 1 2 Mahy W. J. & Van Regenmortel M. H. V. (eds). Desk Encyclopedia of General Virology. — Oxford: Academic Press, 2009. — С. 26. — ISBN 0-12-375146-2.
- ↑ Dimmock, 2007, pp. 15—16.
- ↑ Liberski P. P. Prion diseases: a riddle wrapped in a mystery inside an enigma (англ.) // Folia Neuropathol : journal. — 2008. — Vol. 46, no. 2. — P. 93—116. — PMID 18587704.
- ↑ Инге-Вечтомов С. Г. Генетика с основами селекции. — СПб.: Издательство Н-Л, 2010. — С. 298. — 718 с. — ISBN 978-5-94869-105-3.
- ↑ Belay E. D. and Schonberger L. B. Desk Encyclopedia of Human and Medical Virology (англ.). — Boston: Academic Press, 2009. — P. 497—504. — ISBN 0-12-375147-0.
- ↑ Lupi O., Dadalti P., Cruz E., Goodheart C. Did the first virus self-assemble from self-replicating prion proteins and RNA? (англ.) // Med. Hypotheses : journal. — 2007. — Vol. 69, no. 4. — P. 724—730. — doi:10.1016/j.mehy.2007.03.031. — PMID 17512677.
- ↑ Holmes E. C. Viral evolution in the genomic age // PLoS Biol.. — 2007. — Т. 5, № 10. — С. e278. — doi:10.1371/journal.pbio.0050278. — PMID 17914905. — PMC 1994994. Архивировано 5 ноября 2013 года.
- ↑ Wimmer E., Mueller S., Tumpey T. M., Taubenberger J. K. Synthetic viruses: a new opportunity to understand and prevent viral disease (англ.) // Nature Biotechnology : journal. — Nature Publishing Group, 2009. — Vol. 27, no. 12. — P. 1163—1172. — doi:10.1038/nbt.1593. — PMID 20010599. — PMC 2819212.
- ↑ Horn M. Chlamydiae as symbionts in eukaryotes // Annual Review of Microbiology. — 2008. — Т. 62. — С. 113—131. — doi:10.1146/annurev.micro.62.081307.162818. — PMID 18473699. Архивировано 11 ноября 2013 года.
- ↑ Ammerman N. C., Beier-Sexton M., Azad A. F. Laboratory maintenance of Rickettsia rickettsii // Current Protocols (англ.) (рус. in Microbiology. — 2008. — Т. Chapter 3. — С. Unit 3A.5. — doi:10.1002/9780471729259.mc03a05s11. — PMID 19016440. — PMC 2725428.
- ↑ Seed K. D., Lazinski D. W., Calderwood S. B., Camilli A. A bacteriophage encodes its own CRISPR/Cas adaptive response to evade host innate immunity (англ.) // Nature : journal. — 2013. — Vol. 494, no. 7438. — P. 489—491. — doi:10.1038/nature11927. — PMID 23446421. — PMC 3587790. Архивировано 5 марта 2016 года.
- ↑ 1 2 Collier, 1998, pp. 33—55.
- ↑ Stefan Sirucek. Ancient “Giant Virus” Revived From Siberian Permafrost, National Geographic (3 марта 2014). Архивировано 25 июня 2018 года. Дата обращения: 3 марта 2014.
- ↑ Collier, 1998, pp. 33—37.
- ↑ Kiselev N. A., Sherman M. B., Tsuprun V. L. Negative staining of proteins // Electron Microsc. Rev.. — 1990. — Т. 3, № 1. — С. 43—72. — doi:10.1016/0892-0354(90)90013-I. — PMID 1715774.
- ↑ Collier, 1998, p. 40.
- ↑ Caspar D. L., Klug A. Physical principles in the construction of regular viruses (англ.) // Cold Spring Harb. Symp. Quant. Biol. : journal. — 1962. — Vol. 27. — P. 1—24. — PMID 14019094. Архивировано 5 марта 2016 года.
- ↑ Crick F. H., Watson J. D. Structure of small viruses (англ.) // Nature. — 1956. — Vol. 177, no. 4506. — P. 473—475. — doi:10.1038/177473a0. — PMID 13309339. Архивировано 5 июля 2013 года.
- ↑ Falvo, M.R.; S. Washburn, R. Superfine, M. Finch, F. P. Brooks Jr, V. Chi, R. M. Taylor 2nd. Manipulation of individual viruses: friction and mechanical properties (англ.) // Biophysical Journal : journal. — 1997. — Vol. 72, no. 3. — P. 1396—1403. — doi:10.1016/S0006-3495(97)78786-1. — PMID 9138585. — PMC 1184522.
- ↑ Kuznetsov, Yu. G.; A. J. Malkin, R. W. Lucas, M. Plomp, A. McPherson. Imaging of viruses by atomic force microscopy // J Gen Virol. — 2001. — Т. 82, № 9. — С. 2025—2034. — PMID 11514711. (недоступная ссылка)
- ↑ Collier, 1998, p. 37.
- ↑ Collier, 1998, pp. 40, 42.
- ↑ Casens, S. Desk Encyclopedia of General Virology. — Boston: Academic Press, 2009. — С. 167—174. — ISBN 0-12-375146-2.
- ↑ Rossmann M. G., Mesyanzhinov V. V., Arisaka F., Leiman P. G. The bacteriophage T4 DNA injection machine (англ.) // Curr. Opin. Struct. Biol.. — 2004. — Vol. 14, no. 2. — P. 171—180. — doi:10.1016/j.sbi.2004.02.001. — PMID 15093831.
- ↑ Collier, 1998, pp. 42—43.
- ↑ Long G. W., Nobel J., Murphy F. A., Herrmann K. L., Lourie B. Experience with electron microscopy in the differential diagnosis of smallpox (англ.) // Appl Microbiol : journal. — 1970. — Vol. 20, no. 3. — P. 497—504. — PMID 4322005. — PMC 376966.
- ↑ Xiao C., Kuznetsov Y. G., Sun S., Hafenstein S. L., Kostyuchenko V. A., Chipman P. R., Suzan-Monti M., Raoult D., McPherson A., Rossmann M. G. Structural studies of the giant mimivirus // PLoS Biol. — 2009. — Т. 7, вып. 4. — С. e92. — doi:10.1371/journal.pbio.1000092. — PMID 19402750. Архивировано 5 июля 2020 года.
- ↑ Klose T., Kuznetsov Y. G., Xiao C., Sun S., McPherson A., Rossmann M. G. The three-dimensional structure of Mimivirus // Intervirology. — 2010. — Т. 53, вып. 5. — С. 268—273. — doi:10.1159/000312911. — PMID 20551678. Архивировано 5 июня 2022 года.
- ↑ World’s biggest virus discovered in ocean depths near Chile. Дата обращения: 12 октября 2011. Архивировано 1 февраля 2013 года.
- ↑ 1 2 3 Collier, 1998, pp. 96—99.
- ↑ Saunders, Venetia A.; Carter, John. Virology: principles and applications. — Chichester: John Wiley & Sons, 2007. — С. 72. — ISBN 0-470-02387-2.
- ↑ Van Etten J. L., Lane L. C., Dunigan D. D. DNA viruses: the really big ones (giruses) // Annual Review of Microbiology. — 2010. — Т. 64. — С. 83—99. — doi:10.1146/annurev.micro.112408.134338. — PMID 20690825. — PMC 2936810. Архивировано 30 сентября 2021 года.
- ↑ Pressing J., Reanney D. C. Divided genomes and intrinsic noise // J Mol Evol. — 1984. — Т. 20, № 2. — С. 135—146. — doi:10.1007/BF02257374. — PMID 6433032.
- ↑ Duffy S., Holmes E. C. Validation of high rates of nucleotide substitution in geminiviruses: phylogenetic evidence from East African cassava mosaic viruses (англ.) // Journal of General Virology (англ.) (рус. : journal. — Microbiology Society (англ.) (рус., 2009. — Vol. 90, no. Pt 6. — P. 1539—1547. — doi:10.1099/vir.0.009266-0. — PMID 19264617. (недоступная ссылка)
- ↑ Pan X. P., Li L. J., Du W. B., Li M. W., Cao H. C., Sheng J. F. Differences of YMDD mutational patterns, precore/core promoter mutations, serum HBV DNA levels in lamivudine-resistant hepatitis B genotypes B and C (англ.) // J. Viral Hepat. : journal. — 2007. — Vol. 14, no. 11. — P. 767—774. — doi:10.1111/j.1365-2893.2007.00869.x. — PMID 17927612. Архивировано 18 марта 2021 года.
- ↑ Hampson A. W., Mackenzie J. S. The influenza viruses // Med. J. Aust.. — 2006. — Т. 185, № 10 Suppl. — С. S39—43. — PMID 17115950. Архивировано 16 мая 2013 года.
- ↑ Metzner K. J. Detection and significance of minority quasispecies of drug-resistant HIV-1 (англ.) // J HIV Ther : journal. — 2006. — Vol. 11, no. 4. — P. 74—81. — PMID 17578210.
- ↑ Goudsmit, Jaap. Viral Sex. Oxford Univ Press, 1998. ISBN 978-0-19-512496-5, ISBN 0-19-512496-0.
- ↑ Worobey M., Holmes E. C. Evolutionary aspects of recombination in RNA viruses (англ.) // Journal of General Virology (англ.) (рус. : journal. — Microbiology Society (англ.) (рус., 1999. — Vol. 80 (Pt 10). — P. 2535—2543. — PMID 10573145. (недоступная ссылка)
- ↑ Lukashev A. N. Role of recombination in evolution of enteroviruses (англ.) // Rev. Med. Virol. : journal. — 2005. — Vol. 15, no. 3. — P. 157—167. — doi:10.1002/rmv.457. — PMID 15578739.
- ↑ Umene K. Mechanism and application of genetic recombination in herpesviruses (англ.) // Rev. Med. Virol. : journal. — 1999. — Vol. 9, no. 3. — P. 171—182. — doi:10.1002/(SICI)1099-1654(199907/09)9:3<171::AID-RMV243>3.0.CO;2-A. — PMID 10479778.
- ↑ Collier, 1998, pp. 75—91.
- ↑ Dimmock, 2007, p. 70.
- ↑ Boevink P., Oparka K. J. Virus-host interactions during movement processes // Plant Physiol.. — 2005. — Т. 138, № 4. — С. 1815—1821. — doi:10.1104/pp.105.066761. — PMID 16172094. — PMC 1183373.
- ↑ Dimmock, 2007, p. 71.
- ↑ Barman S., Ali A., Hui E. K., Adhikary L., Nayak D. P. Transport of viral proteins to the apical membranes and interaction of matrix protein with glycoproteins in the assembly of influenza viruses (англ.) // Virus Res. : journal. — 2001. — Vol. 77, no. 1. — P. 61—69. — doi:10.1016/S0168-1702(01)00266-0. — PMID 11451488.
- ↑ Shors, 2008, pp. 60, 597.
- ↑ Dimmock, 2007, Chapter 15, Mechanisms in virus latentcy, pp. 243—259.
- ↑ Dimmock, 2007, pp. 185—187.
- ↑ Collier, 1998, p. 78.
- ↑ Shors, 2008, p. 54.
- ↑ Collier, 1998, p. 79.
- ↑ Collier, 1998, pp. 88—89.
- ↑ Staginnus C., Richert-Pöggeler K. R. Endogenous pararetroviruses: two-faced travelers in the plant genome (англ.) // Trends in Plant Science : journal. — Cell Press, 2006. — Vol. 11, no. 10. — P. 485—491. — doi:10.1016/j.tplants.2006.08.008. — PMID 16949329.
- ↑ Collier, 1998, pp. 115—146.
- ↑ Collier, 1998, p. 115.
- ↑ Roulston A., Marcellus R. C., Branton P. E. Viruses and apoptosis // Annu. Rev. Microbiol.. — 1999. — Т. 53. — С. 577—628. — doi:10.1146/annurev.micro.53.1.577. — PMID 10547702.
- ↑ Alwine J. C. Modulation of host cell stress responses by human cytomegalovirus (англ.) // Curr. Top. Microbiol. Immunol. : journal. — 2008. — Vol. 325. — P. 263—279. — doi:10.1007/978-3-540-77349-8_15. — PMID 18637511.
- ↑ Sinclair J. Human cytomegalovirus: Latency and reactivation in the myeloid lineage (англ.) // J. Clin. Virol. : journal. — 2008. — Vol. 41, no. 3. — P. 180—185. — doi:10.1016/j.jcv.2007.11.014. — PMID 18164651.
- ↑ Jordan M. C., Jordan G. W., Stevens J. G., Miller G. Latent herpesviruses of humans // Ann. Intern. Med.. — 1984. — Т. 100, № 6. — С. 866—880. — PMID 6326635.
- ↑ Sissons J. G., Bain M., Wills M. R. Latency and reactivation of human cytomegalovirus // J. Infect.. — 2002. — Т. 44, № 2. — С. 73—77. — doi:10.1053/jinf.2001.0948. — PMID 12076064.
- ↑ Barozzi P., Potenza L., Riva G., Vallerini D., Quadrelli C., Bosco R., Forghieri F., Torelli G., Luppi M. B cells and herpesviruses: a model of lymphoproliferation (англ.) // Autoimmun Rev : journal. — 2007. — Vol. 7, no. 2. — P. 132—136. — doi:10.1016/j.autrev.2007.02.018. — PMID 18035323.
- ↑ Subramanya D., Grivas P. D. HPV and cervical cancer: updates on an established relationship (англ.) // Postgrad Med : journal. — 2008. — Vol. 120, no. 4. — P. 7—13. — doi:10.3810/pgm.2008.11.1928. — PMID 19020360.
- ↑ Crawford, Dorothy H. Viruses: A Very Short Introduction (англ.). — Oxford University Press, 2011. — P. 16. — ISBN 0-19-957485-5.
- ↑ Shors, 2008, p. 388.
- ↑ Shors, 2008, p. 353.
- ↑ Dimmock, 2007, p. 272.
- ↑ Baggesen D. L., Sørensen G., Nielsen E. M., Wegener H. C. Phage typing of Salmonella Typhimurium – is it still a useful tool for surveillance and outbreak investigation? (англ.) // Eurosurveillance (англ.) (рус. : journal. — 2010. — Vol. 15, no. 4. — P. 19471. — PMID 20122382. Архивировано 17 февраля 2013 года.
- ↑ Shors, 2008, pp. 49—50.
- ↑ Systema Naturae 2000: Overview. Дата обращения: 17 марта 2013. Архивировано 26 августа 2005 года.
- ↑ Taxonomicon: Aphanobionta. Дата обращения: 17 марта 2013. Архивировано 14 марта 2022 года.
- ↑ Taxonomicon: Acytota. Дата обращения: 17 марта 2013. Архивировано 7 марта 2016 года.
- ↑ Систематика органического мира: Бесклеточные (Acellularia). Дата обращения: 17 марта 2013. Архивировано из оригинала 23 февраля 2012 года.
- ↑ NCBI taxonomy. Дата обращения: 29 сентября 2017. Архивировано 6 апреля 2018 года.
- ↑ Lwoff A., Horne R. W., Tournier P. A virus system (фр.) // C. R. Hebd. Seances Acad. Sci.. — 1962. — Vol. 254. — P. 4225—4227. — PMID 14467544.
- ↑ Lwoff A., Horne R., Tournier P. A system of viruses // Cold Spring Harb. Symp. Quant. Biol.. — 1962. — Т. 27. — С. 51—55. — PMID 13931895.
- ↑ 1 2 3 Wolf Y. I., Kazlauskas D., Iranzo J., Lucía-Sanz A., Kuhn J. H., Krupovic M., Dolja V. V., Koonin E. V. Origins and Evolution of the Global RNA Virome. (англ.) // MBio. — 2018. — 27 November (vol. 9, no. 6). — doi:10.1128/mBio.02329-18. — PMID 30482837. [исправить]
- ↑ 1 2 3 4 5 Koonin E. V., Krupovic M., Agol V. I. The Baltimore Classification of Viruses 50 Years Later: How Does It Stand in the Light of Virus Evolution? (англ.) // Microbiology And Molecular Biology Reviews : MMBR. — 2021. — 18 August (vol. 85, no. 3). — P. e0005321—0005321. — doi:10.1128/MMBR.00053-21. — PMID 34259570. [исправить]
- ↑ Knipe, 2007, p. 27.
- ↑ Delwart E. L. Viral metagenomics (англ.) // Rev. Med. Virol.. — 2007. — Vol. 17, no. 2. — P. 115—131. — doi:10.1002/rmv.532. — PMID 17295196.
- ↑ ICVC&N, 2018, 3.2.
- ↑ ICVC&N, 2018, 3.23.
- ↑ ICVC&N, 2018, 3.3.
- ↑ King A. M. Q., Lefkowitz E., Adams M. J., Carstens E. B. Virus Taxonomy: Ninth Report of the International Committee on Taxonomy of Viruses (англ.). — Elsevier, 2011. — P. 6. — ISBN 0-12-384684-6.
- ↑ Koonin EV, Dolja VV, Krupovic M, Varsani A, Wolf YI, Yutin N, Zerbini M, Kuhn JH. Create a megataxonomic framework, filling all principal taxonomic ranks, for DNA viruses encoding vertical jelly roll-type major capsid proteins (англ.) (docx). International Committee on Taxonomy of Viruses (18 октября 2019). Дата обращения: 10 июня 2020. Архивировано 4 января 2022 года.
- ↑ Koonin, Eugene & Dolja, Valerian & Krupovic, Mart & Varsani, Arvind & Wolf, Yuri & Yutin, Natalya & Zerbini, Francisco & Kuhn, Jens. Create a megataxonomic framework, filling all principal/primary taxonomic ranks, for dsDNA viruses encoding HK97-type major capsid proteins. — 2019. — doi:10.13140/RG.2.2.16564.19842.
- ↑ 1 2 Baltimore D. The strategy of RNA viruses // Harvey Lect.. — 1974. — Т. 70 Series. — С. 57—74. — PMID 4377923.
- ↑ Mayo M. A. Developments in plant virus taxonomy since the publication of the 6th ICTV Report. International Committee on Taxonomy of Viruses (англ.) // Arch. Virol. : journal. — 1999. — Vol. 144, no. 8. — P. 1659—1666. — doi:10.1007/s007050050620. — PMID 10486120.
- ↑ de Villiers E. M., Fauquet C., Broker T. R., Bernard H. U., zur Hausen H. Classification of papillomaviruses // Virology. — 2004. — Т. 324, № 1. — С. 17—27. — doi:10.1016/j.virol.2004.03.033. — PMID 15183049.
- ↑ Temin H. M., Baltimore D. RNA-directed DNA synthesis and RNA tumor viruses // Adv. Virus Res.. — 1972. — Т. 17. — С. 129—186. — PMID 4348509.
- ↑ Lazarowitz SD (2007) «Plant viruses», in «Fields’ Virology», 5th edition, volume 1, pp. 679—683, Wolters Kluwer / Lippincott Williams & Wilkins, ISBN 0-7817-6060-7
- ↑ Seeger C, Zoulin F, Mason WS (2007) «Hepadnaviruses», in «Fields’ Virology», 5th edition, volume 2, pp. 2977-3029, Wolters Kluwer / Lippincott Williams & Wilkins, ISBN 0-7817-6060-7
- ↑ The Big Picture Book of Viruses:Family Groups – The Baltimore Method. Дата обращения: 21 мая 2015. Архивировано 28 апреля 2013 года.
- ↑ Komaroff A. L. Is human herpesvirus-6 a trigger for chronic fatigue syndrome? (англ.) // J. Clin. Virol. : journal. — 2006. — Vol. 37 Suppl 1. — P. S39—46. — doi:10.1016/S1386-6532(06)70010-5. — PMID 17276367.
- ↑ Chen C., Chiu Y., Wei F., Koong F., Liu H., Shaw C., Hwu H., Hsiao K. High seroprevalence of Borna virus infection in schizophrenic patients, family members and mental health workers in Taiwan (англ.) // Mol Psychiatry : journal. — 1999. — Vol. 4, no. 1. — P. 33—38. — doi:10.1038/sj.mp.4000484. — PMID 10089006.
- ↑ Margolis T. P., Elfman F. L., Leib D., et al. Spontaneous reactivation of herpes simplex virus type 1 in latently infected murine sensory ganglia (англ.) // J. Virol. : journal. — 2007. — Vol. 81, no. 20. — P. 11069—11074. — doi:10.1128/JVI.00243-07. — PMID 17686862. — PMC 2045564.
- ↑ Whitley R. J., Roizman B. Herpes simplex virus infections (англ.) // The Lancet. — Elsevier, 2001. — Vol. 357, no. 9267. — P. 1513—1518. — doi:10.1016/S0140-6736(00)04638-9. — PMID 11377626. Архивировано 30 марта 2018 года.
- ↑ Barton E. S., White D. W., Cathelyn J. S., et al. Herpesvirus latency confers symbiotic protection from bacterial infection (англ.) // Nature : journal. — 2007. — Vol. 447, no. 7142. — P. 326—329. — doi:10.1038/nature05762. — PMID 17507983.
- ↑ Bertoletti A., Gehring A. Immune response and tolerance during chronic hepatitis B virus infection (англ.) // Hepatol. Res. : journal. — 2007. — Vol. 37 Suppl 3. — P. S331—S338. — doi:10.1111/j.1872-034X.2007.00221.x. — PMID 17931183.
- ↑ Rodrigues C., Deshmukh M., Jacob T., Nukala R., Menon S., Mehta A. Significance of HBV DNA by PCR over serological markers of HBV in acute and chronic patients (англ.) // Indian journal of medical microbiology : journal. — 2001. — Vol. 19, no. 3. — P. 141—144. — PMID 17664817. Архивировано 8 января 2007 года.
- ↑ Nguyen V. T., McLaws M. L., Dore G. J. Highly endemic hepatitis B infection in rural Vietnam (англ.) // Journal of Gastroenterology and Hepatology : journal. — 2007. — Vol. 22, no. 12. — P. 2093—2100. — doi:10.1111/j.1440-1746.2007.05010.x. — PMID 17645465.
- ↑ Fowler M. G., Lampe M. A., Jamieson D. J., Kourtis A. P., Rogers M. F. Reducing the risk of mother-to-child human immunodeficiency virus transmission: past successes, current progress and challenges, and future directions (англ.) // American Journal of Obstetrics and Gynecology (англ.) (рус. : journal. — Elsevier, 2007. — Vol. 197, no. 3 Suppl. — P. S3—9. — doi:10.1016/j.ajog.2007.06.048. — PMID 17825648.
- ↑ Sauerbrei A., Wutzler P. The congenital varicella syndrome // Journal of perinatology : official journal of the California Perinatal Association. — 2000. — Т. 20, № 8 Pt 1. — С. 548—554. — PMID 11190597.
- ↑ Garnett G. P. Role of herd immunity in determining the effect of vaccines against sexually transmitted disease (англ.) // J. Infect. Dis. : journal. — 2005. — Vol. 191 Suppl 1. — P. S97—106. — doi:10.1086/425271. — PMID 15627236. (недоступная ссылка)
- ↑ Platonov A. E. Влияние погодных условий на эпидемиологию трансмиссивных инфекций (на примере лихорадки Западного Нила в России) // Vestn. Akad. Med. Nauk SSSR. — 2006. — № 2. — С. 25—29. — PMID 16544901.
- ↑ Shors, 2008, p. 198.
- ↑ Shors, 2008, pp. 199, 209.
- ↑ Shors, 2008, p. 19.
- ↑ Shors, 2008, p. 126.
- ↑ 1 2 Shors, 2008, pp. 193—194.
- ↑ Shors, 2008, p. 194.
- ↑ Shors, 2008, pp. 192—193.
- ↑ * Ranlet P. The British, the Indians, and smallpox: what actually happened at Fort Pitt in 1763? (англ.) // Pa Hist : journal. — 2000. — Vol. 67, no. 3. — P. 427—441. — PMID 17216901.
- Van Rijn K. “Lo! The poor Indian!” colonial responses to the 1862–63 smallpox epidemic in British Columbia and Vancouver Island (англ.) // Can Bull Med Hist : journal. — 2006. — Vol. 23, no. 2. — P. 541—560. — PMID 17214129.
- Patterson K. B., Runge T. Smallpox and the Native American // Am. J. Med. Sci.. — 2002. — Т. 323, № 4. — С. 216—222. — doi:10.1097/00000441-200204000-00009. — PMID 12003378.
- Sessa R., Palagiano C., Scifoni M. G., di Pietro M., Del Piano M. The major epidemic infections: a gift from the Old World to the New? (англ.) // Panminerva Med : journal. — 1999. — Vol. 41, no. 1. — P. 78—84. — PMID 10230264.
- Bianchine P. J., Russo T. A. The role of epidemic infectious diseases in the discovery of America (англ.) // Allergy Proc : journal. — 1992. — Vol. 13, no. 5. — P. 225—232. — doi:10.2500/108854192778817040. — PMID 1483570. Архивировано 6 декабря 2012 года.
- Hauptman L. M. Smallpox and American Indian; Depopulation in Colonial New York (англ.) // N Y State J Med : journal. — 1979. — Vol. 79, no. 12. — P. 1945—1949. — PMID 390434.
- Fortuine R. Smallpox decimates the Tlingit (1787) (англ.) // Alaska Med. — 1988. — Vol. 30, no. 3. — P. 109. — PMID 3041871.
- ↑ Collier, 1998, pp. 409—415.
- ↑ Patterson K. D., Pyle G. F. The geography and mortality of the 1918 influenza pandemic (англ.) // Bull Hist Med. : journal. — 1991. — Vol. 65, no. 1. — P. 4—21. — PMID 2021692.
- ↑ Johnson N. P., Mueller J. Updating the accounts: global mortality of the 1918–1920 “Spanish” influenza pandemic (англ.) // Bull Hist Med : journal. — 2002. — Vol. 76, no. 1. — P. 105—115. — doi:10.1353/bhm.2002.0022. — PMID 11875246.
- ↑ Gao F., Bailes E., Robertson D. L., et al. Origin of HIV-1 in the Chimpanzee Pan troglodytes troglodytes (англ.) // Nature : journal. — 1999. — Vol. 397, no. 6718. — P. 436—441. — doi:10.1038/17130. — PMID 9989410. Архивировано 23 февраля 2005 года.
- ↑ Shors, 2008, p. 447.
- ↑ Mawar N., Saha S., Pandit A., Mahajan U. The third phase of HIV pandemic: social consequences of HIV/AIDS stigma & discrimination & future needs (англ.) // Indian J. Med. Res. : journal. — 2005. — Vol. 122, no. 6. — P. 471—484. — PMID 16517997. Архивировано 4 марта 2016 года. Архивированная копия. Дата обращения: 27 февраля 2013. Архивировано из оригинала 4 марта 2016 года.
- ↑ Status of the global HIV epidemic (PDF) (недоступная ссылка — история). UNAIDS (2008). Дата обращения: 15 сентября 2008. Архивировано 9 марта 2013 года.
- ↑ Towner J. S., Khristova M. L., Sealy T. K., et al. Marburgvirus genomics and association with a large hemorrhagic fever outbreak in Angola (англ.) // J. Virol. : journal. — 2006. — Vol. 80, no. 13. — P. 6497—6516. — doi:10.1128/JVI.00069-06. — PMID 16775337. — PMC 1488971. Архивировано 25 июля 2018 года.
- ↑ Einstein M. H., Schiller J. T., Viscidi R. P., Strickler H. D., Coursaget P., Tan T., Halsey N., Jenkins D. Clinician’s guide to human papillomavirus immunology: knowns and unknowns (англ.) // The Lancet : journal. — Elsevier, 2009. — Vol. 9, no. 6. — P. 347—356. — doi:10.1016/S1473-3099(09)70108-2. — PMID 19467474.
- ↑ Shuda M., Feng H., Kwun H. J., Rosen S. T., Gjoerup O., Moore P. S., Chang Y. T antigen mutations are a human tumor-specific signature for Merkel cell polyomavirus (англ.) // Proceedings of the National Academy of Sciences : journal. — United States National Academy of Sciences, 2008. — Vol. 105, no. 42. — P. 16272—16277. — doi:10.1073/pnas.0806526105. — PMID 18812503. — PMC 2551627.
- ↑ Pulitzer M. P., Amin B. D., Busam K. J. Merkel cell carcinoma: review // Advances in Anatomic Pathology. — 2009. — Т. 16, № 3. — С. 135—144. — doi:10.1097/PAP.0b013e3181a12f5a. — PMID 19395876.
- ↑ Koike K. Hepatitis C virus contributes to hepatocarcinogenesis by modulating metabolic and intracellular signalling pathways (англ.) // J. Gastroenterol. Hepatol. : journal. — 2007. — Vol. 22 Suppl 1. — P. S108—S111. — doi:10.1111/j.1440-1746.2006.04669.x. — PMID 17567457.
- ↑ Hu J., Ludgate L. HIV-HBV and HIV-HCV coinfection and liver cancer development (англ.) // Cancer Treat. Res. : journal. — 2007. — Vol. 133. — P. 241—252. — doi:10.1007/978-0-387-46816-7_9. — PMID 17672044.
- ↑ Bellon M., Nicot C. Telomerase: a crucial player in HTLV-I-induced human T-cell leukemia (англ.) // Cancer genomics & proteomics : journal. — 2007. — Vol. 4, no. 1. — P. 21—25. — PMID 17726237.
- ↑ Schiffman M., Castle P. E., Jeronimo J., Rodriguez A. C., Wacholder S. Human papillomavirus and cervical cancer (англ.) // The Lancet. — Elsevier, 2007. — Vol. 370, no. 9590. — P. 890—907. — doi:10.1016/S0140-6736(07)61416-0. — PMID 17826171.
- ↑ Klein E., Kis L. L., Klein G. Epstein-Barr virus infection in humans: from harmless to life endangering virus-lymphocyte interactions (англ.) // Oncogene (англ.) (рус. : journal. — 2007. — Vol. 26, no. 9. — P. 1297—1305. — doi:10.1038/sj.onc.1210240. — PMID 17322915.
- ↑ zur Hausen H. Novel human polyomaviruses—re-emergence of a well known virus family as possible human carcinogens (англ.) // International Journal of Cancer. Journal International Du Cancer : journal. — 2008. — Vol. 123, no. 2. — P. 247—250. — doi:10.1002/ijc.23620. — PMID 18449881.
- ↑ Alberts, Bruce; Alexander Johnson, Julian Lewis, Martin Raff, Keith Roberts, and Peter Walters. Molecular Biology of the Cell; Fourth Edition (англ.). — New York and London: Garland Science (англ.) (рус., 2002. — ISBN 0-8153-3218-1. Архивная копия от 18 сентября 2009 на Wayback Machine
- ↑ Ding S. W., Voinnet O. Antiviral immunity directed by small RNAs (англ.) // Cell. — Cell Press, 2007. — Vol. 130, no. 3. — P. 413—426. — doi:10.1016/j.cell.2007.07.039. — PMID 17693253. — PMC 2703654.
- ↑ Patton J. T., Vasquez-Del Carpio R., Spencer E. Replication and transcription of the rotavirus genome (англ.) // Curr. Pharm. Des. : journal. — 2004. — Vol. 10, no. 30. — P. 3769—3777. — doi:10.2174/1381612043382620. — PMID 15579070.
- ↑ Jayaram H., Estes M. K., Prasad B. V. Emerging themes in rotavirus cell entry, genome organization, transcription and replication (англ.) // Virus Res. : journal. — 2004. — Vol. 101, no. 1. — P. 67—81. — doi:10.1016/j.virusres.2003.12.007. — PMID 15010218.
- ↑ Greer S., Alexander G. J. Viral serology and detection // Baillieres Clin. Gastroenterol.. — 1995. — Т. 9, № 4. — С. 689—721. — doi:10.1016/0950-3528(95)90057-8. — PMID 8903801.
- ↑ Matter L., Kogelschatz K., Germann D. Serum levels of rubella virus antibodies indicating immunity: response to vaccination of subjects with low or undetectable antibody concentrations (англ.) // J. Infect. Dis. : journal. — 1997. — Vol. 175, no. 4. — P. 749—755. — doi:10.1086/513967. — PMID 9086126.
- ↑ Mallery D. L., McEwan W. A., Bidgood S. R., Towers G. J., Johnson C. M., James L. C. Antibodies mediate intracellular immunity through tripartite motif-containing 21 (TRIM21) (англ.) // Proceedings of the National Academy of Sciences : journal. — United States National Academy of Sciences, 2010. — November (vol. 107, no. 46). — P. 19985—19990. — doi:10.1073/pnas.1014074107. — PMID 21045130. — PMC 2993423.
- ↑ Cascalho M., Platt J. L. Novel functions of B cells // Crit. Rev. Immunol.. — 2007. — Т. 27, № 2. — С. 141—151. — PMID 17725500.
- ↑ Le Page C., Génin P., Baines M. G., Hiscott J. Interferon activation and innate immunity // Rev Immunogenet. — 2000. — Т. 2, № 3. — С. 374—386. — PMID 11256746.
- ↑ Maurice R. Hilleman. Strategies and mechanisms for host and pathogen survival in acute and persistent viral infections (англ.) // Proceedings of the National Academy of Sciences. — National Academy of Sciences, 2004-10-05. — Vol. 101, iss. suppl 2. — P. 14560—14566. — doi:10.1073/pnas.0404758101.
- ↑ Asaria P., MacMahon E. Measles in the United Kingdom: can we eradicate it by 2010? (англ.) // BMJ : journal. — 2006. — Vol. 333, no. 7574. — P. 890—895. — doi:10.1136/bmj.38989.445845.7C. — PMID 17068034. — PMC 1626346. Архивировано 7 марта 2016 года.
- ↑ Lane J. M. Mass vaccination and surveillance/containment in the eradication of smallpox (англ.) // Curr. Top. Microbiol. Immunol. : journal. — 2006. — Vol. 304. — P. 17—29. — doi:10.1007/3-540-36583-4_2. — PMID 16989262.
- ↑ Arvin A. M., Greenberg H. B. New viral vaccines // Virology. — 2006. — Т. 344, № 1. — С. 240—249. — doi:10.1016/j.virol.2005.09.057. — PMID 16364754.
- ↑ Pastoret P. P., Schudel A. A., Lombard M. Conclusions—future trends in veterinary vaccinology (англ.) // Rev. – Off. Int. Epizoot. : journal. — 2007. — Vol. 26, no. 2. — P. 489—494. — PMID 17892169.
- ↑ Palese P. Making better influenza virus vaccines? // Emerging Infect. Dis.. — 2006. — Т. 12, № 1. — С. 61—65. — doi:10.3201/eid1201.051043. — PMID 16494719. Архивировано 2 октября 2012 года.
- ↑ Thomssen R. Live attenuated versus killed virus vaccines // Monographs in allergy. — 1975. — Т. 9. — С. 155—176. — PMID 1090805.
- ↑ McLean A. A. Development of vaccines against hepatitis A and hepatitis B (англ.) // Rev. Infect. Dis. : journal. — 1986. — Vol. 8, no. 4. — P. 591—598. — PMID 3018891.
- ↑ Casswall T. H., Fischler B. Vaccination of the immunocompromised child // Expert review of vaccines. — 2005. — Т. 4, № 5. — С. 725—738. — doi:10.1586/14760584.4.5.725. — PMID 16221073.
- ↑ Barnett E. D., Wilder-Smith A., Wilson M. E. Yellow fever vaccines and international travelers // Expert Rev Vaccines. — 2008. — Т. 7, № 5. — С. 579—587. — doi:10.1586/14760584.7.5.579. — PMID 18564013.
- ↑ Magden J., Kääriäinen L., Ahola T. Inhibitors of virus replication: recent developments and prospects (англ.) // Appl. Microbiol. Biotechnol. : journal. — 2005. — Vol. 66, no. 6. — P. 612—621. — doi:10.1007/s00253-004-1783-3. — PMID 15592828.
- ↑ Mindel A., Sutherland S. Genital herpes — the disease and its treatment including intravenous acyclovir (англ.) // J. Antimicrob. Chemother. : journal. — 1983. — Vol. 12 Suppl B. — P. 51—59. — PMID 6355051.
- ↑ Witthöft T., Möller B., Wiedmann K. H., et al. Safety, tolerability and efficacy of peginterferon alpha-2a and ribavirin in chronic hepatitis C in clinical practice: The German Open Safety Trial (англ.) // J. Viral Hepat. : journal. — 2007. — Vol. 14, no. 11. — P. 788—796. — doi:10.1111/j.1365-2893.2007.00871.x. — PMID 17927615. — PMC 2156112.
- ↑ Rudin D., Shah S. M., Kiss A., Wetz R. V., Sottile V. M. Interferon and lamivudine vs. interferon for hepatitis B e antigen-positive hepatitis B treatment: meta-analysis of randomized controlled trials (англ.) // Liver Int. : journal. — 2007. — Vol. 27, no. 9. — P. 1185—1193. — doi:10.1111/j.1478-3231.2007.01580.x. — PMID 17919229. — PMC 2156150.
- ↑ Dimmock, 2007, p. 3.
- ↑ Goris N., Vandenbussche F., De Clercq K. Potential of antiviral therapy and prophylaxis for controlling RNA viral infections of livestock (англ.) // Antiviral Res. : journal. — 2008. — Vol. 78, no. 1. — P. 170—178. — doi:10.1016/j.antiviral.2007.10.003. — PMID 18035428.
- ↑ Carmichael L. An annotated historical account of canine parvovirus (англ.) // J. Vet. Med. B Infect. Dis. Vet. Public Health : journal. — 2005. — Vol. 52, no. 7—8. — P. 303—311. — doi:10.1111/j.1439-0450.2005.00868.x. — PMID 16316389.
- ↑ Tinsley T. W., Harrap K. A. Viruses of Invertebrates (англ.). Дата обращения: 27 февраля 2013. Архивировано 7 июня 2018 года.
- ↑ Invertebrate iridescent viruses (Iridoviridae) (англ.). Дата обращения: 27 февраля 2013. Архивировано 13 сентября 2019 года.
- ↑ Вирусы беспозвоночных. Дата обращения: 27 февраля 2013. Архивировано 24 февраля 2020 года.
- ↑ Chen Y. P., Zhao Y., Hammond J., Hsu H., Evans J. D., Feldlaufer M. F. Multiple virus infections in the honey bee and genome divergence of honey bee viruses (англ.) // Journal of Invertebrate Pathology : journal. — Vol. 87, no. 2—3. — P. 84—93. — doi:10.1016/j.jip.2004.07.005. — PMID 15579317.
- ↑ Rybicki EP (2015) «A Top Ten list for economically important plant viruses», Archives of Virology 160:17-20
- ↑ Shors, 2008, p. 584.
- ↑ Shors, 2008, pp. 562—587.
- ↑ Dinesh-Kumar S. P., Tham Wai-Hong, Baker B. J. Structure—function analysis of the tobacco mosaic virus resistance gene N (англ.) // Proceedings of the National Academy of Sciences : journal. — National Academy of Sciences, 2000. — Vol. 97, no. 26. — P. 14789—14794. — doi:10.1073/pnas.97.26.14789. — PMID 11121079. — PMC 18997. Архивировано 19 марта 2017 года.
- ↑ Shors, 2008, pp. 573—576.
- ↑ Soosaar J. L., Burch-Smith T. M., Dinesh-Kumar S. P. Mechanisms of plant resistance to viruses // Nat. Rev. Microbiol. — 2005. — Т. 3, № 10. — С. 789—798. — doi:10.1038/nrmicro1239. — PMID 16132037.
- ↑ Lomonossoff G. P. Virus Particles and the Uses of Such Particles in Bio- and Nanotechnology // Recent Advances in Plant Virology. — Caister Academic Press (англ.) (рус., 2011. — ISBN 978-1-904455-75-2.
- ↑ Упадышев М. Т. Оздоровление садовых растений от вирусов // Защита и карантин растений. — 2012-01-01. — Вып. 5. — ISSN 1026-8634. (недоступная ссылка)
- ↑ 1 2 3 Вирусы грибов. Дата обращения: 27 февраля 2013. Архивировано 5 сентября 2019 года.
- ↑ McGraw-Hill Science & Technology Encyclopedia: Fungal virus (англ.). Дата обращения: 27 февраля 2013. Архивировано 4 марта 2016 года.
- ↑ Дьяков Ю. Т., Шнырева А. В., Сергеев А. Ю. Введение в генетику грибов. — М.: изд. центр «Академия», 2005. — С. 57—58. — 304 с. — ISBN 5-7695-2174-0.
- ↑ Белякова Г. А., Дьяков Ю. Т., Тарасов К. Л. Ботаника: в 4 томах. — М.: изд. центр «Академия», 2006. — Т. 1. Водоросли и грибы. — С. 73. — 320 с. — ISBN 5-7695-2731-5.
- ↑ International Society for Viruses of Microorganisms: Protist Viruses (англ.). Дата обращения: 28 февраля 2013. (недоступная ссылка)
- ↑ Philippe Colson, Gregory Gimenez, Mickaël Boyer, Ghislain Fournous, Didier Raoult. The Giant Cafeteria roenbergensis Virus That Infects a Widespread Marine Phagocytic Protist Is a New Member of the Fourth Domain of Life (англ.). Архивировано 31 мая 2020 года.
- ↑ Wommack K. E., Colwell R. R. Virioplankton: viruses in aquatic ecosystems // Microbiol. Mol. Biol. Rev.. — 2000. — Т. 64, № 1. — С. 69—114. — doi:10.1128/MMBR.64.1.69-114.2000. — PMID 10704475. — PMC 98987.
- ↑ Bergh O., Børsheim K. Y., Bratbak G., Heldal M. High abundance of viruses found in aquatic environments (англ.) // Nature : journal. — 1989. — Vol. 340, no. 6233. — P. 467—468. — doi:10.1038/340467a0. — PMID 2755508.
- ↑ Shors, 2008, pp. 595—597.
- ↑ Bickle T. A., Krüger D. H. Biology of DNA restriction (англ.) // Microbiology and Molecular Biology Reviews (англ.) (рус.. — American Society for Microbiology (англ.) (рус., 1993. — 1 June (vol. 57, no. 2). — P. 434—450. — PMID 8336674. — PMC 372918.
- ↑ Barrangou R., Fremaux C., Deveau H. et al. CRISPR provides acquired resistance against viruses in prokaryotes (англ.) // Science. — 2007. — Vol. 315, no. 5819. — P. 1709—1712. — doi:10.1126/science.1138140. — PMID 17379808.
- ↑ Brouns S. J., Jore M. M., Lundgren M., et al. Small CRISPR RNAs guide antiviral defense in prokaryotes (англ.) // Science. — 2008. — Vol. 321, no. 5891. — P. 960—964. — doi:10.1126/science.1159689. — PMID 18703739.
- ↑ В. М. Жданов. Человек и вирусы // Наука и человечество, 1984. — М.: Знание. — С. 44—55.
- ↑ Xiang X., Chen L., Huang X., Luo Y., She Q., Huang L. Sulfolobus tengchongensis spindle-shaped virus STSV1: virus-host interactions and genomic features (англ.) // J. Virol. : journal. — 2005. — Vol. 79, no. 14. — P. 8677—8686. — doi:10.1128/JVI.79.14.8677-8686.2005. — PMID 15994761. — PMC 1168784. Архивировано 16 октября 2019 года.
- ↑ Robb F., Antranikian G., Grogan D., Driessen A. (Editors) (2007) «Thermophiles: Biology and Technology at High Temperatures», CRC Press, ISBN 978-0849392146, pp. 231—232
- ↑ Krupovic M., Quemin E. R. J., Bamford D. H., Forterre P., Prangishvili D. Unification of the Globally Distributed Spindle-Shaped Viruses of the Archaea // Journal of Virology. — 2013. — 11 декабря (т. 88, № 4). — С. 2354—2358. — ISSN 0022-538X. — doi:10.1128/JVI.02941-13. [исправить]
- ↑ Hochstein Rebecca, Bollschweiler Daniel, Engelhardt Harald, Lawrence C. Martin, Young Mark. Large Tailed Spindle Viruses of Archaea: a New Way of Doing Viral Business // Journal of Virology. — 2015. — 17 июня (т. 89, № 18). — С. 9146—9149. — ISSN 0022-538X. — doi:10.1128/JVI.00612-15. [исправить]
- ↑ Uneven Distribution of Viruses Suggests Surprising Evolutionary Power Архивная копия от 8 августа 2017 на Wayback Machine // Scientific American
- ↑ Prangishvili D., Forterre P., Garrett R. A. Viruses of the Archaea: a unifying view // Nat. Rev. Microbiol.. — 2006. — Т. 4, № 11. — С. 837—848. — doi:10.1038/nrmicro1527. — PMID 17041631.
- ↑ Prangishvili D., Garrett R.A. Exceptionally diverse morphotypes and genomes of crenarchaeal hyperthermophilic viruses (англ.) // Biochem. Soc. Trans. : journal. — 2004. — Vol. 32, no. Pt 2. — P. 204—208. — doi:10.1042/BST0320204. — PMID 15046572. (недоступная ссылка)
- ↑ Mojica F. J., Díez-Villaseñor C., García-Martínez J., Soria E. Intervening sequences of regularly spaced prokaryotic repeats derive from foreign genetic elements (англ.) // J. Mol. Evol. : journal. — 2005. — Vol. 60, no. 2. — P. 174—182. — doi:10.1007/s00239-004-0046-3. — PMID 15791728.
- ↑ Makarova K. S., Grishin N. V., Shabalina S. A., Wolf Y. I., Koonin E. V. A putative RNA-interference-based immune system in prokaryotes: computational analysis of the predicted enzymatic machinery, functional analogies with eukaryotic RNAi, and hypothetical mechanisms of action (англ.) // Biol. Direct : journal. — 2006. — Vol. 1. — P. 7. — doi:10.1186/1745-6150-1-7. — PMID 16545108. — PMC 1462988. Архивировано 10 мая 2013 года.
- ↑ 1 2 La Scola B., Desnues C., Pagnier I., Robert C., Barrassi L., Fournous G., Merchat M., Suzan-Monti M., Forterre P., Koonin E., Raoult D. The virophage as a unique parasite of the giant mimivirus (англ.) // Nature. — 2008. — Vol. 455, iss. 7209. — P. 100—104. — doi:10.1038/nature07218. — PMID 18690211.
- ↑ Helen Pearson. ‘Virophage’ suggests viruses are alive (2008). Дата обращения: 2 февраля 2013. Архивировано 10 февраля 2013 года.
- ↑ Александр Марков. Вирусы тоже болеют вирусными заболеваниями (8 сентября 2008). Дата обращения: 2 февраля 2013. Архивировано 10 февраля 2013 года.
- ↑ Desnues C., Boyer M., Raoult D. Sputnik, a virophage infecting the viral domain of life // Adv Virus Res. — 2012. — Т. 82. — С. 63—89. — doi:10.1016/B978-0-12-394621-8.00013-3. — PMID 22420851.
- ↑ Claverie J. M., Abergel C. Mimivirus and its virophage // Annu Rev Genet. — 2009. — Т. 43. — С. 49—66. — doi:10.1146/annurev-genet-102108-134255. — PMID 19653859.
- ↑ Zhou Jinglie, Zhang Weijia, Yan Shuling, Xiao Jinzhou, Zhang Yuanyuan, Li Bailin, Pan Yingjie, Wang Yongjie. Diversity of Virophages in Metagenomic Data Sets // Journal of Virology. — 2013. — Vol. 87, no. 8. — P. 4225—4236. — doi:10.1128/JVI.03398-12. — PMID 23408616. [исправить]
- ↑ Zhang X., Sun S., Xiang Y., Wong J., Klose T., Raoult D., Rossmann M. G. Structure of Sputnik, a virophage, at 3.5-Å resolution. (англ.) // Proceedings of the National Academy of Sciences of the United States of America. — 2012. — Vol. 109, no. 45. — P. 18431—18436. — doi:10.1073/pnas.1211702109. — PMID 23091035. [исправить]
- ↑ La Scola B., Campocasso A., N’Dong R., Fournous G., Barrassi L., Flaudrops C., Raoult D. Tentative characterization of new environmental giant viruses by MALDI-TOF mass spectrometry // Intervirology. — 2010. — Т. 53, вып. 5. — С. 344—353. — doi:10.1159/000312919. — PMID 20551686.
- ↑ Bekliz M., Colson P., La Scola B. The Expanding Family of Virophages // Viruses. — 2016. — Vol. 8, no. 11. — doi:10.3390/v8110317. — PMID 27886075. [исправить]
- ↑ ScienceNow — «Ancient Virus Gave Wasps Their Sting». Дата обращения: 26 марта 2011. Архивировано из оригинала 15 апреля 2011 года.
- ↑ Элементы — новости науки: Наездники подавляют иммунную защиту своих жертв при помощи прирученных вирусов. Дата обращения: 19 августа 2009. Архивировано 17 ноября 2016 года.
- ↑ Breitbart M. Marine viruses: truth or dare // Annual Review of Marine Science. — 2012. — Т. 4. — С. 425—448. — PMID 22457982.
- ↑ Shors, 2008, p. 4.
- ↑ Shors, 2008, p. 5.
- ↑ Shors, 2008, p. 593.
- ↑ Suttle C. A. Viruses in the sea (англ.) // Nature. — 2005. — Vol. 437, no. 7057. — P. 356—361. — doi:10.1038/nature04160. — PMID 16163346. (недоступная ссылка)
- ↑ National Center for Environmental Health. Harmful Algal Blooms (англ.) (недоступная ссылка — история). Centers for disease control and prevention (24 июля 2012). Дата обращения: 8 марта 2013. Архивировано 21 марта 2013 года.
- ↑ 1 2 3 4 Suttle C. A. Marine viruses—major players in the global ecosystem (англ.) // Nature Reviews. Microbiology : journal. — 2007. — Vol. 5, no. 10. — P. 801—812. — doi:10.1038/nrmicro1750. — PMID 17853907. Архивировано 4 марта 2016 года.
- ↑ Hall, A. J., Jepson, P. D., Goodman, S. J. & Harkonen, T. «Phocine distemper virus in the North and European Seas — data and models, nature and nurture». Biol. Conserv. 131, 221—229 (2006).
- ↑ Forterre P., Philippe H. The last universal common ancestor (LUCA), simple or complex? (англ.) // The Biological Bulletin : journal. — 1999. — Vol. 196, no. 3. — P. 373—375. — doi:10.2307/1542973. — PMID 11536914.
- ↑ Collier, 1998, p. 8.
- ↑ Lodish, Harvey; Berk, Arnold; Zipursky, S. Lawrence; Matsudaira, Paul; Baltimore, David; Darnell, James. Viruses:Structure, Function, and Uses Retrieved on September 16, 2008
- ↑ Matsuzaki S., Rashel M., Uchiyama J., Sakurai S., Ujihara T., Kuroda M., Ikeuchi M., Tani T., Fujieda M., Wakiguchi H., Imai S. Bacteriophage therapy: a revitalized therapy against bacterial infectious diseases (англ.) // Journal of Infection and Chemotherapy : Official Journal of the Japan Society of Chemotherapy : journal. — 2005. — October (vol. 11, no. 5). — P. 211—219. — doi:10.1007/s10156-005-0408-9. — PMID 16258815.
- ↑ Gleba, Y. Y.; Giritch, A. Plant Viral Vectors for Protein Expression // Recent Advances in Plant Virology. — Caister Academic Press (англ.) (рус., 2011. — ISBN 978-1-904455-75-2.
- ↑ Fischlechner M., Donath E. Viruses as Building Blocks for Materials and Devices (англ.) // Angewandte Chemie International Edition : journal. — 2007. — Vol. 46, no. 18. — P. 3184—3193. — doi:10.1002/anie.200603445. — PMID 17348058.
- ↑ Soto C. M., Blum A. S., Vora G. J., et al. Fluorescent signal amplification of carbocyanine dyes using engineered viral nanoparticles (англ.) // J. Am. Chem. Soc. : journal. — 2006. — Vol. 128, no. 15. — P. 5184—5189. — doi:10.1021/ja058574x. — PMID 16608355.
- ↑ Blum A. S., Soto C. M., Wilson C. D. et al. An Engineered Virus as a Scaffold for Three-Dimensional Self-Assembly on the Nanoscale (англ.) // Small : journal. — 2005. — Vol. 7. — P. 702. — doi:10.1002/smll.200500021. — PMID 17193509.
- ↑ Coleman J. R., Papamichail D., Skiena S., Futcher B., Wimmer E., Mueller S. Virus attenuation by genome-scale changes in codon pair bias (англ.) // Science : journal. — 2008. — Vol. 320, no. 5884. — P. 1784—1787. — doi:10.1126/science.1155761. — PMID 18583614. — PMC 2754401.
- ↑ Genomes. NIH viral genome database. Ncbi.nlm.nih.gov. Дата обращения: 7 мая 2012. Архивировано 14 февраля 2014 года.
- ↑ Shors, 2008, p. 331.
- ↑ 1 2 3 Artenstein A. W., Grabenstein J. D. Smallpox vaccines for biodefense: need and feasibility (англ.) // Expert Review of Vaccines : journal. — 2008. — Vol. 7, no. 8. — P. 1225—1237. — doi:10.1586/14760584.7.8.1225. — PMID 18844596. Архивировано 2 июля 2016 года.
- ↑ Aragón T. J., Ulrich S., Fernyak S., Rutherford G. W. Risks of serious complications and death from smallpox vaccination: a systematic review of the United States experience, 1963–1968 (англ.) // BMC public health : journal. — 2003. — Vol. 3. — P. 26. — doi:10.1186/1471-2458-3-26. — PMID 12911836. — PMC 194634. Архивировано 4 октября 2013 года.
- ↑ Weiss M. M., Weiss P. D., Mathisen G., Guze P. Rethinking smallpox // Clin. Infect. Dis.. — 2004. — Т. 39, № 11. — С. 1668—1673. — doi:10.1086/425745. — PMID 15578369.
- ↑ 1 2 3 4 Georgios Pappas, Savvas Seitaridis, Nikolaos Akritidis, Epaminondas Tsianos. Infectious Diseases in Cinema: Virus Hunters and Killer Microbes (англ.) // Clinical Infectious Diseases : Oxford Journals. — 2003. — Vol. 37, no. 7. — P. 939—942. — doi:10.1086/377740. Архивировано 20 октября 2014 года.
Литература[править | править код]
- Букринская А. Г. Вирусология : Учеб. пособие. — М.: Медицина, 1986. — 336 с.
- Зуев В. А. Многоликий вирус. — М.: АСТ, 2020. — 304 с. — (Наука и жизнь). — 3000 экз. — ISBN 978-5-17-118736-1.
- Mayo M. A., Pringle C. R. Virus taxonomy — 1997 (англ.) // Journal of General Virology (англ.) (рус.. — Microbiology Society (англ.) (рус., 1998. — No. 79. — P. 649—657. Архивировано 29 сентября 2007 года.
- Collier, Leslie; Balows, Albert; Sussman, Max. Topley and Wilson’s Microbiology and Microbial Infections / Mahy, Brian and Collier, Leslie. Arnold. — ninth edition. — Virology, 1998. — Т. 1. — ISBN 0-340-66316-2.
- Dimmock N. J., Easton, Andrew J.; Leppard, Keith. Introduction to Modern Virology. — sixth edition. — Blackwell Publishing, 2007. — ISBN 1-4051-3645-6.
- Knipe, David M.; Howley, Peter M.; Griffin, Diane E.; Lamb, Robert A.; Martin, Malcolm A.; Roizman, Bernard; Straus Stephen E. Fields Virology. — Lippincott Williams & Wilkins., 2007. — ISBN 0-7817-6060-7.
- Shors, Teri. Understanding Viruses. — Jones and Bartlett Publishers, 2008. — ISBN 0-7637-2932-9.
- The International Code of Virus Classification and Nomenclature : October 2018 : [англ.] // ICTV. — 2018. (Дата обращения: 17 мая 2020).
Ссылки[править | править код]
- The Universal Virus Database ICTVdB. Дата обращения: 13 декабря 2007. Архивировано из оригинала 13 декабря 2007 года.
- ViralZone Ресурс Швейцарского института биоинформатики, предоставляющий информацию обо всех семействах вирусов, сопровождается общей молекулярной и эпидемиологической информацией. (англ.)
- Онлайн-семинар Дейвида Балтимора: «Введение в вирусы и ВИЧ». Дата обращения: 13 октября 2011. Архивировано из оригинала 13 октября 2011 года. (англ.)
- Онлайн-семинар Эри Гелениуса: «Введение в вирусы». Дата обращения: 14 октября 2011. Архивировано из оригинала 14 октября 2011 года. (англ.)
- 3D-структуры вирусов в EM Data Bank (EMDB). (англ.)
- Пташник, Ольга; Волкова, Ольга. Вирусы. Раскрась вирусные частицы. // Сайт Biomolecula.ru (17 февраля 2017). Дата обращения: 5 апреля 2018.
- Лапочкин, Юрий. Вирусные геномы в системе эволюции. // Сайт Biomolecula.ru (28 ноября 2014). Дата обращения: 5 апреля 2018.
- Ржешевский, Алексей. Вирусы и человек. Противостояние длиной в тысячелетия. // Сайт Biomolecula.ru (8 ноября 2015). Дата обращения: 5 апреля 2018.
- Чугунов, Антон. Нобелевскую премию 2008 года по физиологии и медицине вручили за вирусологические исследования. // Сайт Biomolecula.ru (6 ноября 2008). Дата обращения: 5 апреля 2018.
- Ожаровская, Татьяна. Фаговый домик. А в ваши строительные магазины уже завезли фаговые нанопровода? // Сайт Biomolecula.ru (11 ноября 2016). Дата обращения: 5 апреля 2018.
- Закубанский, Александр. Вирусы-платформы: яд во благо. // Сайт Biomolecula.ru (5 октября 2012). Дата обращения: 5 апреля 2018.
- Минина, Елизавета. Анти-CRISPR: ответ вирусов. // Сайт Biomolecula.ru (6 декабря 2017). Дата обращения: 5 апреля 2018.
- Цветные изображения вирусов из книги Карла Циммера Планета вирусов (2011) .
Уязвимости для Android
По статистике каждая пятая программа для ОС Android — с уязвимостью (или, иными словами, — с «дырой»), что позволяет злоумышленникам успешно внедрять мобильных троянцев на устройства и выполнять нужные им действия.
Аудитор безопасности в Dr.Web для Android произведет диагностику и анализ безопасности мобильного устройства, предложит решения для устранения выявленных проблем и уязвимостей.
Описание уязвимостей
Скачать Dr.Web для Android
For a more accessible and less technical introduction to this topic, see Introduction to viruses.
| Virus | |
|---|---|

|
|
| SARS-CoV-2, a member of the subfamily Coronavirinae | |
| Virus classification |
|
| (unranked): | Virus |
| Realms | |
|
A virus is a submicroscopic infectious agent that replicates only inside the living cells of an organism.[1] Viruses infect all life forms, from animals and plants to microorganisms, including bacteria and archaea.[2][3] Since Dmitri Ivanovsky’s 1892 article describing a non-bacterial pathogen infecting tobacco plants and the discovery of the tobacco mosaic virus by Martinus Beijerinck in 1898,[4] more than 11,000 of the millions of virus species have been described in detail.[5][6] Viruses are found in almost every ecosystem on Earth and are the most numerous type of biological entity.[7][8] The study of viruses is known as virology, a subspeciality of microbiology.
When infected, a host cell is often forced to rapidly produce thousands of copies of the original virus. When not inside an infected cell or in the process of infecting a cell, viruses exist in the form of independent viral particles, or virions, consisting of (i) the genetic material, i.e., long molecules of DNA or RNA that encode the structure of the proteins by which the virus acts; (ii) a protein coat, the capsid, which surrounds and protects the genetic material; and in some cases (iii) an outside envelope of lipids. The shapes of these virus particles range from simple helical and icosahedral forms to more complex structures. Most virus species have virions too small to be seen with an optical microscope and are one-hundredth the size of most bacteria.
The origins of viruses in the evolutionary history of life are unclear: some may have evolved from plasmids—pieces of DNA that can move between cells—while others may have evolved from bacteria. In evolution, viruses are an important means of horizontal gene transfer, which increases genetic diversity in a way analogous to sexual reproduction.[9] Viruses are considered by some biologists to be a life form, because they carry genetic material, reproduce, and evolve through natural selection, although they lack the key characteristics, such as cell structure, that are generally considered necessary criteria for defining life. Because they possess some but not all such qualities, viruses have been described as “organisms at the edge of life”[10] and as replicators.[11]
Viruses spread in many ways. One transmission pathway is through disease-bearing organisms known as vectors: for example, viruses are often transmitted from plant to plant by insects that feed on plant sap, such as aphids; and viruses in animals can be carried by blood-sucking insects. Many viruses, including influenza viruses, SARS-CoV-2, chickenpox, smallpox, and measles, spread in the air by coughing and sneezing. Norovirus and rotavirus, common causes of viral gastroenteritis, are transmitted by the faecal–oral route, passed by hand-to-mouth contact or in food or water. The infectious dose of norovirus required to produce infection in humans is fewer than 100 particles.[12] HIV is one of several viruses transmitted through sexual contact and by exposure to infected blood. The variety of host cells that a virus can infect is called its host range. This can be narrow, meaning a virus is capable of infecting few species, or broad, meaning it is capable of infecting many.[13]
Viral infections in animals provoke an immune response that usually eliminates the infecting virus. Immune responses can also be produced by vaccines, which confer an artificially acquired immunity to the specific viral infection. Some viruses, including those that cause HIV/AIDS, HPV infection, and viral hepatitis, evade these immune responses and result in chronic infections. Several classes of antiviral drugs have been developed.
Etymology
The word is from the Latin neuter vīrus referring to poison and other noxious liquids, from the same Indo-European base as Sanskrit viṣa, Avestan vīša, and ancient Greek ἰός (all meaning ‘poison’), first attested in English in 1398 in John Trevisa’s translation of Bartholomeus Anglicus’s De Proprietatibus Rerum.[14][15] Virulent, from Latin virulentus (‘poisonous’), dates to c. 1400.[16][17] A meaning of ‘agent that causes infectious disease’ is first recorded in 1728,[15] long before the discovery of viruses by Dmitri Ivanovsky in 1892. The English plural is viruses (sometimes also vira),[18] whereas the Latin word is a mass noun, which has no classically attested plural (vīra is used in Neo-Latin[19]). The adjective viral dates to 1948.[20] The term virion (plural virions), which dates from 1959,[21] is also used to refer to a single viral particle that is released from the cell and is capable of infecting other cells of the same type.[22]
Origins
Viruses are found wherever there is life and have probably existed since living cells first evolved.[23] The origin of viruses is unclear because they do not form fossils, so molecular techniques are used to investigate how they arose.[24] In addition, viral genetic material occasionally integrates into the germline of the host organisms, by which they can be passed on vertically to the offspring of the host for many generations. This provides an invaluable source of information for paleovirologists to trace back ancient viruses that have existed up to millions of years ago. There are three main hypotheses that aim to explain the origins of viruses:[25]
- Regressive hypothesis
- Viruses may have once been small cells that parasitised larger cells. Over time, genes not required by their parasitism were lost. The bacteria rickettsia and chlamydia are living cells that, like viruses, can reproduce only inside host cells. They lend support to this hypothesis, as their dependence on parasitism is likely to have caused the loss of genes that enabled them to survive outside a cell. This is also called the ‘degeneracy hypothesis’,[26][27] or ‘reduction hypothesis’.[28]
- Cellular origin hypothesis
- Some viruses may have evolved from bits of DNA or RNA that “escaped” from the genes of a larger organism. The escaped DNA could have come from plasmids (pieces of naked DNA that can move between cells) or transposons (molecules of DNA that replicate and move around to different positions within the genes of the cell).[29] Once called jumping genes, transposons are examples of mobile genetic elements and could be the origin of some viruses. They were discovered in maize by Barbara McClintock in 1950.[30] This is sometimes called the ‘vagrancy hypothesis’,[26][31] or the ‘escape hypothesis’.[28]
- Co-evolution hypothesis
- This is also called the ‘virus-first hypothesis’[28] and proposes that viruses may have evolved from complex molecules of protein and nucleic acid at the same time that cells first appeared on Earth and would have been dependent on cellular life for billions of years. Viroids are molecules of RNA that are not classified as viruses because they lack a protein coat. They have characteristics that are common to several viruses and are often called subviral agents.[32] Viroids are important pathogens of plants.[33] They do not code for proteins but interact with the host cell and use the host machinery for their replication.[34] The hepatitis delta virus of humans has an RNA genome similar to viroids but has a protein coat derived from hepatitis B virus and cannot produce one of its own. It is, therefore, a defective virus. Although hepatitis delta virus genome may replicate independently once inside a host cell, it requires the help of hepatitis B virus to provide a protein coat so that it can be transmitted to new cells.[35] In similar manner, the sputnik virophage is dependent on mimivirus, which infects the protozoan Acanthamoeba castellanii.[36] These viruses, which are dependent on the presence of other virus species in the host cell, are called ‘satellites’ and may represent evolutionary intermediates of viroids and viruses.[37][38]
In the past, there were problems with all of these hypotheses: the regressive hypothesis did not explain why even the smallest of cellular parasites do not resemble viruses in any way. The escape hypothesis did not explain the complex capsids and other structures on virus particles. The virus-first hypothesis contravened the definition of viruses in that they require host cells.[28] Viruses are now recognised as ancient and as having origins that pre-date the divergence of life into the three domains.[39] This discovery has led modern virologists to reconsider and re-evaluate these three classical hypotheses.[39]
The evidence for an ancestral world of RNA cells[40] and computer analysis of viral and host DNA sequences are giving a better understanding of the evolutionary relationships between different viruses and may help identify the ancestors of modern viruses. To date, such analyses have not proved which of these hypotheses is correct.[40] It seems unlikely that all currently known viruses have a common ancestor, and viruses have probably arisen numerous times in the past by one or more mechanisms.[41]
Microbiology
Life properties
Scientific opinions differ on whether viruses are a form of life or organic structures that interact with living organisms.[11] They have been described as “organisms at the edge of life”,[10] since they resemble organisms in that they possess genes, evolve by natural selection,[42] and reproduce by creating multiple copies of themselves through self-assembly. Although they have genes, they do not have a cellular structure, which is often seen as the basic unit of life. Viruses do not have their own metabolism and require a host cell to make new products. They therefore cannot naturally reproduce outside a host cell[43]—although some bacteria such as rickettsia and chlamydia are considered living organisms despite the same limitation.[44][45] Accepted forms of life use cell division to reproduce, whereas viruses spontaneously assemble within cells. They differ from autonomous growth of crystals as they inherit genetic mutations while being subject to natural selection. Virus self-assembly within host cells has implications for the study of the origin of life, as it lends further credence to the hypothesis that life could have started as self-assembling organic molecules.[2]
Structure
Virions of some of the most common human viruses with their relative size. The nucleic acids are not to scale.
Diagram of how a virus capsid can be constructed using multiple copies of just two protein molecules
Structure of icosahedral adenovirus. Electron micrograph with an illustration to show shape
Structure of chickenpox virus. They have a lipid envelope
Viruses display a wide diversity of sizes and shapes, called ‘morphologies’. In general, viruses are much smaller than bacteria and more than a thousand bacteriophage viruses would fit inside an Escherichia coli bacterium’s cell.[46] Many viruses that have been studied are spherical and have a diameter between 20 and 300 nanometres. Some filoviruses, which are filaments, have a total length of up to 1400 nm; their diameters are only about 80 nm.[47] Most viruses cannot be seen with an optical microscope, so scanning and transmission electron microscopes are used to visualise them.[48] To increase the contrast between viruses and the background, electron-dense “stains” are used. These are solutions of salts of heavy metals, such as tungsten, that scatter the electrons from regions covered with the stain. When virions are coated with stain (positive staining), fine detail is obscured. Negative staining overcomes this problem by staining the background only.[49]
A complete virus particle, known as a virion, consists of nucleic acid surrounded by a protective coat of protein called a capsid. These are formed from protein subunits called capsomeres.[50] Viruses can have a lipid “envelope” derived from the host cell membrane. The capsid is made from proteins encoded by the viral genome and its shape serves as the basis for morphological distinction.[51][52] Virally-coded protein subunits will self-assemble to form a capsid, in general requiring the presence of the virus genome. Complex viruses code for proteins that assist in the construction of their capsid. Proteins associated with nucleic acid are known as nucleoproteins, and the association of viral capsid proteins with viral nucleic acid is called a nucleocapsid. The capsid and entire virus structure can be mechanically (physically) probed through atomic force microscopy.[53][54] In general, there are five main morphological virus types:
- Helical
- These viruses are composed of a single type of capsomere stacked around a central axis to form a helical structure, which may have a central cavity, or tube. This arrangement results in virions which can be short and highly rigid rods, or long and very flexible filaments. The genetic material (typically single-stranded RNA, but single-stranded DNA in some cases) is bound into the protein helix by interactions between the negatively charged nucleic acid and positive charges on the protein. Overall, the length of a helical capsid is related to the length of the nucleic acid contained within it, and the diameter is dependent on the size and arrangement of capsomeres. The well-studied tobacco mosaic virus[55] and inovirus[56] are examples of helical viruses.
- Icosahedral
- Most animal viruses are icosahedral or near-spherical with chiral icosahedral symmetry. A regular icosahedron is the optimum way of forming a closed shell from identical subunits. The minimum number of capsomeres required for each triangular face is 3, which gives 60 for the icosahedron. Many viruses, such as rotavirus, have more than 60 capsomers and appear spherical but they retain this symmetry. To achieve this, the capsomeres at the apices are surrounded by five other capsomeres and are called pentons. Capsomeres on the triangular faces are surrounded by six others and are called hexons.[57] Hexons are in essence flat and pentons, which form the 12 vertices, are curved. The same protein may act as the subunit of both the pentamers and hexamers or they may be composed of different proteins.[58]
- Prolate
- This is an icosahedron elongated along the fivefold axis and is a common arrangement of the heads of bacteriophages. This structure is composed of a cylinder with a cap at either end.[59]
- Enveloped
- Some species of virus envelop themselves in a modified form of one of the cell membranes, either the outer membrane surrounding an infected host cell or internal membranes such as a nuclear membrane or endoplasmic reticulum, thus gaining an outer lipid bilayer known as a viral envelope. This membrane is studded with proteins coded for by the viral genome and host genome; the lipid membrane itself and any carbohydrates present originate entirely from the host. Influenza virus, HIV (which causes AIDS), and severe acute respiratory syndrome coronavirus 2 (which causes COVID-19)[60] use this strategy. Most enveloped viruses are dependent on the envelope for their infectivity.[61]
- Complex
- These viruses possess a capsid that is neither purely helical nor purely icosahedral, and that may possess extra structures such as protein tails or a complex outer wall. Some bacteriophages, such as Enterobacteria phage T4, have a complex structure consisting of an icosahedral head bound to a helical tail, which may have a hexagonal base plate with protruding protein tail fibres. This tail structure acts like a molecular syringe, attaching to the bacterial host and then injecting the viral genome into the cell.[62]
The poxviruses are large, complex viruses that have an unusual morphology. The viral genome is associated with proteins within a central disc structure known as a nucleoid. The nucleoid is surrounded by a membrane and two lateral bodies of unknown function. The virus has an outer envelope with a thick layer of protein studded over its surface. The whole virion is slightly pleomorphic, ranging from ovoid to brick-shaped.[63]
Giant viruses
Mimivirus is one of the largest characterised viruses, with a capsid diameter of 400 nm. Protein filaments measuring 100 nm project from the surface. The capsid appears hexagonal under an electron microscope, therefore the capsid is probably icosahedral.[64] In 2011, researchers discovered the largest then known virus in samples of water collected from the ocean floor off the coast of Las Cruces, Chile. Provisionally named Megavirus chilensis, it can be seen with a basic optical microscope.[65] In 2013, the Pandoravirus genus was discovered in Chile and Australia, and has genomes about twice as large as Megavirus and Mimivirus.[66] All giant viruses have dsDNA genomes and they are classified into several families: Mimiviridae, Pithoviridae, Pandoraviridae, Phycodnaviridae, and the Mollivirus genus.[67]
Some viruses that infect Archaea have complex structures unrelated to any other form of virus, with a wide variety of unusual shapes, ranging from spindle-shaped structures to viruses that resemble hooked rods, teardrops or even bottles. Other archaeal viruses resemble the tailed bacteriophages, and can have multiple tail structures.[68]
Genome
| Property | Parameters |
|---|---|
| Nucleic acid |
|
| Shape |
|
| Strandedness |
|
| Sense |
|
An enormous variety of genomic structures can be seen among viral species; as a group, they contain more structural genomic diversity than plants, animals, archaea, or bacteria. There are millions of different types of viruses,[6] although fewer than 7,000 types have been described in detail.[69] As of January 2021, the NCBI Virus genome database has more than 193,000 complete genome sequences,[70] but there are doubtlessly many more to be discovered.[71][72]
A virus has either a DNA or an RNA genome and is called a DNA virus or an RNA virus, respectively. The vast majority of viruses have RNA genomes. Plant viruses tend to have single-stranded RNA genomes and bacteriophages tend to have double-stranded DNA genomes.[73]
Viral genomes are circular, as in the polyomaviruses, or linear, as in the adenoviruses. The type of nucleic acid is irrelevant to the shape of the genome. Among RNA viruses and certain DNA viruses, the genome is often divided up into separate parts, in which case it is called segmented. For RNA viruses, each segment often codes for only one protein and they are usually found together in one capsid. All segments are not required to be in the same virion for the virus to be infectious, as demonstrated by brome mosaic virus and several other plant viruses.[47]
A viral genome, irrespective of nucleic acid type, is almost always either single-stranded (ss) or double-stranded (ds). Single-stranded genomes consist of an unpaired nucleic acid, analogous to one-half of a ladder split down the middle. Double-stranded genomes consist of two complementary paired nucleic acids, analogous to a ladder. The virus particles of some virus families, such as those belonging to the Hepadnaviridae, contain a genome that is partially double-stranded and partially single-stranded.[73]
For most viruses with RNA genomes and some with single-stranded DNA (ssDNA) genomes, the single strands are said to be either positive-sense (called the ‘plus-strand’) or negative-sense (called the ‘minus-strand’), depending on if they are complementary to the viral messenger RNA (mRNA). Positive-sense viral RNA is in the same sense as viral mRNA and thus at least a part of it can be immediately translated by the host cell. Negative-sense viral RNA is complementary to mRNA and thus must be converted to positive-sense RNA by an RNA-dependent RNA polymerase before translation. DNA nomenclature for viruses with genomic ssDNA is similar to RNA nomenclature, in that positive-strand viral ssDNA is identical in sequence to the viral mRNA and is thus a coding strand, while negative-sense viral ssDNA is complementary to the viral mRNA and is thus a template strand.[73] Several types of ssDNA and ssRNA viruses have genomes that are ambisense in that transcription can occur off both strands in a double-stranded replicative intermediate. Examples include geminiviruses, which are ssDNA plant viruses and arenaviruses, which are ssRNA viruses of animals.[74]
Genome size
Genome size varies greatly between species. The smallest—the ssDNA circoviruses, family Circoviridae—code for only two proteins and have a genome size of only two kilobases;[75] the largest—the pandoraviruses—have genome sizes of around two megabases which code for about 2500 proteins.[66] Virus genes rarely have introns and often are arranged in the genome so that they overlap.[76]
In general, RNA viruses have smaller genome sizes than DNA viruses because of a higher error-rate when replicating, and have a maximum upper size limit.[24] Beyond this, errors when replicating render the virus useless or uncompetitive. To compensate, RNA viruses often have segmented genomes—the genome is split into smaller molecules—thus reducing the chance that an error in a single-component genome will incapacitate the entire genome. In contrast, DNA viruses generally have larger genomes because of the high fidelity of their replication enzymes.[77] Single-strand DNA viruses are an exception to this rule, as mutation rates for these genomes can approach the extreme of the ssRNA virus case.[78]
Genetic mutation and recombination
Antigenic shift, or reassortment, can result in novel and highly pathogenic strains of human flu
Viruses undergo genetic change by several mechanisms. These include a process called antigenic drift where individual bases in the DNA or RNA mutate to other bases. Most of these point mutations are “silent”—they do not change the protein that the gene encodes—but others can confer evolutionary advantages such as resistance to antiviral drugs.[79][80] Antigenic shift occurs when there is a major change in the genome of the virus. This can be a result of recombination or reassortment. When this happens with influenza viruses, pandemics might result.[81] RNA viruses often exist as quasispecies or swarms of viruses of the same species but with slightly different genome nucleoside sequences. Such quasispecies are a prime target for natural selection.[82]
Segmented genomes confer evolutionary advantages; different strains of a virus with a segmented genome can shuffle and combine genes and produce progeny viruses (or offspring) that have unique characteristics. This is called reassortment or ‘viral sex’.[83]
Genetic recombination is a process by which a strand of DNA (or RNA) is broken and then joined to the end of a different DNA (or RNA) molecule. This can occur when viruses infect cells simultaneously and studies of viral evolution have shown that recombination has been rampant in the species studied.[84] Recombination is common to both RNA and DNA viruses.[85][86]
Coronaviruses have a single-strand positive-sense RNA genome. Replication of the genome is catalyzed by an RNA-dependent RNA polymerase. The mechanism of recombination used by coronaviruses likely involves template switching by the polymerase during genome replication.[87] This process appears to be an adaptation for coping with genome damage.[88]
Replication cycle
A typical virus replication cycle
Some bacteriophages inject their genomes into bacterial cells (not to scale)
Viral populations do not grow through cell division, because they are acellular. Instead, they use the machinery and metabolism of a host cell to produce multiple copies of themselves, and they assemble in the cell.[89] When infected, the host cell is forced to rapidly produce thousands of copies of the original virus.[90]
Their life cycle differs greatly between species, but there are six basic stages in their life cycle:[91]
Attachment is a specific binding between viral capsid proteins and specific receptors on the host cellular surface. This specificity determines the host range and type of host cell of a virus. For example, HIV infects a limited range of human leucocytes. This is because its surface protein, gp120, specifically interacts with the CD4 molecule—a chemokine receptor—which is most commonly found on the surface of CD4+ T-Cells. This mechanism has evolved to favour those viruses that infect only cells in which they are capable of replication. Attachment to the receptor can induce the viral envelope protein to undergo changes that result in the fusion of viral and cellular membranes, or changes of non-enveloped virus surface proteins that allow the virus to enter.[92]
Penetration or viral entry follows attachment: Virions enter the host cell through receptor-mediated endocytosis or membrane fusion. The infection of plant and fungal cells is different from that of animal cells. Plants have a rigid cell wall made of cellulose, and fungi one of chitin, so most viruses can get inside these cells only after trauma to the cell wall.[93] Nearly all plant viruses (such as tobacco mosaic virus) can also move directly from cell to cell, in the form of single-stranded nucleoprotein complexes, through pores called plasmodesmata.[94] Bacteria, like plants, have strong cell walls that a virus must breach to infect the cell. Given that bacterial cell walls are much thinner than plant cell walls due to their much smaller size, some viruses have evolved mechanisms that inject their genome into the bacterial cell across the cell wall, while the viral capsid remains outside.[95]
Uncoating is a process in which the viral capsid is removed: This may be by degradation by viral enzymes or host enzymes or by simple dissociation; the end-result is the releasing of the viral genomic nucleic acid.[96]
Replication of viruses involves primarily multiplication of the genome. Replication involves the synthesis of viral messenger RNA (mRNA) from “early” genes (with exceptions for positive-sense RNA viruses), viral protein synthesis, possible assembly of viral proteins, then viral genome replication mediated by early or regulatory protein expression. This may be followed, for complex viruses with larger genomes, by one or more further rounds of mRNA synthesis: “late” gene expression is, in general, of structural or virion proteins.[97]
Assembly – Following the structure-mediated self-assembly of the virus particles, some modification of the proteins often occurs. In viruses such as HIV, this modification (sometimes called maturation) occurs after the virus has been released from the host cell.[98]
Release – Viruses can be released from the host cell by lysis, a process that kills the cell by bursting its membrane and cell wall if present: this is a feature of many bacterial and some animal viruses. Some viruses undergo a lysogenic cycle where the viral genome is incorporated by genetic recombination into a specific place in the host’s chromosome. The viral genome is then known as a “provirus” or, in the case of bacteriophages a “prophage”.[99] Whenever the host divides, the viral genome is also replicated. The viral genome is mostly silent within the host. At some point, the provirus or prophage may give rise to the active virus, which may lyse the host cells.[100] Enveloped viruses (e.g., HIV) typically are released from the host cell by budding. During this process, the virus acquires its envelope, which is a modified piece of the host’s plasma or other, internal membrane.[101]
Genome replication
The genetic material within virus particles, and the method by which the material is replicated, varies considerably between different types of viruses.
- DNA viruses
- The genome replication of most DNA viruses takes place in the cell’s nucleus. If the cell has the appropriate receptor on its surface, these viruses enter the cell either by direct fusion with the cell membrane (e.g., herpesviruses) or—more usually—by receptor-mediated endocytosis. Most DNA viruses are entirely dependent on the host cell’s DNA and RNA synthesising machinery and RNA processing machinery. Viruses with larger genomes may encode much of this machinery themselves. In eukaryotes, the viral genome must cross the cell’s nuclear membrane to access this machinery, while in bacteria it need only enter the cell.[102]
- RNA viruses
- Replication of RNA viruses usually takes place in the cytoplasm. RNA viruses can be placed into four different groups depending on their modes of replication. The polarity (whether or not it can be used directly by ribosomes to make proteins) of single-stranded RNA viruses largely determines the replicative mechanism; the other major criterion is whether the genetic material is single-stranded or double-stranded. All RNA viruses use their own RNA replicase enzymes to create copies of their genomes.[103]
- Reverse transcribing viruses
- Reverse transcribing viruses have ssRNA (Retroviridae, Metaviridae, Pseudoviridae) or dsDNA (Caulimoviridae, and Hepadnaviridae) in their particles. Reverse transcribing viruses with RNA genomes (retroviruses) use a DNA intermediate to replicate, whereas those with DNA genomes (pararetroviruses) use an RNA intermediate during genome replication. Both types use a reverse transcriptase, or RNA-dependent DNA polymerase enzyme, to carry out the nucleic acid conversion. Retroviruses integrate the DNA produced by reverse transcription into the host genome as a provirus as a part of the replication process; pararetroviruses do not, although integrated genome copies of especially plant pararetroviruses can give rise to infectious virus.[104] They are susceptible to antiviral drugs that inhibit the reverse transcriptase enzyme, e.g. zidovudine and lamivudine. An example of the first type is HIV, which is a retrovirus. Examples of the second type are the Hepadnaviridae, which includes Hepatitis B virus.[105]
Cytopathic effects on the host cell
The range of structural and biochemical effects that viruses have on the host cell is extensive.[106] These are called ‘cytopathic effects’.[107] Most virus infections eventually result in the death of the host cell. The causes of death include cell lysis, alterations to the cell’s surface membrane and apoptosis.[108] Often cell death is caused by cessation of its normal activities because of suppression by virus-specific proteins, not all of which are components of the virus particle.[109] The distinction between cytopathic and harmless is gradual. Some viruses, such as Epstein–Barr virus, can cause cells to proliferate without causing malignancy,[110] while others, such as papillomaviruses, are established causes of cancer.[111]
Dormant and latent infections
Some viruses cause no apparent changes to the infected cell. Cells in which the virus is latent and inactive show few signs of infection and often function normally.[112] This causes persistent infections and the virus is often dormant for many months or years. This is often the case with herpes viruses.[113][114]
Host range
Viruses are by far the most abundant biological entities on Earth and they outnumber all the others put together.[115] They infect all types of cellular life including animals, plants, bacteria and fungi.[69] Different types of viruses can infect only a limited range of hosts and many are species-specific. Some, such as smallpox virus for example, can infect only one species—in this case humans,[116] and are said to have a narrow host range. Other viruses, such as rabies virus, can infect different species of mammals and are said to have a broad range.[117] The viruses that infect plants are harmless to animals, and most viruses that infect other animals are harmless to humans.[118] The host range of some bacteriophages is limited to a single strain of bacteria and they can be used to trace the source of outbreaks of infections by a method called phage typing.[119] The complete set of viruses in an organism or habitat is called the virome; for example, all human viruses constitute the human virome.[120]
Novel viruses
A novel virus is one that has not previously been recorded. It can be a virus that is isolated from its natural reservoir or isolated as the result of spread to an animal or human host where the virus had not been identified before. It can be an emergent virus, one that represents a new virus, but it can also be an extant virus that has not been previously identified.[121] The SARS-CoV-2 coronavirus that caused the pandemic of covid disease is an example of a novel virus.[122]
Classification
Classification seeks to describe the diversity of viruses by naming and grouping them on the basis of similarities. In 1962, André Lwoff, Robert Horne, and Paul Tournier were the first to develop a means of virus classification, based on the Linnaean hierarchical system.[123] This system based classification on phylum, class, order, family, genus, and species. Viruses were grouped according to their shared properties (not those of their hosts) and the type of nucleic acid forming their genomes.[124] In 1966, the International Committee on Taxonomy of Viruses (ICTV) was formed. The system proposed by Lwoff, Horne and Tournier was initially not accepted by the ICTV because the small genome size of viruses and their high rate of mutation made it difficult to determine their ancestry beyond order. As such, the Baltimore classification system has come to be used to supplement the more traditional hierarchy.[125] Starting in 2018, the ICTV began to acknowledge deeper evolutionary relationships between viruses that have been discovered over time and adopted a 15-rank classification system ranging from realm to species.[126] Additionally, some species within the same genus are grouped into a genogroup.[127][128]
ICTV classification
The ICTV developed the current classification system and wrote guidelines that put a greater weight on certain virus properties to maintain family uniformity. A unified taxonomy (a universal system for classifying viruses) has been established.[129] Only a small part of the total diversity of viruses has been studied.[130] As of 2022, 6 realms, 10 kingdoms, 17 phyla, 2 subphyla, 40 classes, 72 orders, 8 suborders, 264 families, 182 subfamilies, 2,818 genera, 84 subgenera, and 11,273 species of viruses have been defined by the ICTV.[5]
The general taxonomic structure of taxon ranges and the suffixes used in taxonomic names are shown hereafter. As of 2022, the ranks of subrealm, subkingdom, and subclass are unused, whereas all other ranks are in use.[5]
- Realm (-viria)
- Subrealm (-vira)
- Kingdom (-virae)
- Subkingdom (-virites)
- Phylum (-viricota)
- Subphylum (-viricotina)
- Class (-viricetes)
- Subclass (-viricetidae)
- Order (-virales)
- Suborder (-virineae)
- Family (-viridae)
- Subfamily (-virinae)
- Genus (-virus)
- Subgenus (-virus)
- Species
- Subgenus (-virus)
- Genus (-virus)
- Subfamily (-virinae)
- Family (-viridae)
- Suborder (-virineae)
- Order (-virales)
- Subclass (-viricetidae)
- Class (-viricetes)
- Subphylum (-viricotina)
- Phylum (-viricota)
- Subkingdom (-virites)
- Kingdom (-virae)
- Subrealm (-vira)
Baltimore classification
The Baltimore Classification of viruses is based on the method of viral mRNA synthesis
The Nobel Prize-winning biologist David Baltimore devised the Baltimore classification system.[131][132] The ICTV classification system is used in conjunction with the Baltimore classification system in modern virus classification.[133][134][135]
The Baltimore classification of viruses is based on the mechanism of mRNA production. Viruses must generate mRNAs from their genomes to produce proteins and replicate themselves, but different mechanisms are used to achieve this in each virus family. Viral genomes may be single-stranded (ss) or double-stranded (ds), RNA or DNA, and may or may not use reverse transcriptase (RT). In addition, ssRNA viruses may be either sense (+) or antisense (−). This classification places viruses into seven groups:
- I: dsDNA viruses (e.g. Adenoviruses, Herpesviruses, Poxviruses)
- II: ssDNA viruses (+ strand or “sense”) DNA (e.g. Parvoviruses)
- III: dsRNA viruses (e.g. Reoviruses)
- IV:(+)ssRNA viruses (+ strand or sense) RNA (e.g. Coronaviruses, Picornaviruses, Togaviruses)
- V: (−)ssRNA viruses (− strand or antisense) RNA (e.g. Orthomyxoviruses, Rhabdoviruses)
- VI: ssRNA-RT viruses (+ strand or sense) RNA with DNA intermediate in life-cycle (e.g. Retroviruses)
- VII: dsDNA-RT viruses DNA with RNA intermediate in life-cycle (e.g. Hepadnaviruses)
Role in human disease
Overview of the main types of viral infection and the most notable species involved[136]
Examples of common human diseases caused by viruses include the common cold, influenza, chickenpox, and cold sores. Many serious diseases such as rabies, Ebola virus disease, AIDS (HIV), avian influenza, and SARS are caused by viruses. The relative ability of viruses to cause disease is described in terms of virulence. Other diseases are under investigation to discover if they have a virus as the causative agent, such as the possible connection between human herpesvirus 6 (HHV6) and neurological diseases such as multiple sclerosis and chronic fatigue syndrome.[137] There is controversy over whether the bornavirus, previously thought to cause neurological diseases in horses, could be responsible for psychiatric illnesses in humans.[138]
Viruses have different mechanisms by which they produce disease in an organism, which depends largely on the viral species. Mechanisms at the cellular level primarily include cell lysis, the breaking open and subsequent death of the cell. In multicellular organisms, if enough cells die, the whole organism will start to suffer the effects. Although viruses cause disruption of healthy homeostasis, resulting in disease, they may exist relatively harmlessly within an organism. An example would include the ability of the herpes simplex virus, which causes cold sores, to remain in a dormant state within the human body. This is called latency[139] and is a characteristic of the herpes viruses, including Epstein–Barr virus, which causes glandular fever, and varicella zoster virus, which causes chickenpox and shingles. Most people have been infected with at least one of these types of herpes virus.[140] These latent viruses might sometimes be beneficial, as the presence of the virus can increase immunity against bacterial pathogens, such as Yersinia pestis.[141]
Some viruses can cause lifelong or chronic infections, where the viruses continue to replicate in the body despite the host’s defence mechanisms.[142] This is common in hepatitis B virus and hepatitis C virus infections. People chronically infected are known as carriers, as they serve as reservoirs of infectious virus.[143] In populations with a high proportion of carriers, the disease is said to be endemic.[144]
Epidemiology
Viral epidemiology is the branch of medical science that deals with the transmission and control of virus infections in humans. Transmission of viruses can be vertical, which means from mother to child, or horizontal, which means from person to person. Examples of vertical transmission include hepatitis B virus and HIV, where the baby is born already infected with the virus.[145] Another, more rare, example is the varicella zoster virus, which, although causing relatively mild infections in children and adults, can be fatal to the foetus and newborn baby.[146]
Horizontal transmission is the most common mechanism of spread of viruses in populations.[147] Horizontal transmission can occur when body fluids are exchanged during sexual activity, by exchange of saliva or when contaminated food or water is ingested. It can also occur when aerosols containing viruses are inhaled or by insect vectors such as when infected mosquitoes penetrate the skin of a host.[147] Most types of viruses are restricted to just one or two of these mechanisms and they are referred to as “respiratory viruses” or “enteric viruses” and so forth. The rate or speed of transmission of viral infections depends on factors that include population density, the number of susceptible individuals, (i.e., those not immune),[148] the quality of healthcare and the weather.[149]
Epidemiology is used to break the chain of infection in populations during outbreaks of viral diseases.[150] Control measures are used that are based on knowledge of how the virus is transmitted. It is important to find the source, or sources, of the outbreak and to identify the virus. Once the virus has been identified, the chain of transmission can sometimes be broken by vaccines. When vaccines are not available, sanitation and disinfection can be effective. Often, infected people are isolated from the rest of the community, and those that have been exposed to the virus are placed in quarantine.[151] To control the outbreak of foot-and-mouth disease in cattle in Britain in 2001, thousands of cattle were slaughtered.[152] Most viral infections of humans and other animals have incubation periods during which the infection causes no signs or symptoms.[153] Incubation periods for viral diseases range from a few days to weeks, but are known for most infections.[154] Somewhat overlapping, but mainly following the incubation period, there is a period of communicability—a time when an infected individual or animal is contagious and can infect another person or animal.[154] This, too, is known for many viral infections, and knowledge of the length of both periods is important in the control of outbreaks.[155] When outbreaks cause an unusually high proportion of cases in a population, community, or region, they are called epidemics. If outbreaks spread worldwide, they are called pandemics.[156]
Epidemics and pandemics
A pandemic is a worldwide epidemic. The 1918 flu pandemic, which lasted until 1919, was a category 5 influenza pandemic caused by an unusually severe and deadly influenza A virus. The victims were often healthy young adults, in contrast to most influenza outbreaks, which predominantly affect juvenile, elderly, or otherwise-weakened patients.[157] Older estimates say it killed 40–50 million people,[158] while more recent research suggests that it may have killed as many as 100 million people, or 5% of the world’s population in 1918.[159]
Although viral pandemics are rare events, HIV—which evolved from viruses found in monkeys and chimpanzees—has been pandemic since at least the 1980s.[160] During the 20th century there were four pandemics caused by influenza virus and those that occurred in 1918, 1957 and 1968 were severe.[161] Most researchers believe that HIV originated in sub-Saharan Africa during the 20th century;[162] it is now a pandemic, with an estimated 37.9 million people now living with the disease worldwide.[163] There were about 770,000 deaths from AIDS in 2018.[164] The Joint United Nations Programme on HIV/AIDS (UNAIDS) and the World Health Organization (WHO) estimate that AIDS has killed more than 25 million people since it was first recognised on 5 June 1981, making it one of the most destructive epidemics in recorded history.[165] In 2007 there were 2.7 million new HIV infections and 2 million HIV-related deaths.[166]
Ebola (top) and Marburg viruses (bottom)
Several highly lethal viral pathogens are members of the Filoviridae. Filoviruses are filament-like viruses that cause viral hemorrhagic fever, and include ebolaviruses and marburgviruses. Marburg virus, first discovered in 1967, attracted widespread press attention in April 2005 for an outbreak in Angola.[167] Ebola virus disease has also caused intermittent outbreaks with high mortality rates since 1976 when it was first identified. The worst and most recent one is the 2013–2016 West Africa epidemic.[168]
Except for smallpox, most pandemics are caused by newly evolved viruses. These “emergent” viruses are usually mutants of less harmful viruses that have circulated previously either in humans or other animals.[169]
Severe acute respiratory syndrome (SARS) and Middle East respiratory syndrome (MERS) are caused by new types of coronaviruses. Other coronaviruses are known to cause mild infections in humans,[170] so the virulence and rapid spread of SARS infections—that by July 2003 had caused around 8,000 cases and 800 deaths—was unexpected and most countries were not prepared.[171]
A related coronavirus, severe acute respiratory syndrome coronavirus 2 (SARS-Cov-2), thought to have originated in bats, emerged in Wuhan, China in November 2019 and spread rapidly around the world. Infections with the virus caused the COVID-19 pandemic that started in 2020.[122][172][173] Unprecedented restrictions in peacetime were placed on international travel,[174] and curfews were imposed in several major cities worldwide in response to the pandemic.[175]
Cancer
Viruses are an established cause of cancer in humans and other species. Viral cancers occur only in a minority of infected persons (or animals). Cancer viruses come from a range of virus families, including both RNA and DNA viruses, and so there is no single type of “oncovirus” (an obsolete term originally used for acutely transforming retroviruses). The development of cancer is determined by a variety of factors such as host immunity[176] and mutations in the host.[177] Viruses accepted to cause human cancers include some genotypes of human papillomavirus, hepatitis B virus, hepatitis C virus, Epstein–Barr virus, Kaposi’s sarcoma-associated herpesvirus and human T-lymphotropic virus. The most recently discovered human cancer virus is a polyomavirus (Merkel cell polyomavirus) that causes most cases of a rare form of skin cancer called Merkel cell carcinoma.[178]
Hepatitis viruses can develop into a chronic viral infection that leads to liver cancer.[179][180] Infection by human T-lymphotropic virus can lead to tropical spastic paraparesis and adult T-cell leukaemia.[181] Human papillomaviruses are an established cause of cancers of cervix, skin, anus, and penis.[182] Within the Herpesviridae, Kaposi’s sarcoma-associated herpesvirus causes Kaposi’s sarcoma and body-cavity lymphoma, and Epstein–Barr virus causes Burkitt’s lymphoma, Hodgkin’s lymphoma, B lymphoproliferative disorder, and nasopharyngeal carcinoma.[183] Merkel cell polyomavirus closely related to SV40 and mouse polyomaviruses that have been used as animal models for cancer viruses for over 50 years.[184]
Host defence mechanisms
The body’s first line of defence against viruses is the innate immune system. This comprises cells and other mechanisms that defend the host from infection in a non-specific manner. This means that the cells of the innate system recognise, and respond to, pathogens in a generic way, but, unlike the adaptive immune system, it does not confer long-lasting or protective immunity to the host.[185]
RNA interference is an important innate defence against viruses.[186] Many viruses have a replication strategy that involves double-stranded RNA (dsRNA). When such a virus infects a cell, it releases its RNA molecule or molecules, which immediately bind to a protein complex called a dicer that cuts the RNA into smaller pieces. A biochemical pathway—the RISC complex—is activated, which ensures cell survival by degrading the viral mRNA. Rotaviruses have evolved to avoid this defence mechanism by not uncoating fully inside the cell, and releasing newly produced mRNA through pores in the particle’s inner capsid. Their genomic dsRNA remains protected inside the core of the virion.[187][188]
When the adaptive immune system of a vertebrate encounters a virus, it produces specific antibodies that bind to the virus and often render it non-infectious. This is called humoral immunity. Two types of antibodies are important. The first, called IgM, is highly effective at neutralising viruses but is produced by the cells of the immune system only for a few weeks. The second, called IgG, is produced indefinitely. The presence of IgM in the blood of the host is used to test for acute infection, whereas IgG indicates an infection sometime in the past.[189] IgG antibody is measured when tests for immunity are carried out.[190]
Antibodies can continue to be an effective defence mechanism even after viruses have managed to gain entry to the host cell. A protein that is in cells, called TRIM21, can attach to the antibodies on the surface of the virus particle. This primes the subsequent destruction of the virus by the enzymes of the cell’s proteosome system.[191]
Two rotaviruses: the one on the right is coated with antibodies that prevent its attachment to cells and infecting them.
A second defence of vertebrates against viruses is called cell-mediated immunity and involves immune cells known as T cells. The body’s cells constantly display short fragments of their proteins on the cell’s surface, and, if a T cell recognises a suspicious viral fragment there, the host cell is destroyed by ‘killer T’ cells and the virus-specific T-cells proliferate. Cells such as the macrophage are specialists at this antigen presentation.[192] The production of interferon is an important host defence mechanism. This is a hormone produced by the body when viruses are present. Its role in immunity is complex; it eventually stops the viruses from reproducing by killing the infected cell and its close neighbours.[193]
Not all virus infections produce a protective immune response in this way. HIV evades the immune system by constantly changing the amino acid sequence of the proteins on the surface of the virion. This is known as “escape mutation” as the viral epitopes escape recognition by the host immune response. These persistent viruses evade immune control by sequestration, blockade of antigen presentation, cytokine resistance, evasion of natural killer cell activities, escape from apoptosis, and antigenic shift.[194] Other viruses, called ‘neurotropic viruses’, are disseminated by neural spread where the immune system may be unable to reach them due to immune privilege.[195]
Prevention and treatment
Because viruses use vital metabolic pathways within host cells to replicate, they are difficult to eliminate without using drugs that cause toxic effects to host cells in general. The most effective medical approaches to viral diseases are vaccinations to provide immunity to infection, and antiviral drugs that selectively interfere with viral replication.
Vaccines
Vaccination is a cheap and effective way of preventing infections by viruses. Vaccines were used to prevent viral infections long before the discovery of the actual viruses. Their use has resulted in a dramatic decline in morbidity (illness) and mortality (death) associated with viral infections such as polio, measles, mumps and rubella.[196] Smallpox infections have been eradicated.[197] Vaccines are available to prevent over thirteen viral infections of humans,[198] and more are used to prevent viral infections of animals.[199] Vaccines can consist of live-attenuated or killed viruses, viral proteins (antigens), or RNA.[200][201] Live vaccines contain weakened forms of the virus, which do not cause the disease but, nonetheless, confer immunity. Such viruses are called attenuated. Live vaccines can be dangerous when given to people with a weak immunity (who are described as immunocompromised), because in these people, the weakened virus can cause the original disease.[202] Biotechnology and genetic engineering techniques are used to produce subunit vaccines. These vaccines use only the capsid proteins of the virus. Hepatitis B vaccine is an example of this type of vaccine.[203] Subunit vaccines are safe for immunocompromised patients because they cannot cause the disease.[204] The yellow fever virus vaccine, a live-attenuated strain called 17D, is probably the safest and most effective vaccine ever generated.[205]
Antiviral drugs
Antiviral drugs are often nucleoside analogues (fake DNA building-blocks), which viruses mistakenly incorporate into their genomes during replication.[206] The life-cycle of the virus is then halted because the newly synthesised DNA is inactive. This is because these analogues lack the hydroxyl groups, which, along with phosphorus atoms, link together to form the strong “backbone” of the DNA molecule. This is called DNA chain termination.[207] Examples of nucleoside analogues are aciclovir for Herpes simplex virus infections and lamivudine for HIV and hepatitis B virus infections. Aciclovir is one of the oldest and most frequently prescribed antiviral drugs.[208]
Other antiviral drugs in use target different stages of the viral life cycle. HIV is dependent on a proteolytic enzyme called the HIV-1 protease for it to become fully infectious. There is a large class of drugs called protease inhibitors that inactivate this enzyme.[209] There are around thirteen classes of antiviral drugs each targeting different viruses or stages of viral replication.[206]
Hepatitis C is caused by an RNA virus. In 80% of people infected, the disease is chronic, and without treatment, they are infected for the remainder of their lives. There are effective treatments that use direct-acting antivirals.[210] The treatment of chronic carriers of the hepatitis B virus has also been developed by using similar strategies that include lamivudine and other anti-viral drugs.[211]
Infection in other species
Viruses infect all cellular life and, although viruses occur universally, each cellular species has its own specific range that often infects only that species.[212] Some viruses, called satellites, can replicate only within cells that have already been infected by another virus.[36]
Animal viruses
Viruses are important pathogens of livestock. Diseases such as foot-and-mouth disease and bluetongue are caused by viruses.[213] Companion animals such as cats, dogs, and horses, if not vaccinated, are susceptible to serious viral infections. Canine parvovirus is caused by a small DNA virus and infections are often fatal in pups.[214] Like all invertebrates, the honey bee is susceptible to many viral infections.[215] Most viruses co-exist harmlessly in their host and cause no signs or symptoms of disease.[4]
Plant viruses
Peppers infected by mild mottle virus
There are many types of plant viruses, but often they cause only a loss of yield, and it is not economically viable to try to control them. Plant viruses are often spread from plant to plant by organisms, known as vectors. These are usually insects, but some fungi, nematode worms, single-celled organisms, and parasitic plants are vectors.[216] When control of plant virus infections is considered economical, for perennial fruits, for example, efforts are concentrated on killing the vectors and removing alternate hosts such as weeds.[217] Plant viruses cannot infect humans and other animals because they can reproduce only in living plant cells.[218]
Originally from Peru, the potato has become a staple crop worldwide.[219] The potato virus Y causes disease in potatoes and related species including tomatoes and peppers. In the 1980s, this virus acquired economical importance when it proved difficult to control in seed potato crops. Transmitted by aphids, this virus can reduce crop yields by up to 80 per cent, causing significant losses to potato yields.[220]
Plants have elaborate and effective defence mechanisms against viruses. One of the most effective is the presence of so-called resistance (R) genes. Each R gene confers resistance to a particular virus by triggering localised areas of cell death around the infected cell, which can often be seen with the unaided eye as large spots. This stops the infection from spreading.[221] RNA interference is also an effective defence in plants.[222] When they are infected, plants often produce natural disinfectants that kill viruses, such as salicylic acid, nitric oxide, and reactive oxygen molecules.[223]
Plant virus particles or virus-like particles (VLPs) have applications in both biotechnology and nanotechnology. The capsids of most plant viruses are simple and robust structures and can be produced in large quantities either by the infection of plants or by expression in a variety of heterologous systems. Plant virus particles can be modified genetically and chemically to encapsulate foreign material and can be incorporated into supramolecular structures for use in biotechnology.[224]
Bacterial viruses
Transmission electron micrograph of multiple bacteriophages attached to a bacterial cell wall
Bacteriophages are a common and diverse group of viruses and are the most abundant biological entity in aquatic environments—there are up to ten times more of these viruses in the oceans than there are bacteria,[225] reaching levels of 250,000,000 bacteriophages per millilitre of seawater.[226] These viruses infect specific bacteria by binding to surface receptor molecules and then entering the cell. Within a short amount of time, in some cases, just minutes, bacterial polymerase starts translating viral mRNA into protein. These proteins go on to become either new virions within the cell, helper proteins, which help assembly of new virions, or proteins involved in cell lysis. Viral enzymes aid in the breakdown of the cell membrane, and, in the case of the T4 phage, in just over twenty minutes after injection over three hundred phages could be released.[227]
The major way bacteria defend themselves from bacteriophages is by producing enzymes that destroy foreign DNA. These enzymes, called restriction endonucleases, cut up the viral DNA that bacteriophages inject into bacterial cells.[228] Bacteria also contain a system that uses CRISPR sequences to retain fragments of the genomes of viruses that the bacteria have come into contact with in the past, which allows them to block the virus’s replication through a form of RNA interference.[229][230] This genetic system provides bacteria with acquired immunity to infection.[231]
Archaeal viruses
Some viruses replicate within archaea: these are DNA viruses with unusual and sometimes unique shapes.[7][68] These viruses have been studied in most detail in the thermophilic archaea, particularly the orders Sulfolobales and Thermoproteales.[232] Defences against these viruses involve RNA interference from repetitive DNA sequences within archaean genomes that are related to the genes of the viruses.[233][234] Most archaea have CRISPR–Cas systems as an adaptive defence against viruses. These enable archaea to retain sections of viral DNA, which are then used to target and eliminate subsequent infections by the virus using a process similar to RNA interference.[235]
Role in aquatic ecosystems
Viruses are the most abundant biological entity in aquatic environments.[2] There are about ten million of them in a teaspoon of seawater.[236] Most of these viruses are bacteriophages infecting heterotrophic bacteria and cyanophages infecting cyanobacteria and they are essential to the regulation of saltwater and freshwater ecosystems.[237]
Bacteriophages are harmless to plants and animals, and are essential to the regulation of marine and freshwater ecosystems[238] are important mortality agents of phytoplankton, the base of the foodchain in aquatic environments.[239] They infect and destroy bacteria in aquatic microbial communities, and are one of the most important mechanisms of recycling carbon and nutrient cycling in marine environments. The organic molecules released from the dead bacterial cells stimulate fresh bacterial and algal growth, in a process known as the viral shunt.[240] In particular, lysis of bacteria by viruses has been shown to enhance nitrogen cycling and stimulate phytoplankton growth.[241] Viral activity may also affect the biological pump, the process whereby carbon is sequestered in the deep ocean.[242]
Microorganisms constitute more than 90% of the biomass in the sea. It is estimated that viruses kill approximately 20% of this biomass each day and that there are 10 to 15 times as many viruses in the oceans as there are bacteria and archaea.[243] Viruses are also major agents responsible for the destruction of phytoplankton including harmful algal blooms,[244]
The number of viruses in the oceans decreases further offshore and deeper into the water, where there are fewer host organisms.[242]
In January 2018, scientists reported that 800 million viruses, mainly of marine origin, are deposited daily from the Earth‘s atmosphere onto every square meter of the planet’s surface, as the result of a global atmospheric stream of viruses, circulating above the weather system but below the altitude of usual airline travel, distributing viruses around the planet.[245][246]
Like any organism, marine mammals are susceptible to viral infections. In 1988 and 2002, thousands of harbour seals were killed in Europe by phocine distemper virus.[247] Many other viruses, including caliciviruses, herpesviruses, adenoviruses and parvoviruses, circulate in marine mammal populations.[242]
In December 2022, scientists reported the first observation of virovory via an experiment on pond water containing chlorovirus, which commonly infects green algae in freshwater environments. When all other microbial food sources were removed from the water, the ciliate Halteria was observed to have increased in number due to the active consumption of chlorovirus as a food source instead of its typical bacterivore diet.[248][249]
Role in evolution
Viruses are an important natural means of transferring genes between different species, which increases genetic diversity and drives evolution.[9][250] It is thought that viruses played a central role in early evolution, before the diversification of the last universal common ancestor into bacteria, archaea and eukaryotes.[251] Viruses are still one of the largest reservoirs of unexplored genetic diversity on Earth.[242]
Applications
Life sciences and medicine
Scientist studying the H5N1 influenza virus
Viruses are important to the study of molecular and cell biology as they provide simple systems that can be used to manipulate and investigate the functions of cells.[252] The study and use of viruses have provided valuable information about aspects of cell biology.[253] For example, viruses have been useful in the study of genetics and helped our understanding of the basic mechanisms of molecular genetics, such as DNA replication, transcription, RNA processing, translation, protein transport, and immunology.
Geneticists often use viruses as vectors to introduce genes into cells that they are studying. This is useful for making the cell produce a foreign substance, or to study the effect of introducing a new gene into the genome. Similarly, virotherapy uses viruses as vectors to treat various diseases, as they can specifically target cells and DNA. It shows promising use in the treatment of cancer and in gene therapy. Eastern European scientists have used phage therapy as an alternative to antibiotics for some time, and interest in this approach is increasing, because of the high level of antibiotic resistance now found in some pathogenic bacteria.[254]
The expression of heterologous proteins by viruses is the basis of several manufacturing processes that are currently being used for the production of various proteins such as vaccine antigens and antibodies. Industrial processes have been recently developed using viral vectors and several pharmaceutical proteins are currently in pre-clinical and clinical trials.[255]
Virotherapy
Virotherapy involves the use of genetically modified viruses to treat diseases.[256] Viruses have been modified by scientists to reproduce in cancer cells and destroy them but not infect healthy cells. Talimogene laherparepvec (T-VEC), for example, is a modified herpes simplex virus that has had a gene, which is required for viruses to replicate in healthy cells, deleted and replaced with a human gene (GM-CSF) that stimulates immunity. When this virus infects cancer cells, it destroys them and in doing so the presence the GM-CSF gene attracts dendritic cells from the surrounding tissues of the body. The dendritic cells process the dead cancer cells and present components of them to other cells of the immune system.[257] Having completed successful clinical trials, the virus gained approval for the treatment of melanoma in late 2015.[258] Viruses that have been reprogrammed to kill cancer cells are called oncolytic viruses.[259]
Materials science and nanotechnology
Current trends in nanotechnology promise to make much more versatile use of viruses.[260] From the viewpoint of a materials scientist, viruses can be regarded as organic nanoparticles.
Their surface carries specific tools that enable them to cross the barriers of their host cells. The size and shape of viruses and the number and nature of the functional groups on their surface are precisely defined. As such, viruses are commonly used in materials science as scaffolds for covalently linked surface modifications. A particular quality of viruses is that they can be tailored by directed evolution. The powerful techniques developed by life sciences are becoming the basis of engineering approaches towards nanomaterials, opening a wide range of applications far beyond biology and medicine.[261]
Because of their size, shape, and well-defined chemical structures, viruses have been used as templates for organising materials on the nanoscale. Recent examples include work at the Naval Research Laboratory in Washington, D.C., using Cowpea mosaic virus (CPMV) particles to amplify signals in DNA microarray based sensors. In this application, the virus particles separate the fluorescent dyes used for signalling to prevent the formation of non-fluorescent dimers that act as quenchers.[262] Another example is the use of CPMV as a nanoscale breadboard for molecular electronics.[263]
Synthetic viruses
Many viruses can be synthesised de novo (“from scratch”). The first synthetic virus was created in 2002.[264] Although somewhat of a misconception, it is not the actual virus that is synthesised, but rather its DNA genome (in case of a DNA virus), or a cDNA copy of its genome (in case of RNA viruses). For many virus families the naked synthetic DNA or RNA (once enzymatically converted back from the synthetic cDNA) is infectious when introduced into a cell. That is, they contain all the necessary information to produce new viruses. This technology is now being used to investigate novel vaccine strategies.[265] The ability to synthesise viruses has far-reaching consequences, since viruses can no longer be regarded as extinct, as long as the information of their genome sequence is known and permissive cells are available. As of June 2021, the full-length genome sequences of 11,464 different viruses, including smallpox, are publicly available in an online database maintained by the National Institutes of Health.[266]
Weapons
The ability of viruses to cause devastating epidemics in human societies has led to the concern that viruses could be weaponised for biological warfare. Further concern was raised by the successful recreation of the infamous 1918 influenza virus in a laboratory.[267]
The smallpox virus devastated numerous societies throughout history before its eradication. There are only two centres in the world authorised by the WHO to keep stocks of smallpox virus: the State Research Center of Virology and Biotechnology VECTOR in Russia and the Centers for Disease Control and Prevention in the United States.[268] It may be used as a weapon,[268] as the vaccine for smallpox sometimes had severe side-effects, it is no longer used routinely in any country. Thus, much of the modern human population has almost no established resistance to smallpox and would be vulnerable to the virus.[268]
See also
- Cross-species transmission
- Glossary of virology
- Law of declining virulence – Disproved hypothesis of epidemiologist Theobald Smith
- Non-cellular life
- Retrozyme
- Smallest organisms
- Theory of virulence – Theory by biologist Paul W. Ewald
- Viral metagenomics
- Viroplasm
- Zoonosis
References
Notes
- ^ Wu KJ (15 April 2020). “There are more viruses than stars in the universe. Why do only some infect us? – More than a quadrillion quadrillion individual viruses exist on Earth, but most are not poised to hop into humans. Can we find the ones that are?”. National Geographic Society. Retrieved 18 May 2020.
- ^ a b c Koonin EV, Senkevich TG, Dolja VV (September 2006). “The ancient Virus World and evolution of cells”. Biology Direct. 1 (1): 29. doi:10.1186/1745-6150-1-29. PMC 1594570. PMID 16984643.
- ^ Zimmer C (26 February 2021). “The Secret Life of a Coronavirus – An oily, 100-nanometer-wide bubble of genes has killed more than two million people and reshaped the world. Scientists don’t quite know what to make of it”. The New York Times. Archived from the original on 28 December 2021. Retrieved 28 February 2021.
- ^ a b Dimmock p. 4
- ^ a b c “Virus Taxonomy: 2022 Release”. talk.ictvonline.org. International Committee on Taxonomy of Viruses. Retrieved 27 April 2023.
- ^ a b Breitbart M, Rohwer F (June 2005). “Here a virus, there a virus, everywhere the same virus?”. Trends in Microbiology. 13 (6): 278–84. doi:10.1016/j.tim.2005.04.003. PMID 15936660.
- ^ a b Lawrence CM, Menon S, Eilers BJ, Bothner B, Khayat R, Douglas T, Young MJ (May 2009). “Structural and functional studies of archaeal viruses”. The Journal of Biological Chemistry. 284 (19): 12599–603. doi:10.1074/jbc.R800078200. PMC 2675988. PMID 19158076.
- ^ Edwards RA, Rohwer F (June 2005). “Viral metagenomics”. Nature Reviews. Microbiology. 3 (6): 504–10. doi:10.1038/nrmicro1163. PMID 15886693. S2CID 8059643.
- ^ a b Canchaya C, Fournous G, Chibani-Chennoufi S, Dillmann ML, Brüssow H (August 2003). “Phage as agents of lateral gene transfer”. Current Opinion in Microbiology. 6 (4): 417–24. doi:10.1016/S1369-5274(03)00086-9. PMID 12941415.
- ^ a b Rybicki EP (1990). “The classification of organisms at the edge of life, or problems with virus systematics”. South African Journal of Science. 86: 182–86.
- ^ a b Koonin EV, Starokadomskyy P (October 2016). “Are viruses alive? The replicator paradigm sheds decisive light on an old but misguided question”. Studies in History and Philosophy of Biological and Biomedical Sciences. 59: 125–34. doi:10.1016/j.shpsc.2016.02.016. PMC 5406846. PMID 26965225.
- ^ Robilotti E, Deresinski S, Pinsky BA (January 2015). “Norovirus”. Clinical Microbiology Reviews. 28 (1): 134–64. doi:10.1128/CMR.00075-14. PMC 4284304. PMID 25567225.
- ^ Shors pp. 123–124
- ^ “Virus, n.”. OED Online. Oxford University Press. March 2015.
- ^ a b Harper D (2011). “virus”. The Online Etymology Dictionary. Retrieved 19 December 2014.
- ^ “Virulent, adj.”. OED Online. Oxford University Press. March 2015.
- ^ Harper D (2011). “virulent”. The Online Etymology Dictionary. Retrieved 19 December 2014.
- ^ Buschard K, Thon R (2003). “Diabetic Animal Models”. In Hau J, Van Hoosier Jr GL (eds.). Handbook of Laboratory Animal Science. Animal Models. Vol. II (Second ed.). CRC Press. pp. 163, 166.
- ^ William T. Stearn: Botanical Latin. History, Grammar, Syntax, Terminology and Vocabulary. David & Charles, third edition, 1983. Quote: “Virus: virus (s.n. II), gen. sing. viri, nom. pl. vira, gen. pl. vīrorum (to be distinguished from virorum, of men).”
- ^ Harper D (2011). “viral”. The Online Etymology Dictionary. Retrieved 19 December 2014.
- ^ Harper D (2011). “virion”. The Online Etymology Dictionary. Retrieved 19 December 2014.
- ^ Casjens S (2010). Mahy BW, Van Regenmortel MH (eds.). Desk Encyclopedia of General Virology. Boston: Academic Press. p. 167. ISBN 978-0-12-375146-1.
- ^ Iyer LM, Balaji S, Koonin EV, Aravind L (April 2006). “Evolutionary genomics of nucleo-cytoplasmic large DNA viruses”. Virus Research. 117 (1): 156–84. doi:10.1016/j.virusres.2006.01.009. PMID 16494962.
- ^ a b Sanjuán R, Nebot MR, Chirico N, Mansky LM, Belshaw R (October 2010). “Viral mutation rates”. Journal of Virology. 84 (19): 9733–48. doi:10.1128/JVI.00694-10. PMC 2937809. PMID 20660197.
- ^ Krupovic M, Dolja VV, Koonin EV (July 2019). “Origin of viruses: primordial replicators recruiting capsids from hosts” (PDF). Nature Reviews. Microbiology. 17 (7): 449–458. doi:10.1038/s41579-019-0205-6. PMID 31142823. S2CID 256744818.
- ^ a b Dimmock p. 16
- ^ Collier p. 11
- ^ a b c d Mahy WJ, Regenmortel MH, eds. (2009). Desk Encyclopedia of General Virology. Oxford: Academic Press. p. 24. ISBN 978-0-12-375146-1.
- ^ Shors p. 810
- ^ McClintock B (June 1950). “The origin and behavior of mutable loci in maize”. Proceedings of the National Academy of Sciences of the United States of America. 36 (6): 344–55. Bibcode:1950PNAS…36..344M. doi:10.1073/pnas.36.6.344. PMC 1063197. PMID 15430309.
- ^ Collier pp. 11–12
- ^ Dimmock p. 55
- ^ Shors pp. 791
- ^ Tsagris EM, Martínez de Alba AE, Gozmanova M, Kalantidis K (November 2008). “Viroids”. Cellular Microbiology. 10 (11): 2168–79. doi:10.1111/j.1462-5822.2008.01231.x. PMID 18764915. S2CID 221581424.
- ^ Shors pp. 460
- ^ a b La Scola B, Desnues C, Pagnier I, Robert C, Barrassi L, Fournous G, et al. (September 2008). “The virophage as a unique parasite of the giant mimivirus”. Nature. 455 (7209): 100–04. Bibcode:2008Natur.455..100L. doi:10.1038/nature07218. PMID 18690211. S2CID 4422249.
- ^ Collier p. 777
- ^ Dimmock pp. 55–57
- ^ a b Mahy WJ, Van Regenmortel MH, eds. (2009). Desk Encyclopedia of General Virology. Oxford: Academic Press. p. 28. ISBN 978-0-12-375146-1.
- ^ a b Mahy WJ, Regenmortel MH, eds. (2009). Desk Encyclopedia of General Virology. Oxford: Academic Press. p. 26. ISBN 978-0-12-375146-1.
- ^ Dimmock pp. 15–16
- ^ Holmes EC (October 2007). “Viral evolution in the genomic age”. PLOS Biology. 5 (10): e278. doi:10.1371/journal.pbio.0050278. PMC 1994994. PMID 17914905.
- ^ Wimmer E, Mueller S, Tumpey TM, Taubenberger JK (December 2009). “Synthetic viruses: a new opportunity to understand and prevent viral disease”. Nature Biotechnology. 27 (12): 1163–72. doi:10.1038/nbt.1593. PMC 2819212. PMID 20010599.
- ^ Horn M (2008). “Chlamydiae as symbionts in eukaryotes”. Annual Review of Microbiology. 62: 113–31. doi:10.1146/annurev.micro.62.081307.162818. PMID 18473699.
- ^ Ammerman NC, Beier-Sexton M, Azad AF (November 2008). “Laboratory maintenance of Rickettsia rickettsii”. Current Protocols in Microbiology. 11 (1): 3A.5.1–3A.5.21. doi:10.1002/9780471729259.mc03a05s11. ISBN 978-0471729259. PMC 2725428. PMID 19016440.
- ^ Krasner p. 98
- ^ a b Collier pp. 33–55
- ^ Collier pp. 33–37
- ^ Kiselev NA, Sherman MB, Tsuprun VL (1990). “Negative staining of proteins”. Electron Microscopy Reviews. 3 (1): 43–72. doi:10.1016/0892-0354(90)90013-I. PMID 1715774.
- ^ Collier p. 40
- ^ Caspar DL, Klug A (1962). “Physical principles in the construction of regular viruses”. Cold Spring Harbor Symposia on Quantitative Biology. 27: 1–24. doi:10.1101/sqb.1962.027.001.005. PMID 14019094.
- ^ Crick FH, Watson JD (March 1956). “Structure of small viruses”. Nature. 177 (4506): 473–75. Bibcode:1956Natur.177..473C. doi:10.1038/177473a0. PMID 13309339. S2CID 5740221.
- ^ Falvo MR, Washburn S, Superfine R, Finch M, Brooks FP, Chi V, Taylor RM (March 1997). “Manipulation of individual viruses: friction and mechanical properties”. Biophysical Journal. 72 (3): 1396–403. Bibcode:1997BpJ….72.1396F. doi:10.1016/S0006-3495(97)78786-1. PMC 1184522. PMID 9138585.
- ^ Kuznetsov YG, Malkin AJ, Lucas RW, Plomp M, McPherson A (September 2001). “Imaging of viruses by atomic force microscopy”. The Journal of General Virology. 82 (Pt 9): 2025–34. doi:10.1099/0022-1317-82-9-2025. PMID 11514711.
- ^ Collier p. 37
- ^ Straus SK, Bo HE (2018). “Filamentous Bacteriophage Proteins and Assembly”. Sub-Cellular Biochemistry. 88: 261–279. doi:10.1007/978-981-10-8456-0_12. ISBN 978-981-10-8455-3. PMID 29900501.
- ^ Collier pp. 40, 42
- ^ Wilson DP (2016). “Protruding Features of Viral Capsids Are Clustered on Icosahedral Great Circles”. PLOS ONE. 11 (4): e0152319. Bibcode:2016PLoSO..1152319W. doi:10.1371/journal.pone.0152319. PMC 4821576. PMID 27045511.
- ^ Casens S (2009). Desk Encyclopedia of General Virology. Boston: Academic Press. pp. 167–74. ISBN 978-0-12-375146-1.
- ^ Dhama K, Khan S, Tiwari R, Sircar S, Bhat S, Malik YS, Singh KP, Chaicumpa W, Bonilla-Aldana DK, Rodriguez-Morales AJ (September 2020). “Coronavirus Disease 2019-COVID-19”. Clinical Microbiology Reviews. 33 (4). doi:10.1128/CMR.00028-20. PMC 7405836. PMID 32580969.
- ^ Collier pp. 42–43
- ^ Rossmann MG, Mesyanzhinov VV, Arisaka F, Leiman PG (April 2004). “The bacteriophage T4 DNA injection machine”. Current Opinion in Structural Biology. 14 (2): 171–80. doi:10.1016/j.sbi.2004.02.001. PMID 15093831.
- ^ Long GW, Nobel J, Murphy FA, Herrmann KL, Lourie B (September 1970). “Experience with electron microscopy in the differential diagnosis of smallpox”. Applied Microbiology. 20 (3): 497–504. doi:10.1128/AEM.20.3.497-504.1970. PMC 376966. PMID 4322005.
- ^ Suzan-Monti M, La Scola B, Raoult D (April 2006). “Genomic and evolutionary aspects of Mimivirus”. Virus Research. 117 (1): 145–55. doi:10.1016/j.virusres.2005.07.011. PMID 16181700.
- ^ Arslan D, Legendre M, Seltzer V, Abergel C, Claverie JM (October 2011). “Distant Mimivirus relative with a larger genome highlights the fundamental features of Megaviridae”. Proceedings of the National Academy of Sciences of the United States of America. 108 (42): 17486–91. Bibcode:2011PNAS..10817486A. doi:10.1073/pnas.1110889108. PMC 3198346. PMID 21987820.
- ^ a b Philippe N, Legendre M, Doutre G, Couté Y, Poirot O, Lescot M, et al. (July 2013). “Pandoraviruses: amoeba viruses with genomes up to 2.5 Mb reaching that of parasitic eukaryotes” (PDF). Science. 341 (6143): 281–86. Bibcode:2013Sci…341..281P. doi:10.1126/science.1239181. PMID 23869018. S2CID 16877147.
- ^ Brandes N, Linial M (April 2019). “Giant Viruses-Big Surprises”. Viruses. 11 (5): 404. doi:10.3390/v11050404. PMC 6563228. PMID 31052218.
- ^ a b Prangishvili D, Forterre P, Garrett RA (November 2006). “Viruses of the Archaea: a unifying view”. Nature Reviews. Microbiology. 4 (11): 837–48. doi:10.1038/nrmicro1527. PMID 17041631. S2CID 9915859.
- ^ a b Dimmock p. 49
- ^ “NCBI Viral Genome database”. ncbi.nlm.nih.gov. Retrieved 15 January 2017.
- ^ Pennisi E (March 2011). “Microbiology. Going viral: exploring the role of viruses in our bodies”. Science. 331 (6024): 1513. Bibcode:2011Sci…331.1513P. doi:10.1126/science.331.6024.1513. PMID 21436418.
- ^ Shi M, Lin XD, Tian JH, Chen LJ, Chen X, Li CX, et al. (December 2016). “Redefining the invertebrate RNA virosphere”. Nature. 540 (7634): 539–43. Bibcode:2016Natur.540..539S. doi:10.1038/nature20167. PMID 27880757. S2CID 1198891.
- ^ a b c Collier pp. 96–99
- ^ Saunders VA, Carter J (2007). Virology: principles and applications. Chichester: John Wiley & Sons. p. 72. ISBN 978-0-470-02387-7.
- ^ Belyi VA, Levine AJ, Skalka AM (December 2010). “Sequences from ancestral single-stranded DNA viruses in vertebrate genomes: the parvoviridae and circoviridae are more than 40 to 50 million years old”. Journal of Virology. 84 (23): 12458–62. doi:10.1128/JVI.01789-10. PMC 2976387. PMID 20861255.
- ^ Brandes N, Linial M (May 2016). “Gene overlapping and size constraints in the viral world”. Biology Direct. 11 (1): 26. doi:10.1186/s13062-016-0128-3. PMC 4875738. PMID 27209091.
- ^ Pressing J, Reanney DC (1984). “Divided genomes and intrinsic noise”. Journal of Molecular Evolution. 20 (2): 135–46. Bibcode:1984JMolE..20..135P. doi:10.1007/BF02257374. PMC 7087551. PMID 6433032.
- ^ Duffy S, Holmes EC (June 2009). “Validation of high rates of nucleotide substitution in geminiviruses: phylogenetic evidence from East African cassava mosaic viruses”. The Journal of General Virology. 90 (Pt 6): 1539–47. doi:10.1099/vir.0.009266-0. PMC 4091138. PMID 19264617.
- ^ Sandbulte MR, Westgeest KB, Gao J, Xu X, Klimov AI, Russell CA, et al. (December 2011). “Discordant antigenic drift of neuraminidase and hemagglutinin in H1N1 and H3N2 influenza viruses”. Proceedings of the National Academy of Sciences of the United States of America. 108 (51): 20748–53. Bibcode:2011PNAS..10820748S. doi:10.1073/pnas.1113801108. PMC 3251064. PMID 22143798.
- ^ Moss RB, Davey RT, Steigbigel RT, Fang F (June 2010). “Targeting pandemic influenza: a primer on influenza antivirals and drug resistance”. The Journal of Antimicrobial Chemotherapy. 65 (6): 1086–93. doi:10.1093/jac/dkq100. PMID 20375034.
- ^ Hampson AW, Mackenzie JS (November 2006). “The influenza viruses”. The Medical Journal of Australia. 185 (S10): S39–43. doi:10.5694/j.1326-5377.2006.tb00705.x. PMID 17115950. S2CID 17069567.
- ^ Metzner KJ (December 2006). “Detection and significance of minority quasispecies of drug-resistant HIV-1”. Journal of HIV Therapy. 11 (4): 74–81. PMID 17578210.
- ^ Goudsmit, Jaap. Viral Sex. Oxford Univ Press, 1998. ISBN 978-0-19-512496-5, 0-19-512496-0
- ^ Worobey M, Holmes EC (October 1999). “Evolutionary aspects of recombination in RNA viruses”. The Journal of General Virology. 80 (10): 2535–43. doi:10.1099/0022-1317-80-10-2535. PMID 10573145.
- ^ Lukashev AN (2005). “Role of recombination in evolution of enteroviruses”. Reviews in Medical Virology. 15 (3): 157–67. doi:10.1002/rmv.457. PMID 15578739. S2CID 26000112.
- ^ Umene K (July 1999). “Mechanism and application of genetic recombination in herpesviruses”. Reviews in Medical Virology. 9 (3): 171–82. doi:10.1002/(SICI)1099-1654(199907/09)9:3<171::AID-RMV243>3.0.CO;2-A. PMID 10479778. S2CID 43110533.
- ^ Su S, Wong G, Shi W, Liu J, Lai ACK, Zhou J, Liu W, Bi Y, Gao GF. Epidemiology, Genetic Recombination, and Pathogenesis of Coronaviruses. Trends Microbiol. 2016 Jun;24(6):490-502. doi: 10.1016/j.tim.2016.03.003. Epub 2016 Mar 21. PMID: 27012512; PMCID: PMC7125511
- ^ Barr JN, Fearns R. How RNA viruses maintain their genome integrity. J Gen Virol. 2010 Jun;91(Pt 6):1373-87. doi: 10.1099/vir.0.020818-0. Epub 2010 Mar 24. PMID: 20335491
- ^ Freed EO (August 2015). “HIV-1 assembly, release and maturation”. Nature Reviews. Microbiology. 13 (8): 484–96. doi:10.1038/nrmicro3490. PMC 6936268. PMID 26119571.
- ^ Yin J, Redovich J (June 2018). “Kinetic Modeling of Virus Growth in Cells”. Microbiology and Molecular Biology Reviews. 82 (2). doi:10.1128/MMBR.00066-17. PMC 5968458. PMID 29592895.
- ^ Collier pp. 75–91
- ^ Más V, Melero JA (2013). “Entry of enveloped viruses into host cells: membrane fusion”. Structure and Physics of Viruses. Subcellular Biochemistry. Vol. 68. pp. 467–87. doi:10.1007/978-94-007-6552-8_16. ISBN 978-94-007-6551-1. PMC 7121288. PMID 23737062.
- ^ Dimmock p. 70
- ^ Boevink P, Oparka KJ (August 2005). “Virus-host interactions during movement processes”. Plant Physiology. 138 (4): 1815–21. doi:10.1104/pp.105.066761. PMC 1183373. PMID 16172094.
- ^ Dimmock p. 71
- ^ Blaas D (May 2016). “Viral entry pathways: the example of common cold viruses”. Wiener Medizinische Wochenschrift. 166 (7–8): 211–26. doi:10.1007/s10354-016-0461-2. PMC 4871925. PMID 27174165.
- ^ Isomura H, Stinski MF (February 2013). “Coordination of late gene transcription of human cytomegalovirus with viral DNA synthesis: recombinant viruses as potential therapeutic vaccine candidates”. Expert Opinion on Therapeutic Targets. 17 (2): 157–66. doi:10.1517/14728222.2013.740460. PMID 23231449. S2CID 11448687.
- ^ Barman S, Ali A, Hui EK, Adhikary L, Nayak DP (September 2001). “Transport of viral proteins to the apical membranes and interaction of matrix protein with glycoproteins in the assembly of influenza viruses”. Virus Research. 77 (1): 61–69. doi:10.1016/S0168-1702(01)00266-0. PMID 11451488.
- ^ Shors pp. 836
- ^ Dimmock, Chapter 15, Mechanisms in virus latency, pp. 243–59
- ^ Dimmock 185–87
- ^ Shors p. 118; Collier p. 78
- ^ Collier p. 79
- ^ Staginnus C, Richert-Pöggeler KR (October 2006). “Endogenous pararetroviruses: two-faced travelers in the plant genome”. Trends in Plant Science. 11 (10): 485–91. doi:10.1016/j.tplants.2006.08.008. PMID 16949329.
- ^ Collier pp. 88–89
- ^ Collier pp. 115–46
- ^ Collier p. 115
- ^ Roulston A, Marcellus RC, Branton PE (1999). “Viruses and apoptosis”. Annual Review of Microbiology. 53: 577–628. doi:10.1146/annurev.micro.53.1.577. PMID 10547702.
- ^ Alwine JC (2008). “Modulation of host cell stress responses by human cytomegalovirus”. Current Topics in Microbiology and Immunology. 325: 263–79. doi:10.1007/978-3-540-77349-8_15. ISBN 978-3-540-77348-1. PMID 18637511.
- ^ Barozzi P, Potenza L, Riva G, Vallerini D, Quadrelli C, Bosco R, et al. (December 2007). “B cells and herpesviruses: a model of lymphoproliferation”. Autoimmunity Reviews. 7 (2): 132–36. doi:10.1016/j.autrev.2007.02.018. PMID 18035323.
- ^ Subramanya D, Grivas PD (November 2008). “HPV and cervical cancer: updates on an established relationship”. Postgraduate Medicine. 120 (4): 7–13. doi:10.3810/pgm.2008.11.1928. PMID 19020360. S2CID 1399003.
- ^ Sinclair J (March 2008). “Human cytomegalovirus: Latency and reactivation in the myeloid lineage”. Journal of Clinical Virology. 41 (3): 180–85. doi:10.1016/j.jcv.2007.11.014. PMID 18164651.
- ^ Jordan MC, Jordan GW, Stevens JG, Miller G (June 1984). “Latent herpesviruses of humans”. Annals of Internal Medicine. 100 (6): 866–80. doi:10.7326/0003-4819-100-6-866. PMID 6326635.
- ^ Sissons JG, Bain M, Wills MR (February 2002). “Latency and reactivation of human cytomegalovirus”. The Journal of Infection. 44 (2): 73–77. doi:10.1053/jinf.2001.0948. PMID 12076064.
- ^ Crawford DH (2011). Viruses: A Very Short Introduction. Oxford University Press, US. pp. 16. ISBN 978-0-19-957485-8.
- ^ Shors p. 643
- ^ Shors p. 631
- ^ Dimmock p. 272
- ^ Baggesen DL, Sørensen G, Nielsen EM, Wegener HC (January 2010). “Phage typing of Salmonella Typhimurium – is it still a useful tool for surveillance and outbreak investigation?”. Euro Surveillance. 15 (4): 19471. PMID 20122382. Retrieved 19 December 2014.
- ^ Parker MT (September 2016). “An Ecological Framework of the Human Virome Provides Classification of Current Knowledge and Identifies Areas of Forthcoming Discovery”. The Yale Journal of Biology and Medicine. 89 (3): 339–51. PMC 5045143. PMID 27698618.
- ^ Woolhouse M, Scott F, Hudson Z, Howey R, Chase-Topping M (October 2012). “Human viruses: discovery and emergence”. Philosophical Transactions of the Royal Society of London. Series B, Biological Sciences. 367 (1604): 2864–71. doi:10.1098/rstb.2011.0354. PMC 3427559. PMID 22966141.
- ^ a b Ashour HM, Elkhatib WF, Rahman MM, Elshabrawy HA (March 2020). “Insights into the Recent 2019 Novel Coronavirus (SARS-CoV-2) in Light of Past Human Coronavirus Outbreaks”. Pathogens. 9 (3): 186. doi:10.3390/pathogens9030186. PMC 7157630. PMID 32143502.
- ^ Lwoff A, Horne RW, Tournier P (June 1962). “[A virus system]”. Comptes Rendus Hebdomadaires des Séances de l’Académie des Sciences (in French). 254: 4225–27. PMID 14467544.
- ^ Lwoff A, Horne R, Tournier P (1962). “A system of viruses”. Cold Spring Harbor Symposia on Quantitative Biology. 27: 51–55. doi:10.1101/sqb.1962.027.001.008. PMID 13931895.
- ^ Fauquet CM, Fargette D (August 2005). “International Committee on Taxonomy of Viruses and the 3,142 unassigned species”. Virology Journal. 2: 64. doi:10.1186/1743-422X-2-64. PMC 1208960. PMID 16105179.
- ^ International Committee on Taxonomy of Viruses Executive Committee (May 2020). “The New Scope of Virus Taxonomy: Partitioning the Virosphere Into 15 Hierarchical Ranks”. Nat Microbiol. 5 (5): 668–674. doi:10.1038/s41564-020-0709-x. PMC 7186216. PMID 32341570.
- ^ Khan MK, Alam MM (July 2021). “Norovirus Gastroenteritis Outbreaks, Genomic Diversity and Evolution: An Overview”. Mymensingh Medical Journal. 30 (3): 863–873. PMID 34226482.
- ^ Eberle J, Gürtler L (2012). “HIV types, groups, subtypes and recombinant forms: errors in replication, selection pressure and quasispecies”. Intervirology. 55 (2): 79–83. doi:10.1159/000331993. PMID 22286874. S2CID 5642060.
- ^ Gorbalenya, Alexander E.; Krupovic, Mart; Mushegian, Arcady; Kropinski, Andrew M.; Siddell, Stuart G.; Varsani, Arvind; Adams, Michael J.; Davison, Andrew J.; Dutilh, Bas E.; Harrach, Balázs; Harrison, Robert L.; Junglen, Sandra; King, Andrew M. Q.; Knowles, Nick J.; Lefkowitz, Elliot J.; Nibert, Max L.; Rubino, Luisa; Sabanadzovic, Sead; Sanfaçon, Hélène; Simmonds, Peter; Walker, Peter J.; Zerbini, F. Murilo; Kuhn, Jens H. (May 2020). “The new scope of virus taxonomy: partitioning the virosphere into 15 hierarchical ranks”. Nature Microbiology. 5 (5): 668–674. doi:10.1038/s41564-020-0709-x. PMC 7186216. PMID 32341570.
- ^ Delwart EL (2007). “Viral metagenomics”. Reviews in Medical Virology. 17 (2): 115–31. doi:10.1002/rmv.532. PMC 7169062. PMID 17295196.
- ^ Temin HM, Baltimore D (1972). “RNA-directed DNA synthesis and RNA tumor viruses”. Advances in Virus Research. 17: 129–86. doi:10.1016/S0065-3527(08)60749-6. ISBN 9780120398171. PMID 4348509.
- ^ Baltimore D (1974). “The strategy of RNA viruses”. Harvey Lectures. 70 Series. 70 Series: 57–74. PMID 4377923.
- ^ van Regenmortel MH, Mahy BW (January 2004). “Emerging issues in virus taxonomy”. Emerging Infectious Diseases. 10 (1): 8–13. doi:10.3201/eid1001.030279. PMC 3322749. PMID 15078590.
- ^ Mayo MA (1999). “Developments in plant virus taxonomy since the publication of the 6th ICTV Report. International Committee on Taxonomy of Viruses”. Archives of Virology. 144 (8): 1659–66. doi:10.1007/s007050050620. PMID 10486120. S2CID 33422303.
- ^ de Villiers EM, Fauquet C, Broker TR, Bernard HU, zur Hausen H (June 2004). “Classification of papillomaviruses”. Virology. 324 (1): 17–27. doi:10.1016/j.virol.2004.03.033. PMID 15183049.
- ^ Mainly Chapter 33 (Disease summaries), pp. 367–92 in:Fisher B, Harvey RP, Champe PC (2007). Lippincott’s Illustrated Reviews: Microbiology. Lippincott’s Illustrated Reviews Series. Hagerstwon, MD: Lippincott Williams & Wilkins. pp. 367–92. ISBN 978-0-7817-8215-9.
- ^ Komaroff AL (December 2006). “Is human herpesvirus-6 a trigger for chronic fatigue syndrome?”. Journal of Clinical Virology. 37 (Suppl 1): S39–46. doi:10.1016/S1386-6532(06)70010-5. PMID 17276367.
- ^ Chen CH, Chiu YL, Wei FC, Koong FJ, Liu HC, Shaw CK, et al. (January 1999). “High seroprevalence of Borna virus infection in schizophrenic patients, family members and mental health workers in Taiwan”. Molecular Psychiatry. 4 (1): 33–38. doi:10.1038/sj.mp.4000484. PMID 10089006. S2CID 19830976.
- ^ Margolis TP, Elfman FL, Leib D, Pakpour N, Apakupakul K, Imai Y, Voytek C (October 2007). “Spontaneous reactivation of herpes simplex virus type 1 in latently infected murine sensory ganglia”. Journal of Virology. 81 (20): 11069–74. doi:10.1128/JVI.00243-07. PMC 2045564. PMID 17686862.
- ^ Whitley RJ, Roizman B (May 2001). “Herpes simplex virus infections”. Lancet. 357 (9267): 1513–18. doi:10.1016/S0140-6736(00)04638-9. PMID 11377626. S2CID 9854903.
- ^ Barton ES, White DW, Cathelyn JS, Brett-McClellan KA, Engle M, Diamond MS, et al. (May 2007). “Herpesvirus latency confers symbiotic protection from bacterial infection”. Nature. 447 (7142): 326–29. Bibcode:2007Natur.447..326B. doi:10.1038/nature05762. PMID 17507983. S2CID 4425405.
- ^ Bertoletti A, Gehring A (October 2007). “Immune response and tolerance during chronic hepatitis B virus infection”. Hepatology Research. 37 (Suppl 3): S331–38. doi:10.1111/j.1872-034X.2007.00221.x. PMID 17931183. S2CID 13386004.
- ^ Rodrigues C, Deshmukh M, Jacob T, Nukala R, Menon S, Mehta A (2001). “Significance of HBV DNA by PCR over serological markers of HBV in acute and chronic patients”. Indian Journal of Medical Microbiology. 19 (3): 141–44. PMID 17664817.
- ^ Nguyen VT, McLaws ML, Dore GJ (December 2007). “Highly endemic hepatitis B infection in rural Vietnam”. Journal of Gastroenterology and Hepatology. 22 (12): 2093–100. doi:10.1111/j.1440-1746.2007.05010.x. PMID 17645465. S2CID 29885790.
- ^ Fowler MG, Lampe MA, Jamieson DJ, Kourtis AP, Rogers MF (September 2007). “Reducing the risk of mother-to-child human immunodeficiency virus transmission: past successes, current progress and challenges, and future directions”. American Journal of Obstetrics and Gynecology. 197 (3 Suppl): S3–9. doi:10.1016/j.ajog.2007.06.048. PMID 17825648.
- ^ Sauerbrei A, Wutzler P (December 2000). “The congenital varicella syndrome”. Journal of Perinatology. 20 (8 Pt 1): 548–54. doi:10.1038/sj.jp.7200457. PMID 11190597. S2CID 7973561.
- ^ a b Antonovics J, Wilson AJ, Forbes MR, Hauffe HC, Kallio ER, Leggett HC, Longdon B, Okamura B, Sait SM, Webster JP (May 2017). “The evolution of transmission mode”. Philosophical Transactions of the Royal Society of London. Series B, Biological Sciences. 372 (1719). doi:10.1098/rstb.2016.0083. PMC 5352810. PMID 28289251.
- ^ Garnett GP (February 2005). “Role of herd immunity in determining the effect of vaccines against sexually transmitted disease”. The Journal of Infectious Diseases. 191 (Suppl 1): S97–106. doi:10.1086/425271. PMID 15627236.
- ^ Platonov AE (2006). “[The influence of weather conditions on the epidemiology of vector-borne diseases by the example of West Nile fever in Russia]”. Vestnik Rossiiskoi Akademii Meditsinskikh Nauk (in Russian) (2): 25–29. PMID 16544901.
- ^ Shors p. 264
- ^ Shors pp. 894
- ^ Jewell CP, Keeling MJ, Roberts GO (December 2009). “Predicting undetected infections during the 2007 foot-and-mouth disease outbreak”. Journal of the Royal Society, Interface. 6 (41): 1145–51. doi:10.1098/rsif.2008.0433. PMC 2817150. PMID 19091686.
- ^ Shors p. 170
- ^ a b Shors pp. 170–72
- ^ Shors p. 272
- ^ Shors pp. 891
- ^ Collier pp. 409–15
- ^ Patterson KD, Pyle GF (1991). “The geography and mortality of the 1918 influenza pandemic”. Bulletin of the History of Medicine. 65 (1): 4–21. PMID 2021692.
- ^ Johnson NP, Mueller J (2002). “Updating the accounts: global mortality of the 1918-1920 “Spanish” influenza pandemic”. Bulletin of the History of Medicine. 76 (1): 105–15. doi:10.1353/bhm.2002.0022. PMID 11875246. S2CID 22974230.
- ^ Eisinger RW, Fauci AS (March 2018). “1”. Emerging Infectious Diseases. 24 (3): 413–16. doi:10.3201/eid2403.171797. PMC 5823353. PMID 29460740.
- ^ Qin Y, Zhao MJ, Tan YY, Li XQ, Zheng JD, Peng ZB, Feng LZ (August 2018). “[History of influenza pandemics in China during the past century]”. Zhonghua Liu Xing Bing Xue Za Zhi = Zhonghua Liuxingbingxue Zazhi (in Chinese). 39 (8): 1028–31. doi:10.3760/cma.j.issn.0254-6450.2018.08.003. PMID 30180422.
- ^ Gao F, Bailes E, Robertson DL, Chen Y, Rodenburg CM, Michael SF, et al. (February 1999). “Origin of HIV-1 in the chimpanzee Pan troglodytes troglodytes”. Nature. 397 (6718): 436–41. Bibcode:1999Natur.397..436G. doi:10.1038/17130. PMID 9989410. S2CID 4432185.
- ^ “Fact Sheet” (PDF). UNAIDS.org. 2018. Retrieved 12 December 2019.
- ^ “UN AIDS DATA2019”. UNAIDS.org. 2019. Retrieved 5 December 2019.
- ^ Mawar N, Saha S, Pandit A, Mahajan U (December 2005). “The third phase of HIV pandemic: social consequences of HIV/AIDS stigma & discrimination & future needs” (PDF). The Indian Journal of Medical Research. 122 (6): 471–84. PMID 16517997. Archived from the original (PDF) on 4 March 2016. Retrieved 19 December 2014.
- ^ “Status of the global HIV epidemic” (PDF). UNAIDS. 2008. Archived from the original (PDF) on 22 November 2015. Retrieved 19 December 2014.
- ^ Towner JS, Khristova ML, Sealy TK, Vincent MJ, Erickson BR, Bawiec DA, et al. (July 2006). “Marburgvirus genomics and association with a large hemorrhagic fever outbreak in Angola”. Journal of Virology. 80 (13): 6497–516. doi:10.1128/JVI.00069-06. PMC 1488971. PMID 16775337.
- ^ “World Health Organisation report, 24 September 2014” (PDF).
- ^ “Virology Journal”. Virology Journal.
- ^ Weiss SR, Leibowitz JL (2011). Coronavirus pathogenesis. Advances in Virus Research. Vol. 81. pp. 85–164. doi:10.1016/B978-0-12-385885-6.00009-2. ISBN 978-0-12-385885-6. PMC 7149603. PMID 22094080.
- ^ Wong AT, Chen H, Liu SH, Hsu EK, Luk KS, Lai CK, et al. (May 2017). “From SARS to Avian Influenza Preparedness in Hong Kong”. Clinical Infectious Diseases. 64 (suppl_2): S98–S104. doi:10.1093/cid/cix123. PMID 28475794.
- ^ Deng SQ, Peng HJ (February 2020). “Characteristics of and Public Health Responses to the Coronavirus Disease 2019 Outbreak in China”. Journal of Clinical Medicine. 9 (2): 575. doi:10.3390/jcm9020575. PMC 7074453. PMID 32093211.
- ^ Han Q, Lin Q, Jin S, You L (April 2020). “Coronavirus 2019-nCoV: A brief perspective from the front line”. The Journal of Infection. 80 (4): 373–77. doi:10.1016/j.jinf.2020.02.010. PMC 7102581. PMID 32109444.
- ^ Londoño E, Ortiz A (16 March 2020). “Coronavirus Travel Restrictions, Across the Globe”. The New York Times.
- ^ “US takes more big pandemic response steps; Europe COVID-19 cases soar”. CIDRAP.
- ^ Einstein MH, Schiller JT, Viscidi RP, Strickler HD, Coursaget P, Tan T, et al. (June 2009). “Clinician’s guide to human papillomavirus immunology: knowns and unknowns”. The Lancet. Infectious Diseases. 9 (6): 347–56. doi:10.1016/S1473-3099(09)70108-2. PMID 19467474.
- ^ Shuda M, Feng H, Kwun HJ, Rosen ST, Gjoerup O, Moore PS, Chang Y (October 2008). “T antigen mutations are a human tumor-specific signature for Merkel cell polyomavirus”. Proceedings of the National Academy of Sciences of the United States of America. 105 (42): 16272–77. Bibcode:2008PNAS..10516272S. doi:10.1073/pnas.0806526105. PMC 2551627. PMID 18812503.
- ^ Pulitzer MP, Amin BD, Busam KJ (May 2009). “Merkel cell carcinoma: review”. Advances in Anatomic Pathology. 16 (3): 135–44. doi:10.1097/PAP.0b013e3181a12f5a. PMID 19395876. S2CID 36110778.
- ^ Koike K (June 2007). “Hepatitis C virus contributes to hepatocarcinogenesis by modulating metabolic and intracellular signaling pathways”. Journal of Gastroenterology and Hepatology. 22 (Suppl 1): S108–11. doi:10.1111/j.1440-1746.2006.04669.x. PMID 17567457. S2CID 25399220.
- ^ Hu J, Ludgate L (2007). “HIV-HBV and HIV-HCV coinfection and liver cancer development”. Cancer Treatment and Research. 133: 241–52. doi:10.1007/978-0-387-46816-7_9. ISBN 978-0-387-46804-4. PMID 17672044.
- ^ Bellon M, Nicot C (2007). “Telomerase: a crucial player in HTLV-I-induced human T-cell leukemia”. Cancer Genomics & Proteomics. 4 (1): 21–25. PMID 17726237.
- ^ Schiffman M, Castle PE, Jeronimo J, Rodriguez AC, Wacholder S (September 2007). “Human papillomavirus and cervical cancer”. Lancet. 370 (9590): 890–907. doi:10.1016/S0140-6736(07)61416-0. PMID 17826171. S2CID 20196938.
- ^ Klein E, Kis LL, Klein G (February 2007). “Epstein-Barr virus infection in humans: from harmless to life endangering virus-lymphocyte interactions”. Oncogene. 26 (9): 1297–305. doi:10.1038/sj.onc.1210240. PMID 17322915.
- ^ Zur Hausen H (July 2008). “Novel human polyomaviruses – re-emergence of a well known virus family as possible human carcinogens”. International Journal of Cancer. 123 (2): 247–50. doi:10.1002/ijc.23620. PMID 18449881. S2CID 9482506.
- ^ Alberta B, Johnson A, Lewis J, Raff M, Roberts K, Walters P (2002). Molecular Biology of the Cell (Fourth ed.). New York and London: Garland Science. ISBN 0-8153-3218-1.
- ^ Ding SW, Voinnet O (August 2007). “Antiviral immunity directed by small RNAs”. Cell. 130 (3): 413–26. doi:10.1016/j.cell.2007.07.039. PMC 2703654. PMID 17693253.
- ^ Patton JT, Vasquez-Del Carpio R, Spencer E (2004). “Replication and transcription of the rotavirus genome”. Current Pharmaceutical Design. 10 (30): 3769–77. doi:10.2174/1381612043382620. PMID 15579070.
- ^ Jayaram H, Estes MK, Prasad BV (April 2004). “Emerging themes in rotavirus cell entry, genome organization, transcription and replication”. Virus Research. 101 (1): 67–81. doi:10.1016/j.virusres.2003.12.007. PMID 15010218.
- ^ Greer S, Alexander GJ (December 1995). “Viral serology and detection”. Bailliere’s Clinical Gastroenterology. 9 (4): 689–721. doi:10.1016/0950-3528(95)90057-8. PMID 8903801.
- ^ Matter L, Kogelschatz K, Germann D (April 1997). “Serum levels of rubella virus antibodies indicating immunity: response to vaccination of subjects with low or undetectable antibody concentrations”. The Journal of Infectious Diseases. 175 (4): 749–55. doi:10.1086/513967. PMID 9086126.
- ^ Mallery DL, McEwan WA, Bidgood SR, Towers GJ, Johnson CM, James LC (November 2010). “Antibodies mediate intracellular immunity through tripartite motif-containing 21 (TRIM21)”. Proceedings of the National Academy of Sciences of the United States of America. 107 (46): 19985–90. Bibcode:2010PNAS..10719985M. doi:10.1073/pnas.1014074107. PMC 2993423. PMID 21045130.
- ^ Cascalho M, Platt JL (2007). “Novel functions of B cells”. Critical Reviews in Immunology. 27 (2): 141–51. doi:10.1615/critrevimmunol.v27.i2.20. PMID 17725500.
- ^ Le Page C, Génin P, Baines MG, Hiscott J (2000). “Interferon activation and innate immunity”. Reviews in Immunogenetics. 2 (3): 374–86. PMID 11256746.
- ^ Hilleman MR (October 2004). “Strategies and mechanisms for host and pathogen survival in acute and persistent viral infections”. Proceedings of the National Academy of Sciences of the United States of America. 101 (Suppl 2): 14560–66. Bibcode:2004PNAS..10114560H. doi:10.1073/pnas.0404758101. PMC 521982. PMID 15297608.
- ^ Libbey JE, Fujinami RS (2014). “Adaptive immune response to viral infections in the central nervous system”. Handbook of Clinical Neurology. 123: 225–47. doi:10.1016/B978-0-444-53488-0.00010-9. ISBN 9780444534880. PMC 4370180. PMID 25015488.
- ^ Asaria P, MacMahon E (October 2006). “Measles in the United Kingdom: can we eradicate it by 2010?”. BMJ. 333 (7574): 890–95. doi:10.1136/bmj.38989.445845.7C. PMC 1626346. PMID 17068034.
- ^ Lane JM (2006). “Mass vaccination and surveillance/containment in the eradication of smallpox”. Current Topics in Microbiology and Immunology. 304: 17–29. doi:10.1007/3-540-36583-4_2. ISBN 978-3-540-29382-8. PMC 7120753. PMID 16989262.
- ^ Arvin AM, Greenberg HB (January 2006). “New viral vaccines”. Virology. 344 (1): 240–49. doi:10.1016/j.virol.2005.09.057. PMID 16364754.
- ^ Pastoret PP, Schudel AA, Lombard M (August 2007). “Conclusions–future trends in veterinary vaccinology”. Revue Scientifique et Technique. 26 (2): 489–94, 495–501, 503–09. doi:10.20506/rst.26.2.1759. PMID 17892169.
- ^ Palese P (January 2006). “Making better influenza virus vaccines?”. Emerging Infectious Diseases. 12 (1): 61–65. doi:10.3201/eid1201.051043. PMC 3291403. PMID 16494719.
- ^ Anand P, Stahel VP (May 2021). “Review the safety of Covid-19 mRNA vaccines: a review”. Patient Safety in Surgery. 15 (1): 20. doi:10.1186/s13037-021-00291-9. PMC 8087878. PMID 33933145.
- ^ Thomssen R (1975). “Live attenuated versus killed virus vaccines”. Monographs in Allergy. 9: 155–76. PMID 1090805.
- ^ McLean AA (1986). “Development of vaccines against hepatitis A and hepatitis B”. Reviews of Infectious Diseases. 8 (4): 591–98. doi:10.1093/clinids/8.4.591. PMID 3018891.
- ^ Casswall TH, Fischler B (October 2005). “Vaccination of the immunocompromised child”. Expert Review of Vaccines. 4 (5): 725–38. doi:10.1586/14760584.4.5.725. PMID 16221073. S2CID 40821818.
- ^ Barnett ED, Wilder-Smith A, Wilson ME (July 2008). “Yellow fever vaccines and international travelers”. Expert Review of Vaccines. 7 (5): 579–87. doi:10.1586/14760584.7.5.579. PMID 18564013. S2CID 19352868.
- ^ a b De Clercq E, Li G (July 2016). “Approved Antiviral Drugs over the Past 50 Years”. Clinical Microbiology Reviews. 29 (3): 695–747. doi:10.1128/CMR.00102-15. PMC 4978613. PMID 27281742.
- ^ Magden J, Kääriäinen L, Ahola T (March 2005). “Inhibitors of virus replication: recent developments and prospects”. Applied Microbiology and Biotechnology. 66 (6): 612–21. doi:10.1007/s00253-004-1783-3. PMC 7082807. PMID 15592828.
- ^ Mindel A, Sutherland S (September 1983). “Genital herpes – the disease and its treatment including intravenous acyclovir”. The Journal of Antimicrobial Chemotherapy. 12 (Suppl B): 51–59. doi:10.1093/jac/12.suppl_b.51. PMID 6355051.
- ^ Palmisano L, Vella S (2011). “A brief history of antiretroviral therapy of HIV infection: success and challenges”. Annali dell’Istituto Superiore di Sanità. 47 (1): 44–48. doi:10.4415/ANN_11_01_10. PMID 21430338.
- ^ Falade-Nwulia O, Suarez-Cuervo C, Nelson DR, Fried MW, Segal JB, Sulkowski MS (May 2017). “Oral Direct-Acting Agent Therapy for Hepatitis C Virus Infection: A Systematic Review”. Annals of Internal Medicine. 166 (9): 637–648. doi:10.7326/M16-2575. PMC 5486987. PMID 28319996.
- ^ Nguyen MH, Wong G, Gane E, Kao JH, Dusheiko G (March 2020). “Hepatitis B Virus: Advances in Prevention, Diagnosis, and Therapy”. Clinical Microbiology Reviews. 33 (2). doi:10.1128/CMR.00046-19. PMC 7048015. PMID 32102898.
- ^ Dimmock p. 3
- ^ Goris N, Vandenbussche F, De Clercq K (April 2008). “Potential of antiviral therapy and prophylaxis for controlling RNA viral infections of livestock”. Antiviral Research. 78 (1): 170–78. doi:10.1016/j.antiviral.2007.10.003. PMID 18035428.
- ^ Carmichael LE (2005). “An annotated historical account of canine parvovirus”. Journal of Veterinary Medicine. B, Infectious Diseases and Veterinary Public Health. 52 (7–8): 303–11. doi:10.1111/j.1439-0450.2005.00868.x. PMID 16316389.
- ^ Chen Y, Zhao Y, Hammond J, Hsu HT, Evans J, Feldlaufer M (October–November 2004). “Multiple virus infections in the honey bee and genome divergence of honey bee viruses”. Journal of Invertebrate Pathology. 87 (2–3): 84–93. doi:10.1016/j.jip.2004.07.005. PMID 15579317.
- ^ Hull, Roger (2002). “12”. Matthews’ Plant Virology (Fourth ed.). Academic Press. pp. 555–556. ISBN 9780123611604. Retrieved 21 February 2022.
- ^ Shors p. 802
- ^ Shors pp. 799–807
- ^ Zaheer K, Akhtar MH (2016). “Potato Production, Usage, and Nutrition – A Review”. Critical Reviews in Food Science and Nutrition. 56 (5): 711–21. doi:10.1080/10408398.2012.724479. PMID 24925679. S2CID 33074838.
- ^ Fuentes S, Jones RA, Matsuoka H, Ohshima K, Kreuze J, Gibbs AJ (July 2019). “Potato virus Y; the Andean connection”. Virus Evolution. 5 (2): vez037. doi:10.1093/ve/vez037. PMC 6755682. PMID 31559020.
- ^ Dinesh-Kumar SP, Tham WH, Baker BJ (December 2000). “Structure-function analysis of the tobacco mosaic virus resistance gene N”. Proceedings of the National Academy of Sciences of the United States of America. 97 (26): 14789–94. Bibcode:2000PNAS…9714789D. doi:10.1073/pnas.97.26.14789. PMC 18997. PMID 11121079.
- ^ Shors pp. 809
- ^ Soosaar JL, Burch-Smith TM, Dinesh-Kumar SP (October 2005). “Mechanisms of plant resistance to viruses”. Nature Reviews. Microbiology. 3 (10): 789–98. doi:10.1038/nrmicro1239. PMID 16132037. S2CID 27311732.
- ^ Lomonossoff GP (2011). “Virus Particles and the Uses of Such Particles in Bio- and Nanotechnology”. Recent Advances in Plant Virology. Caister Academic Press. ISBN 978-1-904455-75-2.
- ^ Wommack KE, Colwell RR (March 2000). “Virioplankton: viruses in aquatic ecosystems”. Microbiology and Molecular Biology Reviews. 64 (1): 69–114. doi:10.1128/MMBR.64.1.69-114.2000. PMC 98987. PMID 10704475.
- ^ Bergh O, Børsheim KY, Bratbak G, Heldal M (August 1989). “High abundance of viruses found in aquatic environments”. Nature. 340 (6233): 467–68. Bibcode:1989Natur.340..467B. doi:10.1038/340467a0. PMID 2755508. S2CID 4271861.
- ^ Shors pp. 834–35
- ^ Bickle TA, Krüger DH (June 1993). “Biology of DNA restriction”. Microbiological Reviews. 57 (2): 434–50. doi:10.1128/MMBR.57.2.434-450.1993. PMC 372918. PMID 8336674.
- ^ Barrangou R, Fremaux C, Deveau H, Richards M, Boyaval P, Moineau S, et al. (March 2007). “CRISPR provides acquired resistance against viruses in prokaryotes”. Science. 315 (5819): 1709–12. Bibcode:2007Sci…315.1709B. doi:10.1126/science.1138140. hdl:20.500.11794/38902. PMID 17379808. S2CID 3888761.
- ^ Brouns SJ, Jore MM, Lundgren M, Westra ER, Slijkhuis RJ, Snijders AP, et al. (August 2008). “Small CRISPR RNAs guide antiviral defense in prokaryotes”. Science. 321 (5891): 960–64. Bibcode:2008Sci…321..960B. doi:10.1126/science.1159689. PMC 5898235. PMID 18703739.
- ^ Mojica FJ, Rodriguez-Valera F (September 2016). “The discovery of CRISPR in archaea and bacteria”. The FEBS Journal. 283 (17): 3162–69. doi:10.1111/febs.13766. hdl:10045/57676. PMID 27234458. S2CID 42827598.
- ^ Prangishvili D, Garrett RA (April 2004). “Exceptionally diverse morphotypes and genomes of crenarchaeal hyperthermophilic viruses” (PDF). Biochemical Society Transactions. 32 (Pt 2): 204–08. doi:10.1042/BST0320204. PMID 15046572.
- ^ Mojica FJ, Díez-Villaseñor C, García-Martínez J, Soria E (February 2005). “Intervening sequences of regularly spaced prokaryotic repeats derive from foreign genetic elements”. Journal of Molecular Evolution. 60 (2): 174–82. Bibcode:2005JMolE..60..174M. doi:10.1007/s00239-004-0046-3. PMID 15791728. S2CID 27481111.
- ^ Makarova KS, Grishin NV, Shabalina SA, Wolf YI, Koonin EV (March 2006). “A putative RNA-interference-based immune system in prokaryotes: computational analysis of the predicted enzymatic machinery, functional analogies with eukaryotic RNAi, and hypothetical mechanisms of action”. Biology Direct. 1: 7. doi:10.1186/1745-6150-1-7. PMC 1462988. PMID 16545108.
- ^ van der Oost J, Westra ER, Jackson RN, Wiedenheft B (July 2014). “Unravelling the structural and mechanistic basis of CRISPR-Cas systems”. Nature Reviews. Microbiology. 12 (7): 479–92. doi:10.1038/nrmicro3279. PMC 4225775. PMID 24909109.
- ^ Dávila-Ramos S, Castelán-Sánchez HG, Martínez-Ávila L, Sánchez-Carbente MD, Peralta R, Hernández-Mendoza A, et al. (2019). “A Review on Viral Metagenomics in Extreme Environments”. Frontiers in Microbiology. 10: 2403. doi:10.3389/fmicb.2019.02403. PMC 6842933. PMID 31749771.
- ^ Zhang QY, Gui JF (December 2018). “Diversity, evolutionary contribution and ecological roles of aquatic viruses”. Science China Life Sciences. 61 (12): 1486–1502. doi:10.1007/s11427-018-9414-7. PMID 30443861. S2CID 53564176.
- ^ Weitz JS, Wilhelm SW (2013). “An ocean of viruses”. The Scientist. 27 (7): 35–39.
- ^ Suttle CA (September 2005). “Viruses in the sea”. Nature. 437 (7057): 356–61. Bibcode:2005Natur.437..356S. doi:10.1038/nature04160. PMID 16163346. S2CID 4370363.
- ^ Wilhelm SW, Suttle CA (1999). “Viruses and nutrient cycles in the sea: viruses play critical roles in the structure and function of aquatic food webs”. BioScience. 49 (10): 781–88. doi:10.2307/1313569. JSTOR 1313569.
- ^ Shelford EJ, Suttle CA (2018). “Virus-mediated transfer of nitrogen from heterotrophic bacteria to phytoplankton”. Biogeosciences. 15 (3): 809–15. Bibcode:2018BGeo…15..809S. doi:10.5194/bg-15-809-2018.
- ^ a b c d Suttle CA (October 2007). “Marine viruses – major players in the global ecosystem”. Nature Reviews. Microbiology. 5 (10): 801–12. doi:10.1038/nrmicro1750. PMID 17853907. S2CID 4658457.
- ^ Wigington CH, Sonderegger D, Brussaard CP, Buchan A, Finke JF, Fuhrman JA, et al. (January 2016). “Re-examination of the relationship between marine virus and microbial cell abundances” (PDF). Nature Microbiology. 1 (15024): 15024. doi:10.1038/nmicrobiol.2015.24. PMID 27572161. S2CID 52829633.
- ^ Brussaard CP (2004). “Viral control of phytoplankton populations – a review”. The Journal of Eukaryotic Microbiology. 51 (2): 125–38. doi:10.1111/j.1550-7408.2004.tb00537.x. PMID 15134247. S2CID 21017882.
- ^ Robbins J (13 April 2018). “Trillions Upon Trillions of Viruses Fall From the Sky Each Day”. The New York Times. Retrieved 14 April 2018.
- ^ Reche I, D’Orta G, Mladenov N, Winget DM, Suttle CA (April 2018). “Deposition rates of viruses and bacteria above the atmospheric boundary layer”. The ISME Journal. 12 (4): 1154–62. doi:10.1038/s41396-017-0042-4. PMC 5864199. PMID 29379178.
- ^ Hall AJ, Jepson PD, Goodman SJ, Harkonen T (2006). “Phocine distemper virus in the North and European Seas – data and models, nature and nurture”. Biological Conservation. 131 (2): 221–29. doi:10.1016/j.biocon.2006.04.008.
- ^ DeLong, John P.; Van Etten, James L.; Al-Ameeli, Zeina; Agarkova, Irina V.; Dunigan, David D. (3 January 2023). “The consumption of viruses returns energy to food chains”. Proceedings of the National Academy of Sciences. 120 (1): e2215000120. Bibcode:2023PNAS..12015000D. doi:10.1073/pnas.2215000120. ISSN 0027-8424. PMC 9910503. PMID 36574690. S2CID 255219850.
- ^ Irving, Michael (28 December 2022). “First “virovore” discovered: An organism that eats viruses”. New Atlas. Archived from the original on 29 December 2022. Retrieved 29 December 2022.
- ^ Broecker F, Moelling K (2019). “What viruses tell us about evolution and immunity: beyond Darwin?”. Annals of the New York Academy of Sciences. 1447 (1): 53–68. Bibcode:2019NYASA1447…53B. doi:10.1111/nyas.14097. PMC 6850104. PMID 31032941.
- ^ Forterre P, Philippe H (June 1999). “The last universal common ancestor (LUCA), simple or complex?”. The Biological Bulletin. 196 (3): 373–75, discussion 375–77. doi:10.2307/1542973. JSTOR 1542973. PMID 11536914.
- ^ Collier p. 8
- ^ Lodish H, Berk A, Zipursky SL, Matsudaira P, Baltimore D, Darnell J (2000). “Viruses: Structure, Function, and Uses”. Molecular Cell Biology (4th ed.). New York: W. H. Freeman.
- ^ Matsuzaki S, Rashel M, Uchiyama J, Sakurai S, Ujihara T, Kuroda M, et al. (October 2005). “Bacteriophage therapy: a revitalized therapy against bacterial infectious diseases”. Journal of Infection and Chemotherapy. 11 (5): 211–19. doi:10.1007/s10156-005-0408-9. PMID 16258815. S2CID 8107934.
- ^ Gleba YY, Giritch A (2011). “Plant Viral Vectors for Protein Expression”. Recent Advances in Plant Virology. Caister Academic Press. ISBN 978-1-904455-75-2.
- ^ Jefferson A, Cadet VE, Hielscher A (September 2015). “The mechanisms of genetically modified vaccinia viruses for the treatment of cancer”. Critical Reviews in Oncology/Hematology. 95 (3): 407–16. doi:10.1016/j.critrevonc.2015.04.001. PMID 25900073.
- ^ Karimkhani C, Gonzalez R, Dellavalle RP (August 2014). “A review of novel therapies for melanoma”. American Journal of Clinical Dermatology. 15 (4): 323–37. doi:10.1007/s40257-014-0083-7. PMID 24928310. S2CID 38864550.
- ^ “FDA approves Amgen’s injected immunotherapy for melanoma”. Reuters. 27 October 2015. Retrieved 24 January 2020.
- ^ Burke J, Nieva J, Borad MJ, Breitbach CJ (August 2015). “Oncolytic viruses: perspectives on clinical development”. Current Opinion in Virology. 13: 55–60. doi:10.1016/j.coviro.2015.03.020. PMID 25989094.
- ^ Dogic Z (2016). “Filamentous Phages As a Model System in Soft Matter Physics”. Frontiers in Microbiology. 7: 1013. doi:10.3389/fmicb.2016.01013. PMC 4927585. PMID 27446051.
- ^ Fischlechner M, Donath E (2007). “Viruses as building blocks for materials and devices”. Angewandte Chemie. 46 (18): 3184–93. doi:10.1002/anie.200603445. PMID 17348058.
- ^ Soto CM, Blum AS, Vora GJ, Lebedev N, Meador CE, Won AP, et al. (April 2006). “Fluorescent signal amplification of carbocyanine dyes using engineered viral nanoparticles”. Journal of the American Chemical Society. 128 (15): 5184–89. doi:10.1021/ja058574x. PMID 16608355.
- ^ Blum AS, Soto CM, Wilson CD, Brower TL, Pollack SK, Schull TL, et al. (July 2005). “An engineered virus as a scaffold for three-dimensional self-assembly on the nanoscale”. Small. 1 (7): 702–06. doi:10.1002/smll.200500021. PMID 17193509.
- ^ Cello J, Paul AV, Wimmer E (August 2002). “Chemical synthesis of poliovirus cDNA: generation of infectious virus in the absence of natural template”. Science. 297 (5583): 1016–18. Bibcode:2002Sci…297.1016C. doi:10.1126/science.1072266. PMID 12114528. S2CID 5810309.
- ^ Coleman JR, Papamichail D, Skiena S, Futcher B, Wimmer E, Mueller S (June 2008). “Virus attenuation by genome-scale changes in codon pair bias”. Science. 320 (5884): 1784–87. Bibcode:2008Sci…320.1784C. doi:10.1126/science.1155761. PMC 2754401. PMID 18583614.
- ^ “NIH viral genome database”. Ncbi.nlm.nih.gov. Retrieved 28 June 2021.
- ^ Zilinskas RA (August 2017). “A brief history of biological weapons programmes and the use of animal pathogens as biological warfare agents”. Revue Scientifique et Technique (International Office of Epizootics). 36 (2): 415–422. doi:10.20506/rst.36.2.2662. PMID 30152475.
- ^ a b c Artenstein AW, Grabenstein JD (October 2008). “Smallpox vaccines for biodefense: need and feasibility”. Expert Review of Vaccines. 7 (8): 1225–37. doi:10.1586/14760584.7.8.1225. PMC 9709930. PMID 18844596. S2CID 33855724.
Bibliography
- Collier L, Balows A, Sussman M (1998). Mahy B, Collier LA (eds.). Topley and Wilson’s Microbiology and Microbial Infections. Virology. Vol. 1 (Ninth ed.). ISBN 0-340-66316-2.
- Dimmock NJ, Easton AJ, Leppard K (2007). Introduction to Modern Virology (Sixth ed.). Blackwell Publishing. ISBN 978-1-4051-3645-7.
- Knipe DM, Howley PM, Griffin DE, Lamb RA, Martin MA, Roizman B, Straus SE (2007). Fields Virology. Lippincott Williams & Wilkins. ISBN 978-0-7817-6060-7.
- Krasner, Robert (2014). The microbial challenge: a public health perspective. Burlington, Mass: Jones & Bartlett Learning. ISBN 978-1-4496-7375-8. OCLC 794228026.</ref>
- Shors T (2017). Understanding Viruses. Jones and Bartlett Publishers. ISBN 978-1-284-02592-7.
External links
Look up genogroup in Wiktionary, the free dictionary.
Look up virus in Wiktionary, the free dictionary.
Термин «вирус», virus, происходит от латинского слова «яд». Вирусы — инфекционные агенты, не существующие самостоятельно. Они встраиваются в клетку и воспроизводят в ней свои гены, переключая ее на производство вирусных белков. Поэтому мы говорим, что вирусы — это неклеточная форма жизни, облигатные паразиты, которые функционируют только внутри организма, а в открытой среде рано или поздно погибают.
В 1892 году отец вирусологии Д. И. Ивановский опубликовал труд, посвященный вирусам. Но и сегодня открытия в этой области продолжаются. Вирусы чрезвычайно малы, их размеры колеблются в интервале от 20 до 300 нм. Они не выращиваются, как бактерии, на искусственных средах, что затрудняет исследования.
Вирусные инфекции делятся на три типа в зависимости от того, как долго вирус находится в клетке, и какие с ней при этом происходят изменения.
1. Литическая
(от греч. лизис — разрушение) инфекция — пораженная клетка гибнет, вирусы покидают ее и «нападают» на другие клетки.
2. Персистентная
(стойкая) инфекция — вирусы оставляют клетку постепенно, клетка живет, но ее функционирование изменено.
3. Латентная (скрытая) инфекция — гены вируса встраиваются в хромосомы хозяйской клетки и передаются дочерним клеткам. Вирусы никуда не мигрируют, могут не активизироваться годами. Однако латентная инфекция может перейти в персистентную или литическую при активизации, размножении и выходе вируса из клетки. В связи с этим вирусы переносят гены от одного организма к другому, копируя части генов разных организмов.
Строение вирусов
1. Вирусы — генетические элементы, одетые в белковую оболочку и переходящие из одной клетки в другую.
2. У вирусов два основных вида симметрии, спиральная и кубическая.
3. Вирион
— отдельная вирусная частица с генетическим материалом в виде ДНК или РНК. Имеет симметричное строение.
4. Капсид — главная белковая оболочка, защищающая вирус от ультрафиолета и действия ферментов нуклеаз, которые разрушают нуклеиновые кислоты. Капсиды имеют многие цепи одного или нескольких типов белков. В состав капсида могут входить не только белки, но и гликопротеины — белки, связанные с углеводами.
5. У некоторых вирусов имеется внешняя оболочка — из липидов, заимствованных из плазмалеммы хозяина. Вирус гриппа имеет наружную оболочку кубического типа симметрии, но при этом спиральный вирион.
На чем основана современная классификация вирусов?
1. Вид и форма нуклеиновой кислоты (РНК — ДНК, линейная — кольцевая, одноцепочечная — двуцепочечная).
2. Тип симметрии — кубическая или спиральная.
3. Присутствие или отсутствие наружной оболочки.
Варианты вирусов по наличию ДНК или РНК
|
Вирусы с ДНК |
Вирусы с РНК |
|||
|
Вирусы с двухцепочечной ДНК. |
Вирусы с одноцепочечной ДНК. |
Вирусы с одноцепочечной РНК. |
Вирусы с двухцепочечной РНК. |
Вирусы с двумя идентичными одноцепочечными РНК. |
|
ДНК могут быть кольцевой или линейной формы (двухцепочечные более разнообразные). |
Такие вирусы могут иметь только кольцевую ДНК. |
Вирус табачной мозаики. Вирус гриппа. |
Вирус гепатита В. |
Некоторые онкогенные (опухолеродные) вирусы (капсид у них кубический). |
|
Бактериофаг Т4, вирус оспы имеют двухцепочечную линейную молекулу ДНК. Кстати, вирус оспы самый крупный и содержит 240 генов! Обезьяний вирус SV40 содержит двухцепочечную кольцевую ДНК. |
Пример — фаг М13, паразитирующий в клетках E. coli. |
Онкогенные ретровирусы имеют внешнюю оболочку. Вирус ВИЧ имеет такое же строение. |
Поговорим о компьютерных вирусах? Нет, не о том, что вчера поймал ваш антивирус. Не о том, что вы скачали под видом инсталлятора очередного Photoshop. Не о rootkit-e, который стоит на вашем сервере, маскируясь под системный процесс. Не о поисковых барах, downloader-ах и другой малвари. Не о коде, который делает плохие вещи от вашего имени и хочет ваши деньги. Нет, всё это коммерция, никакой романтики…
Мы поговорим о компьютерных вирусах, как о коде, который способен порождать собственные копии, изменяясь от поколения к поколению. Которому, как и его биологическим собратьям, необходим файл-носитель, работоспособный, и остающийся работоспособным, чтобы давать жизнь новым поколениям вируса. Которому для размножения необходима благодатная среда, много вкусных исполняемых файлов, а также, много глупых и активных пользователей, чтобы они их запускали. Так что название «вирус» не просто красивый ярлычок для описания вредоносной программы, компьютерный вирус, в его классическом понимании, является сущностью весьма близкой к его биологическому аналогу. Человечество, как это не раз доказывалось, способно создавать весьма изощренные решения, особенно когда дело касается создания чего-нибудь наносящего вред другим людям.
Итак, давным-давно, после того, как DOS пришел к людям, и у каждого программиста появилась своя маленькая вселенная, где адресное пространство было единым, а права на файлы были всегда rwx, появилась мысль о том, может ли программа копировать сама себя. «Конечно, может!», – сказал программист и написал код, который копирует собственный исполняемый файл. Следующая мысль была «а могут ли две программы объединиться в одну?». «Конечно, могут!», – сказал программист и написал первый инфектор. «Только вот зачем?» – подумал он, и это стало началом эпохи компьютерных вирусов. Как оказалось, гадить на компьютере и всячески пытаться избежать обнаружения очень весело, а создание вирусов является очень интересным с точки зрения системного программиста делом. Кроме того, появившиеся на рынке антивирусы предоставляли создателям вирусов серьёзный вызов их профессионализму.
В общем, для статьи вполне достаточно лирики, перейдем к делу. Я хочу рассказать о классическом вирусе, его структуре, основных понятиях, методах детектирования и алгоритмах, которые используются обеими сторонами для победы.
Анатомия вируса
Мы будем говорить о вирусах, живущих в исполняемых файлах форматов PE и ELF, то есть о вирусах, тело которых представляет собой исполняемый код для платформы x86. Кроме того, пусть наш вирус не будет уничтожать исходный файл, полностью сохраняя его работоспособность и корректно инфицируя любой подходящий исполняемый файл. Да, ломать гораздо проще, но мы же договорились говорить о правильных вирусах, да? Чтобы материал был актуальным, я не буду тратить время на рассмотрение инфекторов старого формата COM, хотя именно на нем были обкатаны первые продвинутые техники работы с исполняемым кодом.
Основными частями кода вируса являются infector и payload. Infector – это код, который ищет подходящие для заражения файлы и внедряет в них вирус, стараясь максимально скрыть факт внедрения и при этом не повредить функционалу файла. Payload – это код, который выполняет собственно необходимые вирмейкеру действия, например, рассылает спам, DoS-ит кого-нибудь, или просто оставляет на машине текстовой файлик «Здесь был Виря». Нам совершенно непринципиально, что там внутри payload, главное, что вирмейкер всячески старается скрыть его содержимое.
Начнём со свойств кода вируса. Чтобы код удобней было внедрять, разделять код и данные не хочется, поэтому обычно используется интеграция данных прямо в исполняемый код. Ну, например, так:
jmp message
the_back:
mov eax, 0x4
mov ebx, 0x1
pop ecx ; со стека будет взят адрес «Hello, World»
mov edx, 0xF
int 0x80
...
message:
call the_back ; после исполнения на стеке будет лежать адрес «возврата», т.е. адрес «Hello, Worldn»
db "Hello, World!", 0Dh, 0Ah
Или так:
push 0x68732f2f ; “hs//”
push 0x6e69622f ; “nib/”
mov ebx, esp ; в ESP теперь адрес строки «/bin/sh»
mov al, 11
int 0x80
Все эти варианты кода при определенных условиях можно просто скопировать в память и сделать JMP на первую инструкцию. Правильно написав такой код, позаботившись о правильных смещениях, системных вызовах, чистоте стека до и после исполнения, и т.д., его можно внедрять внутрь буфера с чужим кодом.
Предположим, вирмейкер имеет возможность написать код вируса в таком стиле, и теперь ему надо внедрить его в существующий исполняемый файл. Ему необходимо позаботиться о двух вещах:
- Куда положить вирус? Необходимо найти достаточно места, чтобы вирус туда поместился, записать его туда, по возможности не разломав файл и так, чтобы в области, в которой вирус окажется, было разрешено исполнение кода.
- Как передать управление на вирус? Просто положить вирус в файл недостаточно, надо еще совершить переход на его тело, а после завершения его работы вернуть управление программе-жертве. Или в другом порядке, но, в любом случае, мы же договорились не ломать ничего, да?
Итак, разберемся с внедрением в файл. Современные исполняемые форматы для платформы x86 в Windows и Linux – это PE (Portable Executable) и ELF (Executable and Linkable Format). Вы без проблем найдёте их спецификации в системной документации, и если будете заниматься вопросами защиты исполняемого кода, то точно не пропустите. Исполняемые форматы и системный загрузчик (код операционной системы, который занимается запуском исполняемого файла) являются одним из «слонов», на которых стоит операционная система. Процедура запуска .exe файла является очень сложным алгоритмически процессом с кучей нюансов, и рассказывать об этом можно в десятке статей, которые вы обязательно найдете сами, если тема вас заинтересует. Я ограничусь простым рассмотрением, достаточным для базового понимания процесса запуска. Чтобы в меня не кидались помидорами, далее под компилятором я буду иметь в виду весь комплекс программ, превращающий исходный код в готовый исполняемый файл, то есть, фактически, компилятор + линкер.
Исполняемый файл (PE или ELF) состоит из заголовка и набора секций. Секции – это выровненные (см. ниже) буферы с кодом или данными. При запуске файла секции копируются в память и под них выделяется память, причем совсем необязательно того объёма, который они занимали на диске. Заголовок содержит разметку секций, и сообщает загрузчику, как расположены секции в файле, когда он лежит на диске, и каким образом необходимо расположить их в памяти перед тем, как передать управление коду внутри файла. Для нас интересны три ключевых параметра для каждой секции, это psize, vsize, и flags. Psize (physical size) представляет собой размер секции на диске. Vsize (virtual size) – размер секции в памяти после загрузки файла. Flags – атрибуты секции (rwx). Psize и Vsize могут существенно различаться, например, если программист объявил в программе массив в миллион элементов, но собирается заполнять его в процессе исполнения, компилятор не увеличит psize (на диске содержимое массива хранить до запуска не нужно), а вот vsize увеличит на миллион чего-то там (в runtime для массива должно быть выделено достаточно памяти).
Флаги (атрибуты доступа) будут присвоены страницам памяти, в которые секция будет отображена. Например, секция с исполняемым кодом будет иметь атрибуты r_x (read, execute), а секция данных атрибуты rw_ (read,write). Процессор, попытавшись исполнить код на странице без флага исполнения, сгенерирует исключение, то же касается попытки записи на страницу без атрибута w, поэтому, размещая код вируса, вирмейкер должен учитывать атрибуты страниц памяти, в которых будет располагаться код вируса. Стандартные секции неинициализированных данных (например, область стека программы) до недавнего времени имели атрибуты rwx (read, write, execute), что позволяло копировать код прямо в стек и исполнять его там. Сейчас это считается немодным и небезопасным, и в последних операционных системах область стека предназначена только для данных. Разумеется, программа может и сама изменить атрибуты страницы памяти в runtime, но это усложняет реализацию.
Также, в заголовке лежит Entry Point — адрес первой инструкции, с которой начинается исполнение файла.
Необходимо упомянуть и о таком важном для вирмейкеров свойстве исполняемых файлов, как выравнивание. Для того чтобы файл оптимально читался с диска и отображался в память, секции в исполняемых файлах выровнены по границам, кратным степеням двойки, а свободное место, оставшееся от выравнивания (padding) заполнено чем-нибудь на усмотрение компилятора. Например, логично выравнивать секции по размеру страницы памяти – тогда ее удобно целиком копировать в память и назначать атрибуты. Даже вспоминать не буду про все эти выравнивания, везде, где лежит мало-мальски стандартный кусок данных или кода, его выравнивают (любой программист знает, что в километре ровно 1024 метра). Ну а описание стандартов Portable Executable (PE) и Executable Linux Format (ELF) для работающего с методами защиты исполняемого кода – это настольные книжки.
Так как адреса внутри всех этих секций связаны, просто шлепнуть кусок кода в середину секции, «перевязав» его JMP-ами не получится, исходный файл сломается. Поэтому популярными местами для внедрения кода вируса являются:
- основная кодовая секция (перезапись вирусом начала исполняемого кода начиная прямо с Entry Point).
- padding между окончанием заголовка и первой секцией. Там ничего нет и вполне можно уместить там небольшой вирус (либо его загрузчик) не сломав файл.
- новая секция, которую можно дописать в заголовок и разместить в файле после всех остальных. В этом случае никакие внутренние смещения не поломаются, и с местом проблем тоже нет. Правда последняя секция в файле, в которой разрешено исполнение, конечно же, обратит на себя внимание эвристика.
- padding между окончанием содержимого секции и ее выровненным концом. Это намного сложнее, так как сначала надо этот самый «конец» найти, и не факт, что нам повезет и места будет достаточно. Но для некоторых компиляторов это место можно обнаружить просто по характерным байтам
Есть способы и похитрее, некоторые я опишу во второй статье.
Теперь о передаче управления. Чтобы вирус отработал, его код должен каким-то способом получить управление. Самый очевидный способ: сначала управление получает вирус, а потом, после того, как он отработает – программа-хост. Это самый простой способ, но также имеют право на жизнь и варианты, когда вирус получает управление, например, после завершения работы хоста, или в середине исполнения, «замещая» исполнение какой-нибудь функции. Приведем несколько техник передачи управления (термин Entry Point или EP, используемый далее, – это точка входа, то есть адрес, на который системный загрузчик передаст управление после того, как подготовит исполняемый файл к запуску):
- JMP на тело вируса замещает первые байты, находящиеся в Entry Point файла. Затёртые байты вирус сохраняет в своём теле, и, по окончании собственной работы, восстанавливает их и передает управление на начало восстановленного буфера.
- Способ, похожий на предыдущий, но вместо байтов вирус сохраняет несколько полных машинных инструкций в Entry Point, тогда он может, ничего не восстанавливая (проследив только за корректной очисткой стека), выполнить их после окончания собственной работы и передать управление на адрес инструкции, следующей за «сворованными».
- Как и в случае с внедрением, есть способы и похитрее, но мы их тоже рассмотрим ниже, или отложим до следующей статьи.
Всё это – способы сделать корректную вставку буфера с кодом в некоторый исполняемый файл. При этом п.2 и п.3. подразумевают функционал, позволяющий понять, какие байты являются инструкциями, и где находятся границы между инструкциями. Ведь мы не можем «разорвать» инструкцию пополам, в этом случае все сломается. Таким образом, мы плавно переходим к рассмотрению дизассемблеров в вирусах. Понятие принципа работы дизассемблеров нам понадобится для рассмотрения всех нормальных техник работы с исполняемым кодом, поэтому ничего страшного, если я немного опишу его сейчас.
Если мы внедрим свой код в позицию точно между инструкциями, то сможем сохранить контекст (стек, флаги) и, выполнив код вируса, восстановить все обратно, вернув управление программе-хосту. Конечно, с этим тоже могут быть проблемы, если используются средства контроля целостности кода, антиотладка и т.п., но об этом тоже во второй статье. Для поиска такой позиции нам необходимо вот что:
- поставить указатель точно на начало какой-нибудь инструкции (просто так взять рандомное место в исполняемой секции и начать дизассемблирование с него нельзя, один и тот же байт может быть и опкодом инструкции, и данными)
- определить длину инструкции (для архитектуры x86 инструкции имеют разные длины)
- переместить указатель вперед на эту длину. Мы окажемся на начале следующей инструкции.
- повторять, пока не решим остановиться
Это минимальный функционал, необходимый для того, чтобы не попасть в середину инструкции, а функция, которая принимает указатель на байтовую строку, а в ответ отдает длину инструкции, называется дизассемблером длин. Например, алгоритм заражения может быть таким:
- Выбираем вкусный исполняемый файл (достаточно толстый, чтобы в него поместилось тело вируса, с нужным распределением секций и т.п.).
- Читаем свой код (код тела вируса).
- Берем несколько первых инструкций из файла-жертвы.
- Дописываем их к коду вируса (сохраняем информацию, необходимую для восстановления работоспособности).
- Дописываем к коду вируса переход на инструкцию, продолжающую исполнение кода-жертвы. Таким образом, после исполнения собственного кода вирус корректно исполнит пролог кода-жертвы.
- Создаем новую секцию, записываем туда код вируса и правим заголовок.
- На место этих первых инструкций кладем переход на код вируса.
Это вариант вполне себе корректного вируса, который может внедриться в исполняемый файл, ничего не сломать, скрыто выполнить свой код и вернуть исполнение программе-хосту. Теперь, давайте его ловить.
Анатомия детектора
Вдруг, откуда ни возьмись, появляется рыцарь на белом компе, в левой руке у него отладчик, а в правой – дизассемблер, программист антивирусной компании. Откуда он там взялся? Вы, конечно, догадались. С большой долей вероятности, он появился там из «смежной области». Антивирусная область в плане программирования весьма уважаема теми, кто в теме, ибо возиться этим ребятам приходится с весьма изощренными алгоритмами, причем в довольно стеснённых условиях. Сами посудите: у вас на входе сотня тысяч экземпляров всякой заразы и исполняемый файл, работать вы должны практически в реальном времени, а цена ошибки весьма высока.
Для антивируса, как и для любого конечного автомата, принимающего бинарное решение «да/нет» (инфицирован/здоров), существует два типа ошибок – false positive и false negative (ошибочно признал файл заразным, ошибочно пропустил зараженный). Понятно, что общее число ошибок надо снижать в любом раскладе, но false negative для антивируса куда более неприятна, чем false positive. «После скачивания торрента, перед установкой игры отключите антивирус» — знакомо? Это и есть «false positive» – crack.exe, записывающий что-то в исполняемый .exe файл для достаточно умного эвристического анализатора (см. ниже), выглядит как вирус. Как говорится: «лучше перебдеть, чем недобдеть».
Думаю, не надо описывать вам компоненты современного антивируса, все они крутятся вокруг одного функционала – антивирусного детектора. Монитор, проверяющий файлы на лету, сканирование дисков, проверка почтовых вложений, карантин и запоминание уже проверенных файлов – все это обвязка основного детектирующего ядра. Второй ключевой компонент антивируса – пополняемые базы признаков, без которых поддержание антивируса в актуальном состоянии невозможно. Третий, достаточно важный, но заслуживающий отдельного цикла статей компонент – мониторинг системы на предмет подозрительной деятельности.
Итак (рассматриваем классические вирусы), на входе имеем исполняемый файл и один из сотни тысяч потенциальных вирусов в нем. Давайте детектировать. Пусть это кусок исполняемого кода вируса:
XX XX XX XX XX XX ; начало вируса длиной N байт . . .
68 2F 2F 73 68 push 0x68732f2f ; “hs//”
68 2F 62 69 6E push 0x6e69622f ; “nib/”
8B DC mov ebx, esp ; в ESP теперь адрес строки «/bin/sh»
B0 11 mov al, 11
CD 80 int 0x80
XX XX XX XX ; конец вируса длиной M байт . . .
Сразу хочется просто взять пачку опкодов (68 2F 2F 73 68 68 2F 62 69 6E 8B DC B0 11 CD 80) и поискать эту байтовую строку в файле. Если нашли – попался, гад. Но, увы, оказывается эта же пачка байт встречается и в других файлах (ну мало ли кто вызывает командный интерпретатор), да еще и таких строк для поиска «стотыщ», если искать каждую, то никакая оптимизация не поможет. Единственный, быстрый и правильный способ проверить наличие такой строки в файле – это проверить ее существование по ФИКСИРОВАННОМУ смещению. Откуда его взять?
Вспоминаем «смежную область» — особенно места про то, куда вирус себя кладет и как передает себе управление:
- вирус внедряется в padding между заголовком и началом первой секции. В этом случае можно проверить существование этой байт-строки по смещению
«длина заголовка» + N (где N – число байт от начала вируса до байт-строки) - вирус лежит в новой, отдельной секции. В этом случаем можно проверить существование байт-строки от начала всех секций с кодом
- вирус внедрился в padding между концом кода и концом кодовой секции. Можно использовать отрицательное смещение от конца секции, типа «конец кодовой секции» — М (где M — число байт от конца байт-строки до конца кода вируса) – «длина байт-строки»
Теперь оттуда же про передачу управления:
- вирус записал свои инструкции прямо поверх инструкций в Entry Point. В этом случае ищем байт строку просто по смещению «Entry Point» + N(где N – число байт от начала вируса до байт-строки)
- вирус записал в Entry Point JMP на свое тело. В этом случае надо сначала вычислить куда смотрит этот JMP, а потом искать байт-строку по смещению «адрес перехода JMP» + N(где N – число байт от начала вируса до байт-строки)
Что-то я устал писать «байт-строка», она переменной длины, хранить ее в базе неудобно, и совершенно необязательно, поэтому вместо байт-строки мы будем использовать её длину плюс CRC32 от неё. Такая запись очень короткая и сравнение работает быстро, так как CRC32 алгоритм не из медленных. Гнаться за устойчивостью к коллизиям контрольных сумм смысла нет, так как вероятность коллизии по фиксированным смещениям мизерная. Кроме того, даже в случае коллизии ошибка будет типа «false positive», что не так уж и страшно. Обобщаем все вышеописанное, вот примерная структура записи в нашей антивирусной базе:
- ID вируса
- флаги, указывающие откуда считать смещение (от EP, от конца заголовка, от конца первой секции, от начала всех секций, от адреса перехода инструкции JMP в EP и т.п.)
- смещение (offset)
- длина сигнатуры (Lsig)
- CRC32 сигнатуры (CRCsig)
Оптимизируем вход (оставляем только сигнатуры, которые «влазят» в данный файл, сразу из заголовка подготавливаем набор необходимых смещений) и далее:
{ # для всех подходящих записей
- на основании флагов вычисляем базовое смещение в файле (начало кодовой секции, entry point и т.п.)
- прибавляем к нему offset
- читаем Lsig байт
- считаем от них CRC32
- если совпало – мы поймали вирус
}
Ура, вот наш первый антивирус. Он достаточно крут, так как при помощи достаточно полной базы сигнатур, нормально подобранных флагов и хорошей оптимизации этот детектор способен очень быстро ловить 95% всяких зараз (подавляющее большинство современного malware это просто исполняемые файлы, без всякой способности к мутации). Далее начинается игра «кто быстрее обновит базы сигнатур» и «кому раньше пришлют новый экземпляр какой-нибудь гадости».
Сбор и каталогизация этой «гадости» является задачей весьма нетривиальной, но совершенно необходимой для качественного тестирования детектора. Сбор эталонной базы исполняемых файлов задача непростая: попробуйте найти все экземпляры зараженных файлов (для сложных случаев в нескольких экземплярах), каталогизировать их, перемешать с «чистыми» файлами и регулярно гонять по ним детектор с целью выявления ошибок детектирования. Такая база собирается годами, и является очень ценным активом антивирусных компаний. Возможно, я ошибаюсь, и её реально достать (всякие сервисы online-проверок на вирусы вполне в состоянии предоставить некоторый её аналог), но, когда я занимался этим вопросом, ничего похожего достать было нельзя (по крайней мере, под Linux).
Эвристический анализатор
Какое страшное слово – «эвристический анализатор», сейчас его и не увидишь в интерфейсах антивирусов (наверное, пугает пользователей). Это одна из самых интересных частей антивируса, так как в нее пихают все, что не укладывается ни в один из движков (ни сигнатурный, ни в эмулятор), и похож на доктора, который видит, что пациент кашляет и чихает, но определить конкретную болезнь не может. Это код, который проверяет файл на некоторые характерные признаки заражения. Примеры таких признаков:
- некорректный (испорченный вирусом, но работоспособный) заголовок файла
- JMP прямо в точке входа
- «rwx» на секции кода
Ну, и так далее. Помимо указания факта заражения, эвристик может помочь принять решение – запускать ли более «тяжелый» анализ файла? Каждый признак имеет разный вес, от «подозрительный какой-то» до «не знаю чем, но файл заражен точно». Именно эти признаки дают большинство ошибок «false positive». Не забудем также о том, что именно эвристик может предоставить антивирусной компании экземпляры потенциальных вирусов. Сработал эвристик, но ничего конкретного не было найдено? Значит файл точно является кандидатом на отправку в антивирусную компанию.
Межвидовое взаимодействие и эволюция
Как мы увидели, для быстрого и точного сравнения детектору необходимы сами байты сигнатуры и ее смещение. Или, другим языком, содержимое кода и адрес его расположения в файле-хосте. Поэтому понятно, как развивались идеи сокрытия исполняемого кода вирусов – по двум направлениям:
- сокрытие кода самого вируса;
- сокрытие его точки входа.
Сокрытие кода вируса в результате вылилось в появление полиморфных движков. То есть движков, позволяющих вирусу изменять свой код в каждом новом поколении. В каждом новом зараженном файле тело вируса мутирует, стараясь затруднить обнаружение. Таким образом, затрудняется создание содержимого сигнатуры.
Сокрытие точки входа (Entry Point Obscuring) в результате послужило толчком для появления в вирусных движках автоматических дизассемблеров для определения, как минимум, инструкций перехода. Вирус старается скрыть место, с которого происходит переход на его код, используя из файла то, что в итоге приводит к переходу: JMP, CALL, RET всякие, таблицы адресов и т.п. Таким образом, вирус затрудняет указание смещения сигнатуры.
Гораздо более подробно некоторые алгоритмы таких движков и детектора мы посмотрим во второй статье, которую я планирую написать в ближайшее время.
Параллельно с развитием вирусных движков и противостоящих им детекторов активно развивались коммерческие защиты исполняемых файлов. Появилось огромное количество небольших коммерческих программ, и разработчикам нужны были движки, позволяющие взять EXE-файл и «завернуть» его в некоторый «конверт», который умеет защищенным образом генерировать валидный серийный номер. А кто у нас умеет скрывать исполняемый код, и внедрять его в исполняемые файлы без потери работоспособности? Правильно, те самые разработчики из «смежной области». Поэтому написание хорошего полиморфного вируса и навесной защиты исполняемого файла – это очень похожие задачи, с использованием одних и тех же алгоритмов и инструментов. Так же схожи и процесс анализа вирусов и создания сигнатур и взлом коммерческого ПО. В обоих случаях надо добраться до истинного кода и либо создать сигнатуру, либо достать из него алгоритм генерации серийного номера.
В интернетах существуют несколько страниц по теме «классификация компьютерных вирусов». Но мы же договорились, вирус – это то, что умеет само себя воспроизводить в системе, и чему необходим файл-носитель. Поэтому всякие трояны-руткиты-malware – это не вирусы, а тип payload-кода, который вирус может таскать на себе. Для описываемых в статье технологий классификация компьютерных вирусов может быть только одна: полиморфные и неполиморфные вирусы. То есть меняющиеся от поколения к поколению, либо нет.
Рассмотренный в статье детектор легко детектирует неполиморфные (мономорфными их назвать, что ли) вирусы. Ну а переход к полиморфным вирусам является отличным поводом, наконец, завершить эту статью, пообещав вернуться к более интересным методам сокрытия исполняемого кода во второй части.