ДНК и гены
ДНК ПРОКАРИОТ И ЭУКАРИОТ
 |
|
Справа крупнейшая спираль ДНК человека, выстроенная из людей на пляже в Варне (Болгария), вошедшая в книгу рекордов Гиннесса 23 апреля 2016 года
Дезоксирибонуклеиновая кислота. Общие сведения

- Дезоксирибонуклеиновая кислота
- Строение нуклеиновых кислот
- Репликация
- Строение РНК
- Транскрипция
- Трансляция
- Генетический код
- Геном: гены и хромосомы
- Прокариоты
- Эукариоты
- Строение генов
- Строение генов прокариот
- Строение генов эукариот
- Сравнение строения генов
- Мутации и мутагенез
- Генные мутации
- Хромосомные мутации
- Геномные мутации
- Видео по теме ДНК
- Дополнительный материал
ДНК (дезоксирибонуклеиновая кислота) – своеобразный чертеж жизни, сложный код, в котором заключены данные о наследственной информации. Эта сложная макромолекула способна хранить и передавать наследственную генетическую информацию из поколения в поколение. ДНК определяет такие свойства любого живого организма как наследственность и изменчивость. Закодированная в ней информация задает всю программу развития любого живого организма. Генетически заложенные факторы предопределяют весь ход жизни как человека, так и любого др. организхма. Искусственное или естественное воздействие внешней среды способны лишь в незначительной степени повлиять на общую выраженность отдельных генетических признаков или сказаться на развитии запрограммированных процессов.
Дезоксирибонуклеи́новая кислота (ДНК) — макромолекула (одна из трёх основных, две другие — РНК и белки), обеспечивающая хранение, передачу из поколения в поколение и реализацию генетической программы развития и функционирования живых организмов. ДНК содержит информацию о структуре различных видов РНК и белков.
В клетках эукариот (животных, растений и грибов) ДНК находится в ядре клетки в составе хромосом, а также в некоторых клеточных органоидах (митохондриях и пластидах). В клетках прокариотических организмов (бактерий и архей) кольцевая или линейная молекула ДНК, так называемый нуклеоид, прикреплена изнутри к клеточной мембране. У них и у низших эукариот (например, дрожжей) встречаются также небольшие автономные, преимущественно кольцевые молекулы ДНК, называемые плазмидами.
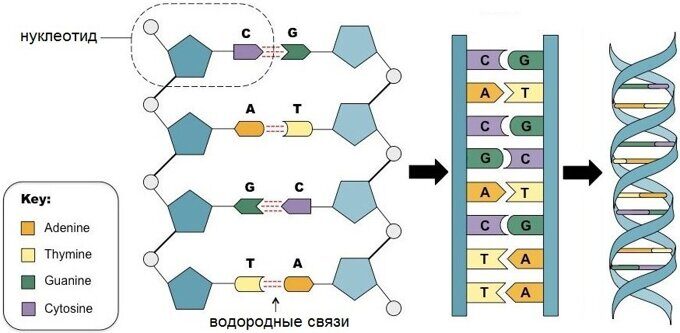
С химической точки зрения ДНК — это длинная полимерная молекула, состоящая из повторяющихся блоков — нуклеотидов. Каждый нуклеотид состоит из азотистого основания, сахара (дезоксирибозы) и фосфатной группы. Связи между нуклеотидами в цепи образуются за счёт дезоксирибозы (С) и фосфатной (Ф) группы (фосфодиэфирные связи).
Рис. 2. Нуклертид состоит из азотистого основания, сахара (дезоксирибозы) и фосфатной группы
В подавляющем большинстве случаев (кроме некоторых вирусов, содержащих одноцепочечную ДНК) макромолекула ДНК состоит из двух цепей, ориентированных азотистыми основаниями друг к другу. Эта двухцепочечная молекула закручена по винтовой линии.
В ДНК встречается четыре вида азотистых оснований (аденин, гуанин, тимин и цитозин). Азотистые основания одной из цепей соединены с азотистыми основаниями другой цепи водородными связями согласно принципу комплементарности: аденин соединяется только с тимином (А-Т), гуанин — только с цитозином (Г-Ц). Именно эти пары и составляют «перекладины» винтовой “лестницы” ДНК (см.: рис. 2, 3 и 4).
Рис. 2. Азотистые основания
Последовательность нуклеотидов позволяет «кодировать» информацию о различных типах РНК, наиболее важными из которых являются информационные, или матричные (мРНК), рибосомальные (рРНК) и транспортные (тРНК). Все эти типы РНК синтезируются на матрице ДНК за счёт копирования последовательности ДНК в последовательность РНК, синтезируемой в процессе транскрипции, и принимают участие в биосинтезе белков (процессе трансляции). Помимо кодирующих последовательностей, ДНК клеток содержит последовательности, выполняющие регуляторные и структурные функции.

Рис. 3. Репликация ДНК
Расположение базовых комбинаций химических соединений ДНК и количественные соотношения между этими комбинациями обеспечивают кодирование наследственной информации.
Образование новой ДНК (репликация)
- Процесс репликации: раскручивание двойной спирали ДНК — синтез комплементарных цепей ДНК-полимеразой — образование двух молекул ДНК из одной.
- Двойная спираль «расстегивается» на две ветви, когда ферменты разрушают связь между базовыми парами химических соединений.
- Каждая ветвь является элементом новой ДНК. Новые базовые пары соединяются в той же последовательности, что и в родительской ветви.
По завершении дупликации образуются две самостоятельные спирали, созданные из химических соединений родительской ДНК и имеющие с ней одинаковый генетический код. Таким путем ДНК способна перерывать информацию от клетки к клетке.
Более подробная информация:
СТРОЕНИЕ НУКЛЕИНОВЫХ КИСЛОТ

Рис. 4 . Азотистые основания: аденин, гуанин, цитозин, тимин
Дезоксирибонуклеиновая кислота (ДНК) относится к нуклеиновым кислотам. Нуклеиновые кислоты – это класс нерегулярных биополимеров, мономерами которых являются нуклеотиды.
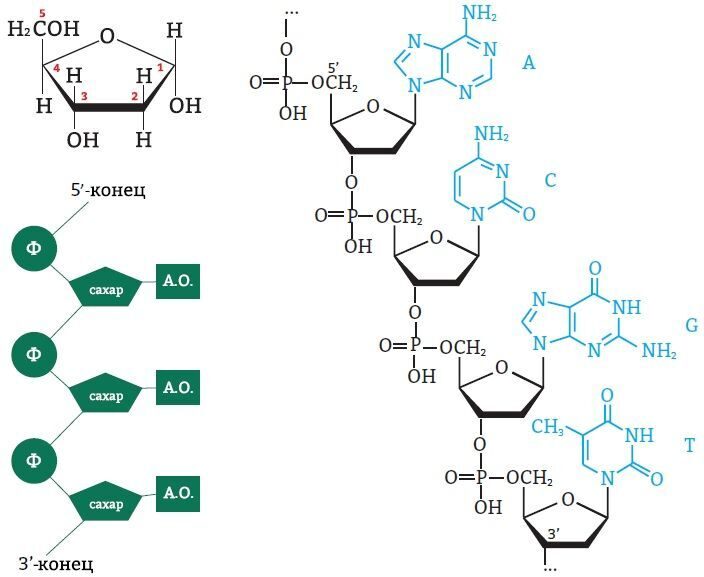
НУКЛЕОТИДЫ состоят из азотистого основания, соединенного с пятиуглеродным углеводом (пентозой) – дезоксирибозой (в случае ДНК) или рибозой (в случае РНК), который соединяется с остатком фосфорной кислоты (H2PO3–).
Азотистые основания бывают двух типов: пиримидиновые основания – урацил (только в РНК), цитозин и тимин, пуриновые основания – аденин и гуанин.
Рис. 5. Структура нуклеотидов (слева), расположение нуклеотида в ДНК (снизу) и типы азотистых оснований (справа): пиримидиновые и пуриновые
Атомы углерода в молекуле пентозы нумеруются числами от 1 до 5. Фосфат соединяется с третьим и пятым атомами углерода. Так нуклеинотиды соединяются в цепь нуклеиновой кислоты. Таким образом, мы можем выделить 3’ и 5’-концы цепи ДНК:
Рис. 6. Выделение 3’ и 5’-концов цепи ДНК
Две цепи ДНК образуют двойную спираль. Эти цепи в спирали сориентированы в противоположных направлениях. В разных цепях ДНК азотистые основания соединены между собой с помощью водородных связей. Аденин всегда соединяется с тимином, а цитозин – с гуанином. Это называется правилом комплементарности (см. принцип комплементарности).
Правило комплементарности:
Например, если нам дана цепь ДНК, имеющая последовательность
3’– ATGTCCTAGCTGCTCG – 5’,
то вторая ей цепь будет комплементарна и направлена в противоположном направлении – от 5’-конца к 3’-концу:
5’– TACAGGATCGACGAGC– 3’.

Рис. 7. Направленность цепей молекулы ДНК и соединение азотистых оснований с помощью водородных связей
РЕПЛИКАЦИЯ ДНК
Репликация ДНК – это процесс удвоения молекулы ДНК путем матричного синтеза. В большинстве случаев естественной репликации ДНК праймером для синтеза ДНК является короткий фрагмент РНК (создаваемый заново). Такой рибонуклеотидный праймер создается ферментом праймазой (ДНК-праймаза у прокариот, ДНК-полимераза у эукариот), и впоследствии заменяется дезоксирибонуклеотидами полимеразой, выполняющей в норме функции репарации (исправления химических повреждений и разрывов в молекле ДНК).
Репликация происходит по полуконсервативному механизму. Это значит, что двойная спираль ДНК расплетается и на каждой из ее цепей по принципу комплементарности достраивается новая цепь. Дочерняя молекула ДНК, таким образом, содержит в себе одну цепь от материнской молекулы и одну вновь синтезированную. Репликация происходит в направлении от 3’ к 5’ концу материнской цепи.

Рис. 8. Репликация (удвоение) молекулы ДНК
ДНК-синтез – это не такой сложный процесс, как может показаться на первый взгляд. Если подумать, то для начала нужно разобраться, что же такое синтез. Это процесс объединения чего-либо в одно целое. Образование новой молекулы ДНК проходит в несколько этапов:
1) ДНК-топоизомераза, располагаясь перед вилкой репликации, разрезает ДНК для того, чтобы облегчить ее расплетание и раскручивание.
2) ДНК-хеликаза вслед за топоизомеразой влияет на процесс «расплетения» спирали ДНК.
3) ДНК-связывающие белки осуществляют связывание нитей ДНК, а также проводят их стабилизацию, не допуская их прилипания друг к другу.
4) ДНК-полимераза δ (дельта), согласовано со скоростью движения репликативной вилки, осуществляет синтез ведущей цепи дочерней ДНК в направлении 5’→3′ на матрице материнской нити ДНК по направлению от ее 3′-конца к 5′-концу (скорость до 100 пар нуклеотидов в секунду). Этим события на данной материнской нити ДНК ограничиваются.
Рис. 9. Схематическое изображение процесса репликации ДНК: (1) Отстающая цепь (запаздывающая нить), (2) Ведущая цепь (лидирующая нить), (3) ДНК-полимераза α (Polα), (4) ДНК-лигаза, (5) РНК-праймер, (6) Праймаза, (7) Фрагмент Оказаки, (8) ДНК-полимераза δ (Polδ), (9) Хеликаза, (10) Однонитевые ДНК-связывающие белки, (11) Топоизомераза.
Далее описан синтез отстающей цепи дочерней ДНК (см. Схему репликативной вилки и функции ферментов репликации)
Нагляднее о репликации ДНК см. видео →
5) Непосредственно сразу после расплетания и стабилизации другой нити материнской молекулы к ней присоединяется ДНК-полимераза α (альфа) и в направлении 5’→3′ синтезирует праймер (РНК-затравку) – последовательность РНК на матрице ДНК длиной от 10 до 200 нуклеотидов. После этого фермент удаляется с нити ДНК.
Вместо ДНК-полимеразы α к 3′-концу праймера присоединяется ДНК-полимераза ε.
6) ДНК-полимераза ε (эпсилон) как бы продолжает удлинять праймер, но в качестве субстрата встраивает дезоксирибонуклеотиды (в количестве 150-200 нуклеотидов). В результате образуется цельная нить из двух частей – РНК (т.е. праймер) и ДНК. ДНК-полимераза ε работает до тех пор, пока не встретит праймер предыдущего фрагмента Оказаки (синтезированный чуть ранее). После этого данный фермент удаляется с цепи.
7) ДНК-полимераза β (бета) встает вместо ДНК-полимеразы ε, движется в том же направлении (5’→3′) и удаляет рибонуклеотиды праймера, одновременно встраивая дезоксирибонуклеотиды на их место. Фермент работает до полного удаления праймера, т.е. пока на его пути не встанет дезоксирибонуклеотид (еще более ранее синтезированный ДНК-полимеразой ε). Связать результат свой работы и впереди стоящую ДНК фермент не в состоянии, поэтому он сходит с цепи.
В результате на матрице материнской нити “лежит” фрагмент дочерней ДНК. Он называется фрагмент Оказаки.
8) ДНК-лигаза производит сшивку двух соседних фрагментов Оказаки, т.е. 5′-конца отрезка, синтезированного ДНК-полимеразой ε, и 3′-конца цепи, встроенного ДНК-полимеразой β.
СТРОЕНИЕ РНК
Рибонуклеиновая кислота (РНК) — одна из трёх основных макромолекул (две другие — ДНК и белки), которые содержатся в клетках всех живых организмов.
Так же, как ДНК, РНК состоит из длинной цепи, в которой каждое звено называется нуклеотидом. Каждый нуклеотид состоит из азотистого основания, сахара рибозы и фосфатной группы. Однако в отличие от ДНК, РНК обычно имеет не две цепи, а одну. Пентоза в РНК представлена рибозой, а не дезоксирибозой (у рибозы присутствует дополнительная гидроксильная группа на втором атоме углевода). Наконец, ДНК отличается от РНК по составу азотистых оснований: вместо тимина (Т) в РНК представлен урацил (U), который также комплементарен аденину.
Последовательность нуклеотидов позволяет РНК кодировать генетическую информацию. Все клеточные организмы используют РНК (мРНК) для программирования синтеза белков.
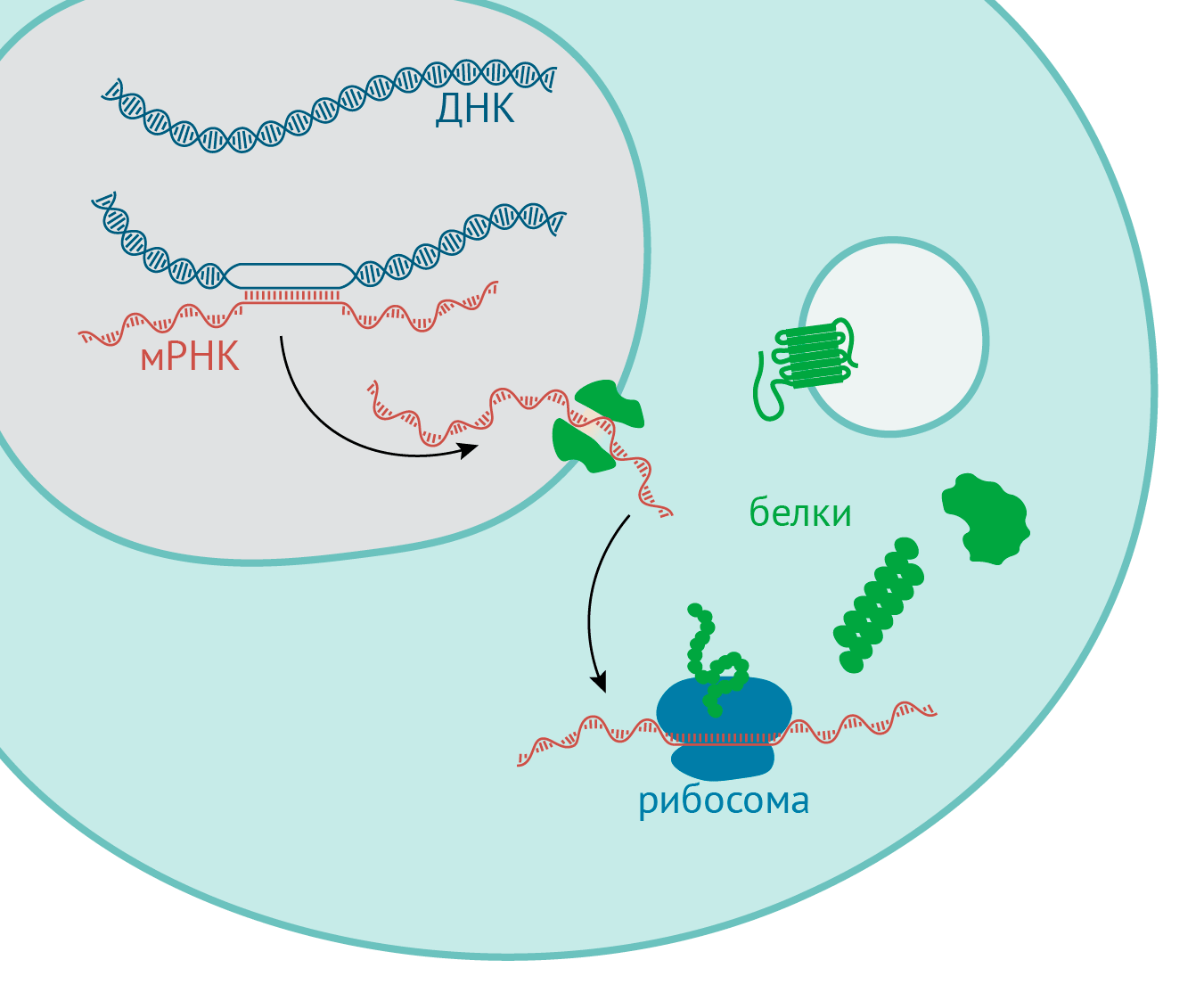
Клеточные РНК образуются в ходе процесса, называемого транскрипцией, то есть синтеза РНК на матрице ДНК, осуществляемого специальными ферментами – РНК-полимеразами.
Затем матричные РНК (мРНК) принимают участие в процессе, называемом трансляцией, т.е. синтеза белка на матрице мРНК при участии рибосом. Другие РНК после транскрипции подвергаются химическим модификациям, и после образования вторичной и третичной структур выполняют функции, зависящие от типа РНК.

Рис. 10. Отличие ДНК от РНК по азотистому основанию: вместо тимина (Т) в РНК представлен урацил (U), который также комплементарен аденину.
ТРАНСКРИПЦИЯ
Транскрипция – это процесс синтеза РНК на матрице ДНК. ДНК раскручивается на одном из участков. На одной из цепей содержится информация, которую необходимо скопировать на молекулу РНК – эта цепь называется кодирующей. Вторая цепь ДНК, комплементарная кодирующей, называется матричной. В процессе транскрипции на матричной цепи в направлении 3’ – 5’ (по цепи ДНК) синтезируется комплементарная ей цепь РНК. Таким образом, создается РНК-копия кодирующей цепи.

Рис. 11. Схематическое изображение транскрипции
Например, если нам дана последовательность кодирующей цепи
3’– ATGTCCTAGCTGCTCG – 5’,
то, по правилу комплементарности, матричная цепь будет нести последовательность
5’– TACAGGATCGACGAGC– 3’,
а синтезируемая с нее РНК – последовательность
3’– AUGUCCUAGCUGCUCG – 5’.
ТРАНСЛЯЦИЯ
Рассмотрим механизм синтеза белка на матрице РНК, а также генетический код и его свойства. Также для наглядности по ниже приведенной ссылке рекомендуем посмотреть небольшое видео о процессах транскрипции и трансляции, происходящих в живой клетке:
|
|
В представленном видоролике (кнопка-ссылка слева) показан процесс образования белка из аминокислот. Наглядно (в анимированном варианте) продемонстрированы процессы транскрипции и трансляции. Биосинтез белка на рибосоме также кратко описан в разделе Аминокислоты белков. Более подробное видео о геноме, ДНК и ее структуре, а также процессах кодировки представленно ниже на данной странице: Видео по теме ДНК |

Рис. 12. Процесс синтеза белка: ДНК кодирует РНК, РНК кодирует белок
Трансляция – это процесс, посредством которого генетическая информация преобразуется в белки, рабочие лошадки клетки. Небольшие молекулы, называемые переносными РНК («тРНК»), играют решающую роль в трансляции; они являются молекулами-адаптерами, которые соответствуют кодонам (строительным блокам генетической информации) с аминокислотами (строительными блоками белков). Организмы несут множество типов тРНК, каждая из которых кодируется одним или несколькими генами («набор генов тРНК»).
Вообще говоря, функция набора генов тРНК – переводить 61 тип кодонов в 20 различных типов аминокислот – сохраняется в разных организмах. Тем не менее, состав набора генов тРНК может значительно варьировать между организмами.
ГЕНЕТИЧЕСКИЙ КОД
Генетический код – способ кодирования аминокислотной последовательности белков с помощью последовательности нуклеотидов. Каждая аминокислота кодируется последовательностью из трех нуклеотидов – кодоном или триплетом.
Генетический код, общий для большинства про- и эукариот. В таблице приведены все 64 кодона и указаны соответствующие аминокислоты. Порядок оснований — от 5′ к 3′ концу мРНК.
Таблица 1. Стандартный генетический код
|
1-е ние |
2-е основание |
3-е ние |
|||||||
|
U |
C |
A |
G |
||||||
|
U |
UUU |
Фенилаланин (Phe/F) |
UCU |
Серин (Ser/S) |
UAU |
Тирозин (Tyr/Y) |
UGU |
Цистеин (Cys/C) |
U |
|
UUC |
UCC |
UAC |
UGC |
C |
|||||
|
UUA |
Лейцин (Leu/L) |
UCA |
UAA |
Стоп-кодон** |
UGA |
Стоп-кодон** |
A |
||
|
UUG |
UCG |
UAG |
Стоп-кодон** |
UGG |
Триптофан (Trp/W) |
G |
|||
|
C |
CUU |
CCU |
Пролин (Pro/P) |
CAU |
Гистидин (His/H) |
CGU |
Аргинин (Arg/R) |
U |
|
|
CUC |
CCC |
CAC |
CGC |
C |
|||||
|
CUA |
CCA |
CAA |
Глутамин (Gln/Q) |
CGA |
A |
||||
|
CUG |
CCG |
CAG |
CGG |
G |
|||||
|
A |
AUU |
Изолейцин (Ile/I) |
ACU |
Треонин (Thr/T) |
AAU |
Аспарагин (Asn/N) |
AGU |
Серин (Ser/S) |
U |
|
AUC |
ACC |
AAC |
AGC |
C |
|||||
|
AUA |
ACA |
AAA |
Лизин (Lys/K) |
AGA |
Аргинин (Arg/R) |
A |
|||
|
AUG |
Метионин* (Met/M) |
ACG |
AAG |
AGG |
G |
||||
|
G |
GUU |
Валин (Val/V) |
GCU |
Аланин (Ala/A) |
GAU |
Аспарагиновая кислота (Asp/D) |
GGU |
Глицин (Gly/G) |
U |
|
GUC |
GCC |
GAC |
GGC |
C |
|||||
|
GUA |
GCA |
GAA |
Глутаминовая кислота (Glu/E) |
GGA |
A |
||||
|
GUG |
GCG |
GAG |
GGG |
G |
Среди триплетов есть 4 специальных последовательности, выполняющих функции «знаков препинания»:
- *Триплет AUG, также кодирующий метионин, называется старт-кодоном. С этого кодона начинается синтез молекулы белка. Таким образом, во время синтеза белка, первой аминокислотой в последовательности всегда будет метионин.
- **Триплеты UAA, UAG и UGA называются стоп-кодонами и не кодируют ни одной аминокислоты. На этих последовательностях синтез белка прекращается.
Свойства генетического кода
1. Триплетность. Каждая аминокислота кодируется последовательностью из трех нуклеотидов – триплетом или кодоном.

2. Непрерывность. Между триплетами нет никаких дополнительных нуклеотидов, информация считывается непрерывно.

3. Неперекрываемость. Один нуклеотид не может входить одновременно в два триплета.

4. Однозначность. Один кодон может кодировать только одну аминокислоту.

5. Вырожденность. Одна аминокислота может кодироваться несколькими разными кодонами.

6. Универсальность. Генетический код одинаков для всех живых организмов.
Пример. Нам дана последовательность кодирующей цепи:
3’– CCGATTGCACGTCGATCGTATA– 5’.
Матричная цепь будет иметь последовательность:
5’– GGCTAACGTGCAGCTAGCATAT– 3’.
Теперь «синтезируем» с этой цепи информационную РНК:
3’– CCGAUUGCACGUCGAUCGUAUA– 5’.
Синтез белка идет в направлении 5’ → 3’, следовательно, нам нужно перевернуть последовательность, чтобы «прочитать» генетический код:
5’– AUAUGCUAGCUGCACGUUAGCC– 3’.
Теперь найдем старт-кодон AUG:
5’– AUAUGCUAGCUGCACGUUAGCC– 3’.
Разделим последовательность на триплеты:
Найдем стоп-кодон и согласно таблице генетического кода запишем последовательность аминокислот:

Центральная догма молекулярной биологии звучит следующим образом: информация с ДНК передается на РНК (транскрипция), с РНК – на белок (трансляция). ДНК также может удваиваться путем репликации, и также возможен процесс обратной транскрипции, когда по матрице РНК синтезируется ДНК, но такой процесс в основном характерен для вирусов.

Рис. 13. Центральная догма молекулярной биологии
ГЕНОМ: ГЕНЫ и ХРОМОСОМЫ
(общие понятия)
Геном – совокупность всех генов организма; его полный хромосомный набор.
Термин “геном” был предложен Г. Винклером в 1920 г. для описания совокупности генов, заключенных в гаплоидном наборе хромосом организмов одного биологического вида. Первоначальный смысл этого термина указывал на то, что понятие генома в отличие от генотипа является генетической характеристикой вида в целом, а не отдельной особи. С развитием молекулярной генетики значение данного термина изменилось. Известно, что ДНК, которая является носителем генетической информации у большинства организмов и, следовательно, составляет основу генома, включает в себя не только гены в современном смысле этого слова. Большая часть ДНК эукариотических клеток представлена некодирующими (“избыточными”) последовательностями нуклеотидов, которые не заключают в себе информации о белках и нуклеиновых кислотах. Таким образом, основную часть генома любого организма составляет вся ДНК его гаплоидного набора хромосом.
Гены — это участки молекул ДНК, кодирующие полипептиды и молекулы РНК
За последнее столетие наше представление о генах существенно изменилось. Ранее геном называли участок хромосомы, кодирующий или определяющий один признак или фенотипическое (видимое) свойство, например цвет глаз.
|
|
|
Рис. 14. Соответствие между кодирующими участками ДНК, мРНК и аминокислотной последовательностью полипептидной цепи. |
В 1940 г. Джордж Бидл и Эдвард Тейтем предложили молекулярное определение гена. Ученые обрабатывали споры гриба Neurospora crassa рентгеновским излучением и другими агентами, вызывающими изменения в последовательности ДНК (мутации), и обнаружили мутантные штаммы гриба, утратившие некоторые специфические ферменты, что в некоторых случаях приводило к нарушению целого метаболического пути. Бидл и Тейтем пришли к выводу, что ген — это участок генетического материала, который определяет или кодирует один фермент. Так появилась гипотеза «один ген — один фермент». Позднее эта концепция была расширена до определения «один ген — один полипептид», поскольку многие гены кодируют белки, не являющиеся ферментами, а полипептид может оказаться субъединицей сложного белкового комплекса.
На рис. 14 показана схема того, как триплеты нуклеотидов в ДНК определяют полипептид – аминокислотную последовательность белка при посредничестве мРНК. Одна из цепей ДНК играет роль матрицы для синтеза мРНК, нуклеотидные триплеты (кодоны) которой комплементарны триплетам ДНК. У некоторых бактерий и многих эукариот кодирующие последовательности прерываются некодирующими участками(так называемыми интронами).
Современное биохимическое определение гена еще более конкретно. Генами называются все участки ДНК, кодирующие первичную последовательность конечных продуктов, к которым относятся полипептиды или РНК, обладающие структурной или каталитической функцией.
Наряду с генами ДНК содержит и другие последовательности, выполняющие исключительно регуляторную функцию. Регуляторные последовательности могут обозначать начало или конец генов, влиять на транскрипцию или указывать место инициации репликации или рекомбинации. Некоторые гены могут экспрессироваться разными путями, при этом один и тот же участок ДНК служит матрицей для образования разных продуктов.
Мы можем приблизительно рассчитать минимальный размер гена, кодирующего средний белок. Каждая аминокислота в полипептидной цепи кодируется последовательностью из трех нуклеотидов; последовательности этих триплетов (кодонов) соответствуют цепочке аминокислот в полипептиде, который кодируется данным геном. Полипептидная цепь из 350 аминокислотных остатков (цепь средней длины) соответствует последовательности из 1050 п.н. (пар нуклеотидов). Однако многие гены эукариот и некоторые гены прокариот прерываются сегментами ДНК, не несущими информации о белке, и поэтому оказываются значительно длиннее, чем показывает простой расчет.
Сколько генов в одной хромосоме?

ДНК прокариот устроена более просто: их клетки не имеют ядра, поэтому ДНК находится непосредственно в цитоплазме в форме нуклеоида.

Прокариоты (Бактерии).

Большинство плазмид состоит всего из нескольких тысяч пар нуклеотидов, некоторые содержат более 10000 п. н. Они несут генетическую информацию и реплицируются с образованием дочерних плазмид, которые попадают в дочерние клетки в процессе деления родительской клетки. Плазмиды обнаружены не только в бактериях, но также в дрожжах и других грибах. Во многих случаях плазмиды не дают никаких преимуществ клеткам-хозяевам, и их единственная задача — независимое воспроизведение. Однако некоторые плазмиды несут полезные для хозяина гены. Например, содержащиеся в плазмидах гены могут придавать клеткам бактерий устойчивость к антибактериальным агентам. Плазмиды, несущие ген β-лактамазы, обеспечивают устойчивость к β-лактамным антибиотикам, таким как пенициллин и амоксициллин. Плазмиды могут переходить от клеток, устойчивых к антибиотикам, к другим клеткам того же или другого вида бактерий, в результате чего эти клетки также становятся резистентными. Интенсивное применение антибиотиков является мощным селективным фактором, способствующим распространению плазмид, кодирующих устойчивость к антибиотикам (а также транспозонов, которые кодируют аналогичные гены) среди болезнетворных бактерий, и приводит к появлению бактериальных штаммов с устойчивостью к нескольким антибиотикам. Врачи начинают понимать опасность широкого использования антибиотиков и назначают их только в случае острой необходимости. По аналогичным причинам ограничивается широкое использование антибиотиков для лечения сельскохозяйственных животных.
См. также: Равин Н.В., Шестаков С.В. Геном прокариот // Вавиловский журнал генетики и селекции, 2013. Т. 17. № 4/2. С. 972–984.
Эукариоты.
Таблица 2. ДНК, гены и хромосомы некоторых организмов
|
Общая ДНК, п.н. |
Число хромосом* |
Примерное число генов |
|
|
Escherichia coli (бактерия) |
4 639 675 |
1 |
4 435 |
|
Saccharomyces cerevisiae (дрожжи) |
12 080 000 |
16** |
5 860 |
|
Caenorhabditis elegans (нематода) |
90 269 800 |
12*** |
23 000 |
|
Arabidopsis thaliana (растение) |
119 186 200 |
10 |
33 000 |
|
Drosophila melanogaster (плодовая мушка) |
120 367 260 |
18 |
20 000 |
|
Oryza sativa (рис) |
480 000 000 |
24 |
57 000 |
|
Mus musculus (мышь) |
2 634 266 500 |
40 |
27 000 |
|
Homo sapiens (человек) |
3 070 128 600 |
46 |
29 000 |
Примечание. Информация постоянно обновляется; для получения более свежей информации обратитесь к сайтам, посвященным отдельным геномным проектам
*Для всех эукариот, кроме дрожжей, приводится диплоидный набор хромосом. Диплоидный набор хромосом (от греч. diploos- двойной и eidos- вид) – двойной набор хромосом (2n), каждая из которых имеет себе гомологичную.
**Гаплоидный набор. Дикие штаммы дрожжей обычно имеют восемь (октаплоидный) или больше наборов таких хромосом.
***Для самок с двумя Х хромосомами. У самцов есть Х хромосома, но нет Y, т. е. всего 11 хромосом.
В клетке дрожжей, одних из самых маленьких эукариот, в 2,6 раза больше ДНК, чем в клетке E. coli (табл. 2). Клетки плодовой мушки Drosophila, классического объекта генетических исследований, содержат в 35 раз больше ДНК, а клетки человека — примерно в 700 раз больше ДНК, чем клетки E. coli. Многие растения и амфибии содержат еще больше ДНК. Генетический материал клеток эукариот организован в виде хромосом. Диплоидный набор хромосом (2n) зависит от вида организма (табл. 2).
Например, в соматической клетке человека 46 хромосом (рис. 17). Каждая хромосома эукариотической клетки, как показано на рис. 17, а, содержит одну очень крупную двухспиральную молекулу ДНК. Двадцать четыре хромосомы человека (22 парные хромосомы и две половые хромосомы X и Y) различаются по длине более чем в 25 раз. Каждая хромосома эукариот содержит определенный набор генов.

Рис. 17. Хромосомы эукариот. а — пара связанных и конденсированных сестринских хроматид из хромосомы человека. В такой форме эукариотические хромосомы пребывают после репликации и в метафазе в процессе митоза. б — полный набор хромосом из лейкоцита одного из авторов книги. В каждой нормальной соматической клетке человека содержится 46 хромосом.
Размер и функция ДНК как матрицы для хранения и передачи наследственного материала объясняют наличие особых структурных элементов в организации этой молекулы. У высших организмов ДНК распределена между хромосомами.
Совокупность ДНК (хромосом) организма называется геномом. Хромосомы находятся в клеточном ядре и формируют структуру, называемую хроматином. Хроматин представляет собой комплекс ДНК и основных белков (гистонов) в соотношении 1:1. Длину ДНК обычно измеряют числом пар комплементарных нуклеотидов (п.н.). Например, 3-я хромосома человека представляет собой молекулу ДНК размером 160 млн п.н.. Выделенная линеаризованная ДНК размером 3*106 п.н. имеет длину примерно 1 мм, следовательно, линеаризованная молекула 3-й хромосомы человека была бы 5 мм в длину, а ДНК всех 23 хромосом (~3*109 п.н., MR = 1,8*1012) гаплоидной клетки – яйцеклетки или сперматозоида – в линеаризованном виде составляла бы 1 м. За исключением половых клеток, все клетки организма человека (их около 1013) содержат двойной набор хромосом. При клеточном делении все 46 молекул ДНК реплицируются и снова организуются в 46 хромосом.
Если соединить между собой молекулы ДНК человеческого генома (22 хромосомы и хромосомы X и Y или Х и Х), получится последовательность длиной около одного метра. Прим.: У всех млекопитающих и других организмов с гетерогаметным мужским полом, у самок две X-хромосомы (XX), а у самцов — одна X-хромосома и одна Y-хромосома (XY).
Большинство клеток человека диплоидны, поэтому общая длина ДНК таких клеток около 2м. У взрослого человека примерно 1014 клеток, таким образом, общая длина всех молекул ДНК составляет 2・1011 км. Для сравнения, окружность Земли — 4・104 км, а расстояние от Земли до Солнца — 1,5・108 км. Вот как удивительно компактно упакована ДНК в наших клетках!
В клетках эукариот есть и другие органеллы, содержащие ДНК, — это митохондрии и хлоропласты. Выдвигалось множество гипотез относительно происхождения ДНК митохондрий и хлоропластов. Общепризнанная сегодня точка зрения заключается в том, что они представляют собой рудименты хромосом древних бактерий, которые проникли в цитоплазму хозяйских клеток и стали предшественниками этих органелл. Митохондриальная ДНК кодирует митохондриальные тРНК и рРНК, а также несколько митохондриальных белков. Более 95% митохондриальных белков кодируется ядерной ДНК.
СТРОЕНИЕ ГЕНОВ
Рассмотрим строение гена у прокариот и эукариот, их сходства и различия. Несмотря на то, что ген — это участок ДНК, кодирующий всего один белок или РНК, кроме непосредственно кодирующей части, он также включает в себя регуляторные и иные структурные элементы, имеющие разное строение у прокариот и эукариот.
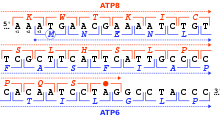
Кодирующая последовательность – основная структурно-функциональная единица гена, именно в ней находятся триплеты нуклеотидов, кодирующие аминокислотную последовательность. Она начинается со старт-кодона и заканчивается стоп-кодоном.
До и после кодирующей последовательности находятся нетранслируемые 5’- и 3’-последовательности. Они выполняют регуляторные и вспомогательные функции, например, обеспечивают посадку рибосомы на и-РНК.
Нетранслируемые и кодирующая последовательности составлют единицу транскрипции – транскрибируемый участок ДНК, то есть участок ДНК, с которого происходит синтез и-РНК.
Терминатор – нетранскрибируемый участок ДНК в конце гена, на котором останавливается синтез РНК.
В начале гена находится регуляторная область, включающая в себя промотор и оператор.
Промотор – последовательность, с которой связывается полимераза в процессе инициации транскрипции. Оператор – это область, с которой могут связываться специальные белки – репрессоры, которые могут уменьшать активность синтеза РНК с этого гена – иначе говоря, уменьшать его экспрессию.
Строение генов у прокариот
Общий план строения генов у прокариот и эукариот не отличается – и те, и другие содержат регуляторную область с промотором и оператором, единицу транскрипции с кодирующей и нетранслируемыми последовательностями и терминатор. Однако организация генов у прокариот и эукариот отличается.

Рис. 18. Схема строения гена у прокариот (бактерий) – изображение увеличивается
В начале и в конце оперона есть единые регуляторные области для нескольких структурных генов. С транскрибируемого участка оперона считывается одна молекула и-РНК, которая содержит несколько кодирующих последовательностей, в каждой из которых есть свой старт- и стоп-кодон. С каждого из таких участков синтезируется один белок. Таким образом, с одной молекулы и-РНК синтезируется несколько молекул белка.
Для прокариот характерно объединение нескольких генов в единую функциональную единицу – оперон. Работу оперона могут регулировать другие гены, которые могут быть заметно удалены от самого оперона – регуляторы. Белок, транслируемый с этого гена называется репрессор. Он связывается с оператором оперона, регулируя экспрессию сразу всех генов, в нем содержащихся.
Для прокариот также характерно явление сопряжения транскрипции и трансляции.

Рис. 19 Явление сопряжения транскрипции и трансляции у прокариот – изображение увеличивается
Такое сопряжение не встречается у эукариот из-за наличия у них ядерной оболочки, отделяющей цитоплазму, где происходит трансляция, от генетического материала, на котором происходит транскрипция. У прокариот во время синтеза РНК на матрице ДНК с синтезируемой молекулой РНК может сразу связываться рибосома. Таким образом, трансляция начинается еще до завершения транскрипции. Более того, с одной молекулой РНК может одновременно связываться несколько рибосом, синтезируя сразу несколько молекул одного белка.
Строение генов у эукариот
Гены и хромосомы эукариот очень сложно организованы
У бактерий многих видов всего одна хромосома, и почти во всех случаях в каждой хромосоме присутствует по одной копии каждого гена. Лишь немногие гены, например гены рРНК, содержатся в нескольких копиях. Гены и регуляторные последовательности составляют практически весь геном прокариот. Более того, почти каждый ген строго соответствует аминокислотной последовательности (или последовательности РНК), которую он кодирует (рис. 14).
Структурная и функциональная организация генов эукариот гораздо сложнее. Исследование хромосом эукариот, а позднее секвенирование полных последовательностей геномов эукариот принесло много сюрпризов. Многие, если не большинство, генов эукариот обладают интересной особенностью: их нуклеотидные последовательности содержат один или несколько участков ДНК, в которых не кодируется аминокислотная последовательность полипептидного продукта. Такие нетранслируемые вставки нарушают прямое соответствие между нуклеотидной последовательностью гена и аминокислотной последовательностью кодируемого полипептида. Эти нетранслируемые сегменты в составе генов называют интронами, или встроенными последовательностями, а кодирующие сегменты — экзонами. У прокариот лишь немногие гены содержат интроны.
Итак, у эукариот практически не встречается объединение генов в опероны, и кодирующая последовательность гена эукариот чаще всего разделена на транслируемые участки – экзоны, и нетранслируемые участки – интроны.
В большинстве случаев функция интронов не установлена. В целом, лишь около 1,5% ДНК человека являются ≪кодирующими≫, т. е. несут информацию о белках или РНК. Однако с учетом крупных интронов получается, что ДНК человека на 30% состоит из генов. Поскольку гены составляют относительно небольшую долю в геноме человека, значительная часть ДНК остается неучтенной.
Рис. 16. Схема строение гена у эукариот – изображение увеличивается
С каждого гена сначала синтезируется незрелая, или пре-РНК, которая содержит в себе как интроны, так и экзоны.
После этого проходит процесс сплайсинга, в результате которого интронные участки вырезаются, и образуется зрелая иРНК, с которой может быть синтезирован белок.
Рис. 20. Процесс альтернативного сплайсинга – изображение увеличивается
Такая организация генов позволяет, например, осуществить процесс альтернативного сплайсинга, когда с одного гена могут быть синтезированы разные формы белка, за счет того, что в процессе сплайсинга экзоны могут сшиваться в разных последовательностях.
Сравнение строения генов прокариот и эукариот
Рис. 21. Отличия в строении генов прокариот и эукариот – изображение увеличивается
МУТАЦИИ И МУТАГЕНЕЗ
Мутацией называется стойкое изменение генотипа, то есть изменение нуклеотидной последовательности.
Процесс, который приводит к возникновению мутаций называется мутагенезом, а организм, все клетки которого несут одну и ту же мутацию — мутантом.
Мутационная теория была впервые сформулирована Гуго де Фризом в 1903 году. Современный ее вариант включает в себя следующие положения:
1. Мутации возникают внезапно, скачкообразно.
2. Мутации передаются из поколения в поколение.
3. Мутации могут быть полезными, вредными или нейтральными, доминантными или рецессивными.
4. Вероятность обнаружения мутаций зависит от числа исследованных особей.
5. Сходные мутации могут возникать повторно.
6. Мутации не направленны.
Мутации могут возникать под действием различных факторов. Различают мутации, возникшие под действием мутагенных воздействий: физических (например, ультрафиолета или радиации), химических (например, колхицина или активных форм кислорода) и биологических (например, вирусов). Также мутации могут быть вызваны ошибками репликации.
В зависимости от условий появления мутации подразделяют на спонтанные — то есть мутации, возникшие в нормальных условиях, и индуцированые — то есть мутации, которые возникли при особых условиях.
Мутации могут возникать не только в ядерной ДНК, но и, например, в ДНК митохондрий или пластид. Соответственно, мы можем выделять ядерные и цитоплазматические мутации.
В результате возникновения мутаций часто могут появляться новые аллели. Если мутантный аллель подавляет действие нормального, мутация называется доминантной. Если нормальный аллель подавляет мутантный, такая мутация называется рецессивной. Большинство мутаций, приводящих к возникновению новых аллелей являются рецессивными.
По эффекту выделяют мутации адаптивные, приводящие к повышению приспособленности организма к среде, нейтральные, не влияющие на выживаемость, вредные, понижающие приспособленность организмов к условиям среды и летальные, приводящие к смерти организма на ранних стадиях развития.
По последствиям выделяются мутации, приводящие к потери функции белка, мутации, приводящие к возникновению у белка новой функции, а также мутации, которые изменяют дозу гена, и, соответственно, дозу белка синтезируемого с него.
Мутация может возникнуть к любой клетке организма. Если мутация возникает в половой клетке, она называется герминативной (герминальной, или генеративной). Такие мутации не проявляются у того организма, у которого они появились, но приводят к появлению мутантов в потомстве и передаются по наследству, поэтому они важны для генетики и эволюции. Если мутация возникает в любой другой клетке, она называется соматической. Такая мутация может в той или иной степени проявляться у того организма, у которого она возникла, например, приводить к образованию раковых опухолей. Однако такая мутация не передается по наследству и не влияет на потомков.
Мутации могут затрагивать разные по размеру участки генома. Выделяют генные, хромосомные и геномные мутации.
Генные мутации
Мутации, которые возникают в масштабе меньшем, чем один ген, называются генными, или точечными (точковыми). Такие мутации приводят к изменению одного и нескольких нуклеотидов в последовательности. Среди генных мутаций выделяют замены, приводящие к замене одного нуклеотида на другой, делеции, приводящие к выпадению одного из нуклеотидов, инсерции, приводящие к добавлению лишнего нуклеотида в последовательность.

Рис. 23. Генные (точечные) мутации
По механизму воздействия на белок, генные мутации делят на: синонимичные, которые (в результате вырожденности генетического кода) не приводят к изменению аминокислотного состава белкового продукта, миссенс-мутации, которые приводят к замене одной аминокислоты на другую и могут влиять на структуру синтезируемого белка, хотя часто они оказываются незначительными, нонсенс-мутации, приводящие к замене кодирующего кодона на стоп-кодон, мутации, приводящие к нарушению сплайсинга:

Рис. 24. Схемы мутаций
Также по механизму воздействия на белок выделяют мутации, приводящие к сдвигу рамки считывания, например, инсерции и делеции. Такие мутации, как и нонсенс-мутации, хоть и возникают в одной точке гена, часто воздействуют на всю структуру белка, что может привести к полному изменению его структуры.
Рис. 25. Схема мутации, приводящей к сдвигу рамки считывания
Хромосомные мутации
Рис. 26. Хромосомные абберации
Хромосомными мутациями называются мутации, которые затрагивают отдельные гены в рамках одной хромосомы. Различают делеции, когда теряется один или несколько генов, дупликации, когда удваивается тот или иной ген или несколько генов, инверсии, когда участок хромосомы поворачивается на 180 градусов, транслокации, когда гены переходят с одной хромосомы на другую.

Рис. 27. Схемы хромосомных мутаций: делеции, дупликации, инверсии
|
|
|
|
Рис. 28. Транслокация |
Рис. 29. Хромосома до и после дупликации |
Геномные мутации
Наконец, геномные мутации затрагивают весь геном целиком, то есть меняется количество хромосом. Выделяют полиплоидии — увеличение плоидности клетки, и анеуплоидии, то есть изменение количества хромосом, например, трисомии (наличие у одной из хромосом дополнительного гомолога) и моносомии (отсутствие у хромосомы гомолога).
Видео по теме ДНК
РЕПЛИКАЦИЯ ДНК, КОДИРОВАНИЕ РНК, СИНТЕЗ БЕЛКА
(Если видео не отображается оно доступно по ссылке→)
См. дополнительно:
- Нуклеиновые кислоты (PDF)
- Общие сведения о секвенировании биополимеров
- Метагеномика и микробиом
- Бактериальный иммунитет и система CRISPR/Cas
- Трансляция белка на рисбосоме (общие сведения)
- Раскрыт секрет спиральной структуры ДНК (новое о ДНК)
- Антимутагенные свойства пробиотиков (в свете защиты ДНК)
- МикроРНК, микробиом кишечника и иммунитет
- Эпигенетика, короткоцепочечные жирные кислоты и врожденная иммунная память
- Замедление старения: роль питательных веществ и микробиоты в модуляции эпигенома (о метилировании ДНК)
Литература в помощь:
Будьте здоровы!
ССЫЛКИ К РАЗДЕЛУ О ПРЕПАРАТАХ ПРОБИОТИКАХ
- ПРОБИОТИКИ
- ПРОБИОТИКИ И ПРЕБИОТИКИ
- СИНБИОТИКИ
- ДОМАШНИЕ ЗАКВАСКИ
- КОНЦЕНТРАТ БИФИДОБАКТЕРИЙ ЖИДКИЙ
- ПРОПИОНИКС
- ЙОДПРОПИОНИКС
- СЕЛЕНПРОПИОНИКС
- ГЕМОПРОПИОВИТ
- БИФИКАРДИО
- ПРОБИОТИКИ С ПНЖК
- МИКРОЭЛЕМЕНТНЫЙ СОСТАВ
- БИФИДОБАКТЕРИИ
- ПРОПИОНОВОКИСЛЫЕ БАКТЕРИИ
- МИКРОБИОМ ЧЕЛОВЕКА
- МИКРОФЛОРА ЖКТ
- ДИСБИОЗ КИШЕЧНИКА
- МИКРОБИОМ и ВЗК
- МИКРОБИОМ И РАК
- МИКРОБИОМ, СЕРДЦЕ И СОСУДЫ
- МИКРОБИОМ И ПЕЧЕНЬ
- МИКРОБИОМ И ПОЧКИ
- МИКРОБИОМ И ЛЕГКИЕ
- МИКРОБИОМ И ПОДЖЕЛУДОЧНАЯ ЖЕЛЕЗА
- МИКРОБИОМ И ЩИТОВИДНАЯ ЖЕЛЕЗА
- МИКРОБИОМ И КОЖНЫЕ БОЛЕЗНИ
- МИКРОБИОМ И КОСТИ
- МИКРОБИОМ И ОЖИРЕНИЕ
- МИКРОБИОМ И САХАРНЫЙ ДИАБЕТ
- МИКРОБИОМ И ФУНКЦИИ МОЗГА
- АНТИОКСИДАНТНЫЕ СВОЙСТВА
- АНТИОКСИДАНТНЫЕ ФЕРМЕНТЫ
- АНТИМУТАГЕННАЯ АКТИВНОСТЬ
- МИКРОБИОМ и ИММУНИТЕТ
- МИКРОБИОМ И АУТОИММУННЫЕ БОЛЕЗНИ
- ПРОБИОТИКИ и ГРУДНЫЕ ДЕТИ
- ПРОБИОТИКИ, БЕРЕМЕННОСТЬ, РОДЫ
- ВИТАМИННЫЙ СИНТЕЗ
- АМИНОКИСЛОТНЫЙ СИНТЕЗ
- АНТИМИКРОБНЫЕ СВОЙСТВА
- КОРОТКОЦЕПОЧЕЧНЫЕ ЖИРНЫЕ КИСЛОТЫ
- СИНТЕЗ БАКТЕРИОЦИНОВ
- АЛИМЕНТАРНЫЕ ЗАБОЛЕВАНИЯ
- МИКРОБИОМ И ПРЕЦИЗИОННОЕ ПИТАНИЕ
- ФУНКЦИОНАЛЬНОЕ ПИТАНИЕ
- ПРОБИОТИКИ ДЛЯ СПОРТСМЕНОВ
- ПРОИЗВОДСТВО ПРОБИОТИКОВ
- ЗАКВАСКИ ДЛЯ ПИЩЕВОЙ ПРОМЫШЛЕННОСТИ
- НОВОСТИ
Упоминаний об эпигенетике вы не встретите в школьном учебнике биологии, а ведь эта наука рассказывает, как клетка реализует свой генетический потенциал, «вылепливая» из одного и того же «теста» (последовательности ДНК) совершенно разные «пироги»: клетки эпителия, легкого, нервной ткани и многие другие. Эпигенетика изучает хроматин: ДНК и ассоциированные с ней РНК и белки, а также взаимодействия между ними. В этой статье, которой мы открываем спецпроект по эпигенетике, вы познакомитесь с основными игроками эпигенетики — молекулами хроматина. Много внимания мы уделим методам его изучения — для более глубокого понимания того, как ученые делают открытия в этой области.
Эпигенетика
Эпигенетика — с одной стороны, молодая и бурно развивающаяся наука. С другой стороны, в этой науке уже сложился фундамент основных понятий и концепций. Цель спецпроекта — рассказать об основных концепциях эпигенетики просто, но в то же время со всех сторон. Читатель узнает, какие молекулы и структуры стоят за передачей наследственной информацией и какими механизмами осуществляется эта передача. Часть контента позаимствована из блога автора Friends of Chromatin.
Генеральный партнер спецпроекта — компания «Диаэм».
Это крупнейший поставщик оборудования, реагентов и расходных материалов для биологических исследований и производств.
Партнер этой статьи — «СкайДжин».
Пять причин выбрать SkyGen:
- Доступ к высококачественной продукции для молекулярной биологии.
- Высококвалифицированная научная поддержка.
- Быстрая логистика и складская программа.
- Удобное и взаимовыгодное сотрудничество.
- Адекватные цены.
Введение
Молекулы и клеточное ядро
По принятой биохимической классификации, в клетках есть молекулы четырех основных типов:
- нуклеиновые кислоты — дезоксирибонуклеиновая (ДНК) и рибонуклеиновая (РНК);
- белки;
- липиды [5];
- углеводы .
Об удивительных углеводах читайте спецпроект «Гликобиология». — Ред.
Кроме этого, клетки наполнены молекулами меньшего размера, и даже атомами (например ионами). Они выполняют две основные функции: регуляторов для основных молекул (увеличивая или уменьшая их химическую активность) и «строительных блоков» для них. Роль «строителей» играют белки — например, они создают двухцепочечную молекулу ДНК из дезоксирибонуклеотидных остатков. Но и сами белки тоже нужно строить: они создаются специальными «машинами» из белков и РНК — рибосомами, где роль главного строителя играет уже РНК.
Итак, молекулы большого размера состоят из меньших строительных блоков, которые часто состоят из еще меньших. ДНК состоит из дезоксирибонуклеотидных остатков, РНК — из рибонуклеотидных, белки — из аминокислот, углеводы — из моносахаридов (которые, кстати, входят и в состав нуклеотидов). Липиды довольно разнообразны и являются единственной химической категорией, которую классифицируют не по строению, а по свойствам: они не смешиваются с водой (на чем основан важный для молекулярной биологии гидрофобный эффект [6]).
Клетки содержат органеллы (как бы очень маленькие клеточные органы). Клеточное ядро — одна из важнейших органелл (чаще всего оно шарообразное или в форме эллипсоида), отделенная ядерной оболочкой от остальной части клетки — цитоплазмы. В ядре происходит часть процессов, лежащих в основе «центральной догмы» молекулярной биологии: ДНК ⇆ РНК → белок (рис. 1).
Рисунок 1. Центральная догма молекулярной биологии. Линейная последовательность ДНК переводится в сложный трехмерный белок через промежуточный продукт — РНК. Только часть этого процесса (транскрипция, или перевод ДНК в РНК) происходит в ядре. После этого РНК, если она кодирует белок (а может и не кодировать), переносится в цитоплазму и транслируется (переводится) в белок на рибосомах, причем каждые три нуклеотида кодируют одну аминокислоту. Рибосомная РНК синтезируется и начинает собираться вместе с другими белками в комплексы тоже в ядре. Интересно, что у некоторых вирусов и «прыгающих» элементов ДНК, в жизни которых есть стадия РНК, возможен обратный перевод РНК в ДНК (обратная транскрипция). Но обратного перевода белков в РНК пока не обнаружили.
ДНК и РНК
ДНК состоит из нуклеотидных остатков. Они, в свою очередь, строятся из трех фрагментов: остатков сахара (дезоксирибозы), трифосфата и азотистых оснований. Несмотря на поразительную длину человеческой ДНК (3,2 миллиарда пар нуклеотидов в каждой клетке, что в развернутом состоянии даст два метра длины!), типов азотистых оснований всего четыре: аденин (А), тимин (Т), цитозин (Ц) и гуанин (Г). ДНК находится в двухцепочечном состоянии, где нуклеотиды одной цепочки образуют водородные связи с нуклеотидами другой. И действует строгое правило: аденин всегда выстраивается напротив тимина, а цитозин — напротив гуанина. Это правило носит название комплементарности. РНК организована сходным образом, с двумя главными отличиями. Во-первых, на месте тимина всегда находится другое азотистое основание: урацил (У). Во-вторых, хотя иногда в клетке встречаются небольшие двухцепочечные РНК из двух комплементарных цепей, чаще всего РНК существует в виде одной цепочки, одни участки которой связаны с белками, а другие комплементарны внутри одной молекулы, образуя сложные пространственные укладки [7].
Комплементарность лежит в основе копирования ДНК, в ходе которой цепи расплетаются, и к каждой цепи специальным арсеналом белков по матричному принципу [8] достраивается комплементарная. Так наследственный материал удваивается, чтобы потом разойтись по дочерним клеткам в ходе клеточного деления. РНК тоже может удваиваться. Это происходит или с маленькими РНК-цепочками у растений, дрожжей и червячка C. elegans [9], или с цепочками большего размера у вирусов, у которых наследственная информация находится в форме РНК, а не ДНК.
Комплементарность также лежит в основе прямой и обратной транскрипции — перевода ДНК в РНК и обратно соответственно. При транскрипции двухцепочечный участок ДНК расплетается, и к цепи, которая кодирует ген, комплементарно достраивается участок РНК, который отделяется от ДНК и после завершения транскрипции живет своей жизнью в клетке.
Хромосомы и хроматин
У эукариот молекулы ДНК в клеточном ядре упакованы в хромосомы, в которых одна длинная молекула ДНК намотана на глобулы из белков-гистонов (как бусины на нитке) и сложена в компактную структуру вмeсте с другими молекулами белков и РНК. Всё вещество хромосом называют хроматином [3]. Еще в 1928 году Эмиль Хайтц заметил, что одни участки хроматина компактнее других, и разделил хромосомное вещество на развернутый эу- и компактный гетерохроматин [10].
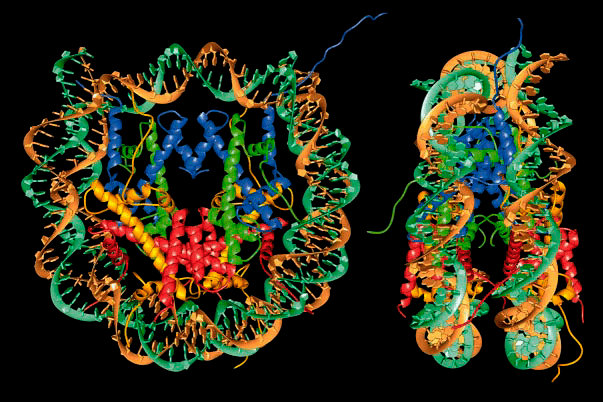
Глобулы из гистонов носят название нуклеосом. Нуклеосомы устроены удивительно регулярно: каждая глобула имеет один и тот же план строения — восемь молекул гистонов (по две молекулы гистонов 2А (H2А), 2B (H2B), 3 (H3) и 4 (H4)) сложены в компактную структуру таким образом, что тетрамер (комплекс из четырех белков) из гистонов H3 и H4 оказывается «зажат» между двумя димерами (комплексами из двух белков) H2A и H2B. Вокруг одной глобулы обернуто 146 пар оснований молекулы ДНК (рис. 2) [11]. Такая упаковка позволяет уложить ДНК длиной в 2 метра в ядро размером в несколько микрометров, а также регулирует чтение генов.
Рисунок 2. Кристаллическая структура нуклеосомы. 146 пар оснований ДНК обернуто вокруг октамера из гистонов.
Части гистонов выступают из нуклеосом и могут быть модифицированы — специальные белки-«писатели» пришивают к гистоновым «хвостам» разные химические группы: метильные, ацетильные, фосфатные, кротонильные, цитратные, серотонильные или же небольшие белки: убиквитин [12] и SUMO [13]. Другие белки — «читатели» — взаимодействуют с метками гистоновых «хвостов», причем в белках-«читателях» есть модули, специфично узнающие определенные метки. Еще есть специальные белки-«стиратели», снимающие метки с гистонов.
После открытий первых гистоновых модификаций Дэвид Аллис и Брайен Штраль выдвинули гипотезу «гистонового кода»: комбинации гистоновых меток являются кодом, который читается другими белками и локально определяет молекулярные процессы, происходящие в местах комбинаций меток [14]. Однако похоже, что картина сложнее, и далеко не только гистоновые метки управляют молекулярными процессами на хроматине. Кроме того, хотя метки и бывают взаимоисключающими, они действуют по отдельности, а не в комбинациях [15]. Белки-читатели узнают отдельные метки, а для регуляции процессов на хроматине иногда может быть достаточно всего одной модификации.
Интересно, что нуклеосомы могут отличаться друг от друга не только модификациями гистонов, но и самими гистонами. Есть так называемые варианты гистонов, которые могут заменять канонические гистоны в нуклеосомах. И структура нуклеосом с такими измененными гистонами слегка меняется.
Как устроено окружение нуклеосом с намотанной на них ДНК? С ними взаимодействуют тысячи белков и молекул РНК. Почти всегда белки и РНК собираются и находятся в больших комплексах. Иногда в одиночку — вне комплексов — они не могут выполнять свою функцию. Такие белковые комплексы — экзосомы и протеасомы [16] — разбирают синтезированные РНК и белки на нуклеотиды и цепочки из нескольких аминокислот соответственно. Большие комплексы из белка и РНК — сплайсосомы и комплексы полиаденилирования — осуществляют процессинг матричных РНК: вырезают из нее некодирующие участки и строят полиА-хвост на 3′-конце.
Короткие и длинные некодирующие РНК участвуют во многих процессах, происходящих в ядре, но в отличие от белков, редко обладают ферментативной активностью и, когда функционируют в комплексах, в основном играют структурную роль. Совсем короткие малые интерферирующие РНК (22 нуклеотидных остатка в длину) участвуют в образовании гетерохроматина и разрушении комплементарных РНК [7].
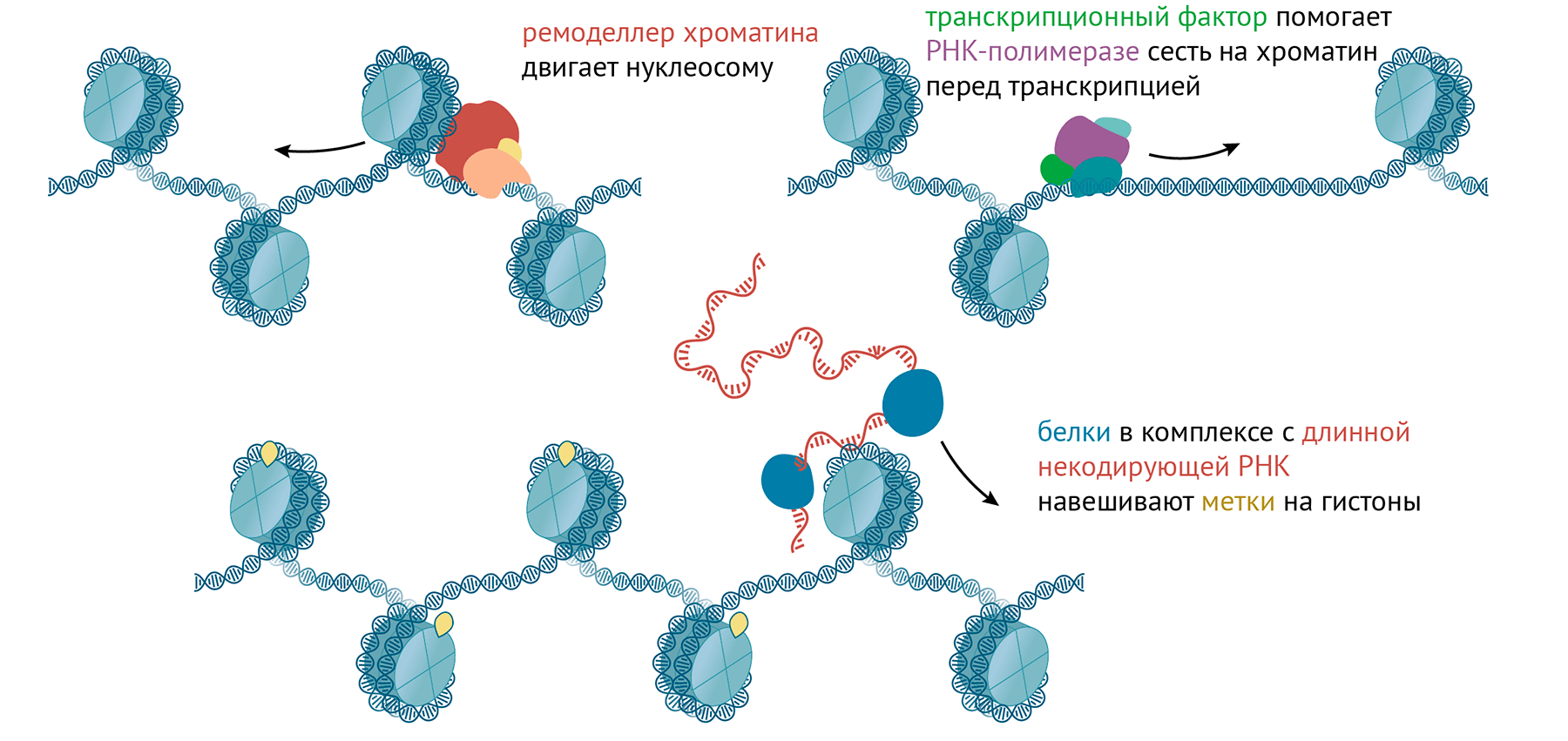
Рисунок 3. Хроматин на уровне нескольких нуклеосом. Кроме упомянутых белков-»читателей», «писателей» и «стирателей», взаимодействующие белки можно разделить по таким функциям:
- Транскрипционные факторы связываются с определенными последовательностями ДНК и помогают другим белкам связаться рядом. Например, они помогают ферменту РНК-полимеразе сесть на ДНК и начать транскрипцию — перевод последовательности ДНК в РНК. Или помогать связаться с ДНК белкам, которые строят гетерохроматин [17].
- Ремоделеры хроматина могут собирать, разбирать или двигать нуклеосомы взад-вперед по ДНК, а иногда накручивать ДНК на нуклеосомы и снимать с них двойную спираль, затрачивая при этом энергию АТФ.
- Хеликазы умеют расплетать двойную спираль ДНК и переплетать уже свернутые молекулы РНК.
- Топоизомеразы могут распутывать переплетенные или перезакрученные двойные спирали ДНК.
Линейные ДНК и РНК
Совокупность молекул ДНК образуют генóм. Ген — это фрагмент линейной последовательности ДНК, который может переводиться в РНК. Перед каждым гéном есть промотор — область, с которой связывается осуществляющий транскрипцию фермент (ДНК-зависимая РНК-полимераза). В конце гена находится терминатор — область, где РНК-полимераза отсоединяется от цепи ДНК, чтобы транскрипция закончилась. Интересно, что на транскрипцию гена могут влиять последовательности ДНК, отстоящие от него иногда на миллионы нуклеотидов, но расположенные близко к промотору в пространстве (об этом мы будем говорить гораздо подробнее дальше). Эти элементы называются энхансерами, если они увеличивают транскрипцию гена, или сайленсерами, если уменьшают. Некоторые элементы ДНК на линейной последовательности — инсуляторы — находятся между энхансером/сайленсером и промотором, не дают сблизиться им в 3D, и, соответственно, не дают повлиять на транскрипцию гена. Также инсуляторы могут ограничивать распространение разных типов хроматина по линейной ДНК.
У эукариот гены состоят из кодирующих и некодирующих участков, сменяющих друг друга на протяжении гена. Когда РНК с такого гена считывается, из нее вырезаются некодирующие участки (интроны), а кодирующие (экзоны) сшиваются. Такой процесс носит название сплайсинга. Иногда только некоторые кодирующие участки могут сшиваться, а другие деградируют (разбираются на строительные блоки). Это уже так называемый альтернативный сплайсинг.
Линейный хроматин
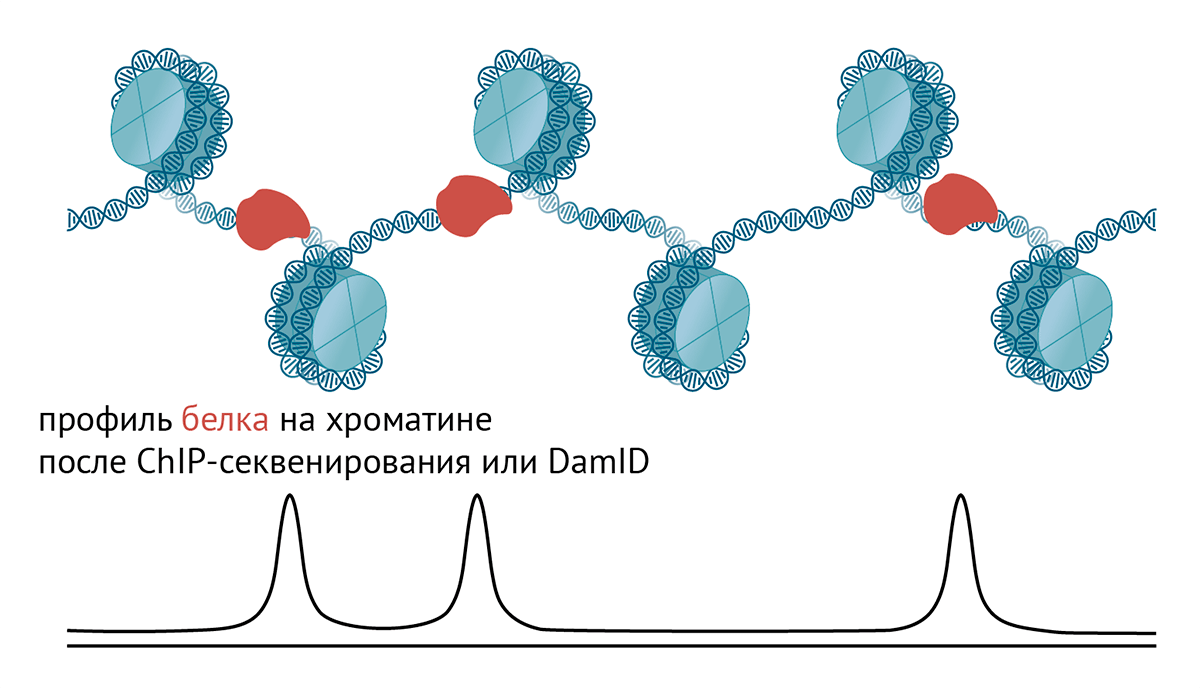
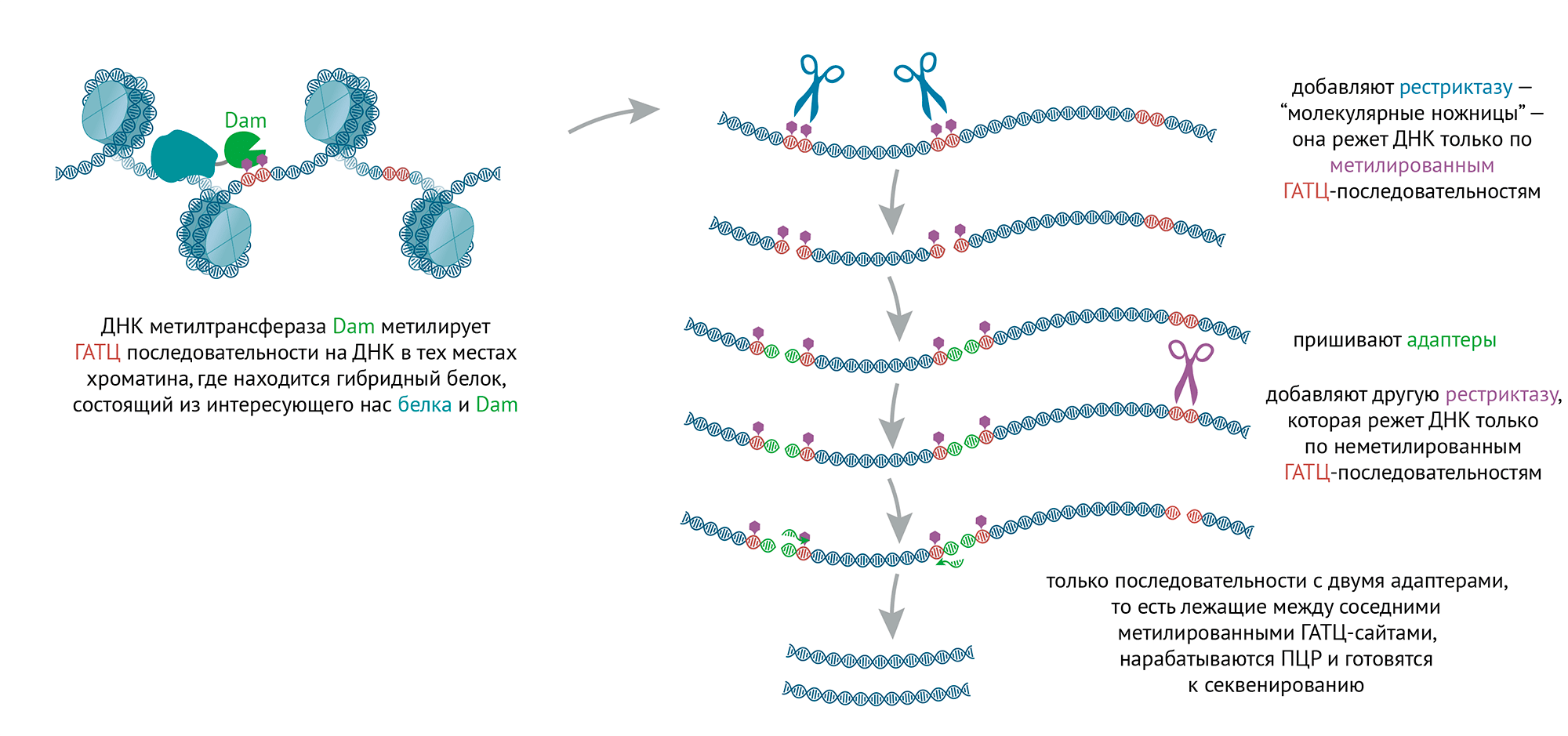
Как расположены белки и РНК на линейной последовательности ДНК? Во-первых, у каждого белка и по-своему модифицированного гистона свой неповторимый паттерн, или профиль связывания с ДНК (рис. 4). Даже когда профили связывания двух разных белков очень похожи, они немного отличаются друг от друга (рис. 5). Во-вторых, профили можно поделить на группы, а по этим группам профилей выделить типы хроматина. И получение профилей связывания, и выделение типов хроматина проводится с помощью компьютерных методов биоинформатики на основе данных ChIP-секвенирования и Dam-ID .
Зачастую эти биоинформатические методы очень сложны и основаны на математической теории; мы разбираем их в заключительной главе этой статьи.
Рисунок 4. Профиль белка на хроматине, полученный после ChIP-seq или Dam-ID. По горизонтали отображена координата линейной ДНК, по вертикали — количество белка на данном участке ДНК.
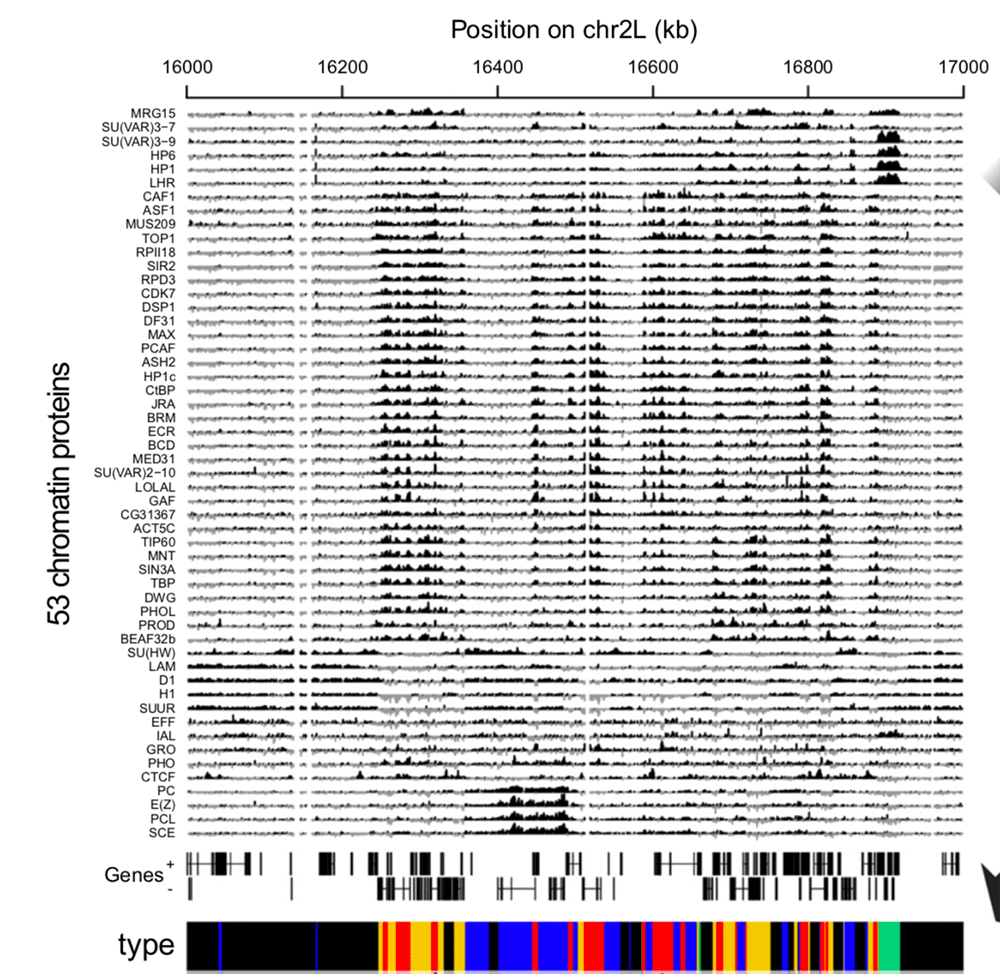
Так, в одной из работ в клетках дрозофилы методом DamID определили профили связывания с ДНК 53-х белков, и на основе этого выделили пять типов хроматина, которые условно назвали «цветами» (рис. 5). Два цвета — красный и желтый — отнесли к разным типам активного хроматина, а еще три — синий, зеленый и черный — к разным типам неактивного.
Рисунок 5. 53 профиля белков на хроматине после Dam-метилирования, объединенные в типы — «цвета» хроматина. Цвет, или тип хроматина, определяется в зависимости от того, какие белки преимущественно связываются с этими участками генома.
Чем отличаются активные цвета хроматина — красный и желтый? Когда ДНК удваивается, сначала удваиваются активные участки, а потом уже неактивные. Причем красные участки удваиваются раньше желтых. Кроме того, в желтом хроматине преимущественно расположены гены, которые активны всегда, а в красном — активные лишь при определенных условиях.
Два из трех неактивных цветов хроматина были известны и до этой работы. Зеленый тип хроматина — так называемый классический гетерохроматин, в котором присутствует белок HP1a (heterochromatic protein 1a, гетерохроматиновый белок 1а) и белки, которые взаимодействуют с ним (см. далее). На «синем» хроматине располагаются белки группы Polycomb, которые регулируют гены, важные для развития организма. Черный хроматин в этой работе открыли впервые. Выяснилось, что это примерно половина генома, и в нем сравнительно мало генов и транскрипции; если в нем и есть гены, то в основном неактивные [18].
В другой работе, основанной на ChiP-seq 18-и гистоновых модификаций, хроматин плодовой мушки дрозофилы поделили на девять типов [19]. Интересно, что многие (а возможно, и все) из них — разновидности уже знакомых нам пяти «цветов» [20]. После дрозофилы хроматин классифицировали на типы и в других организмах [21]: в 15 типах клеток мыши и шести человеческих клеточных линиях хроматин разделили на семь типов [22], а кроме того, похожее деление провели и для червячка C. elegans [23] и нескольких растений [24].
ДНК в 3D
Если взять ДНК из всех хромосом всего одной нашей клетки и вытянуть на полную длину, она составит целых два метра. Но диаметр клеточного ядра, в котором она уложена, — всего несколько микрометров. Следовательно, в ядре ДНК должна быть многократно свернута в пространстве.
Рисунок 6. Фибрилла 30 нм существует только in vitro
на основе модели фибриллы из [77]
Долгое время на основе экспериментов в пробирке, в которых собирали хроматиновые структуры из очищенной ДНК и очищенных гистонов, считалось, что ДНК, намотанная на нуклеосомы, упакована в фибриллу диаметром 30 нм (рис. 6). Однако недавно эксперименты по 3D электронной микроскопии на клетках (их удалось сделать благодаря специальной окраске ДНК) показали, что такой фибриллы нет, зато есть изогнутые цепи диаметром от 5 до 24 нм [25], [26].
Также еще до развития специальных методов для изучения пространственной укладки ДНК проводились эксперименты, показывающие, что ДНК в ядре организована в огромные петли. Эти петли можно было заметить с помощью электронной микроскопии после обработки ядер очень высоко концентрированной солью. Они были прикреплены к белковой основе ядра, оставшейся после того, как из ядра под действием соли уплыло много белков хроматина [27], [28].
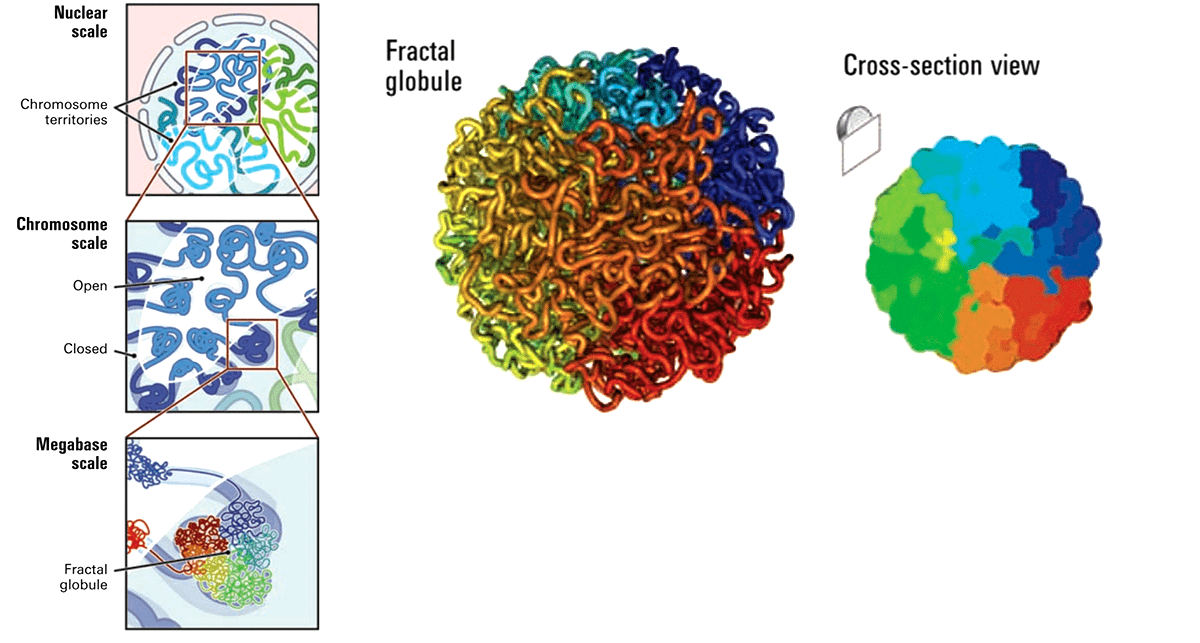
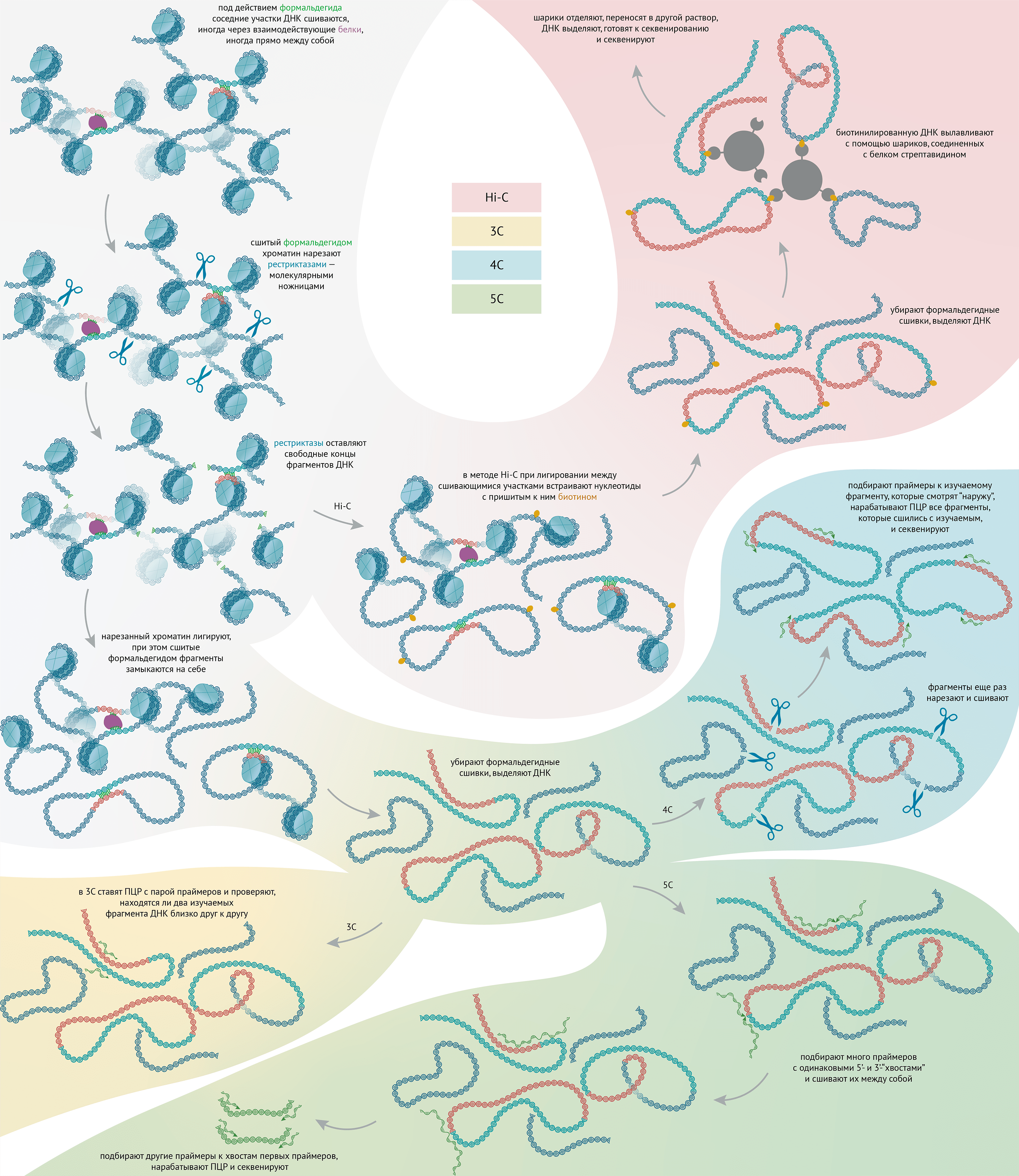
Много новых открытий пришло с развитием методов Hi-C [3], [29] и Micro-C [30], о которых мы расскажем в последней главе этой статьи. Во-первых, выяснилось, что участки ДНК сворачиваются в небольшие глобулы (что-то наподобие шариков из скомканных нитей), которые, в свою очередь, сворачиваются в глобулы всё большего и большего размера (рис. 7) [29]. Глобулы из последовательностей ДНК размером от 100 тысяч до 1,5 миллионов нуклеотидных остатков получили название топологически ассоциированных доменов (ТАДов) [31]. Во-вторых, многие участки ДНК выпетливаются. Как соотносятся петли и глобулы? Похоже, что основания части петель совпадают с основанием глобул [32]! То есть сначала формируются петли, которые иногда «комкаются» в глобулы. Внутри больших петель могут быть петли поменьше. Правда, у плодовой мушки дрозофилы наличие петли зафиксировать на границе ТАДов пока не удалось, только внутри [33].
Рисунок 7. Пространственная упаковка ДНК. Левая панель: укладка хроматина во фрактальные глобулы. Хромосомные территории состоят из глобул поменьше, которые, в свою очередь, состоят из глобул еще поменьше. Правая панель: фрактальная глобула в объеме и в разрезе.
Интересно, что есть как глобулы с активными генами, так и глобулы, в которых гены замалчиваются. И всё вещество хромосом, весь хроматин, можно разделить на два компартмента — А и B, которые отличаются друг от друга тем, как часто контактируют в них участки ДНК. В B побольше, в A поменьше.
Цитологи уже давно подметили, что когда клетка делится, ee хромосомы гораздо компактнее. У многих организмов, в том числе у человека, есть половые и неполовые (соматические) хромосомы. Из всего набора хромосом к каждой неполовой хромосоме можно подобрать очень похожую на нее. Две половые хромосомы могут быть как похожими (например, две Х хромосомы у женщины), так и разными (Х и Y хромосомы у мужчины). Так, по набору половых хромосом у многих организмов, включая человека, в большинстве случаев можно определить пол. (Однако в целом пол — понятие куда более комплексное, чем присутствие определенного набора хромосом [34], [35].)
Две одинаковые по набору и порядку генов хромосомы, соединенные перетяжкой, образуют нечто похожее на букву X, которой часто обозначают хромосомы в школьных учебниках. Как свернуты хромосомы в такие буквы Х? По тем же принципам петель, как и хромосомы в неделящихся в данный момент клетках, только немного по-другому. В случае хромосом клеток, которые делятся в данный момент, часть ДНК закреплена на линейном каркасе из белков, из которого исходят петли ДНК. И это не те же самые петли, что и в неделящихся на данный момент клетках, а другие [36], [37].
Когезины и конденсины
Что меняет форму хромосом, когда клетка собирается делиться? Этим занимаются белки конденсины.
Интересно, что они устроены очень похоже на белки, которые поддерживают петли в ДНК — когезины. Все эти белки собраны в конструкции, подобные кольцу, маленькая часть которого открывается. Именно в это отверстие и продевается ДНК, после чего оно закрывается. В случае когезинов, две двухцепочечные цепи ДНК оказываются зажаты в основании петли и не могут разойтись (как всё это выглядит, см. на видео 1). В случае конденсинов процесс изучен хуже, но, похоже, происходит примерно то же самое, только, в отличие от когезинов, они держат другие петли — не те, что присутствуют в хромосомах, когда клетка не делится (см. раздел о том, как ДНК уложена в ядре). Интересно, что комплексы могут не только держать петли, но и делать их. Этот процесс пока плохо изучен, но похоже, что белки перемещаются по ДНК, выпетливая ее, пока не встречают особые белки, связанные с блокирующими последовательностями ДНК — инсуляторами, — где и застревают [38–42].
Видео 1. Выпетливание хроматина
анимация лаборатории Леонида Мирного
Хроматин в 3D
В одном из предыдущих разделов мы обсудили, что можно выделить разные типы («цвета») активного и неактивного хроматина в зависимости от того, какие белки он связывает. Возникает логичный вопрос: если ДНК свернута в пространстве, как организованы в 3D разные участки линейного хроматина? Если у хроматина можно выделить «цвета», то как покрашено клеточное ядро?
Основной принцип укладки хроматина можно сформулировать так: подобное часто, но не всегда, взаимодействует с подобным. Например, активные и неактивные участки хроматина взаимодействуют с участками такой же активности в пространстве [43]. Также давно известно, что если покрасить клетку на некоторые белки, которые сидят преимущественно в одном из цветов хроматина (например, тот же HP1a), то такие белки образуют отдельные структуры в ядре. HP1a-гетерохроматин, например, образует от одного до трех доменов в зависимости от того, собирается клетка делиться или нет [44]. Еще было показано, что многие домены от 10 до 500 тысяч нуклеотидных остатков длиной образуют кластеры. Их можно увидеть в клетках под микроскопом, если последовательности ДНК этих доменов в клетках спарить со светящейся комплементарной ДНК [45].
В общем-то, тот факт, что ДНК свернута в пространстве выпетливанием в отдельные домены (например, ТАДы), уже подразумевает, что участки хроматина, недалекие друг от друга на линейной последовательности ДНК, с приличной вероятностью будут располагаться недалеко друг от друга в пространстве. И действительно, линейные хроматиновые домены коррелируют, то есть похожи на пространственные домены ДНК [46]. Уже известны целые ТАДы, целиком покрашенные в отдельный цвет хроматина (например, синие Polycomb-ТАДы у дрозофилы [47]).
Как связаны между собой функции разных белков?
Каждый белок в клетке взаимодействует с другими белками или иными молекулами, и следовательно, у него есть какая-то функция. А взаимодействует с Б, структура Б меняется, взаимодействие Б с другими молекулами меняется, запускается каскад. Конечно, речь идет только о значимом взаимодействии А с Б, меняющим Б, а не о любом.
Часто таких функций несколько, причем на первый взгляд они могут быть не связаны. Например, как в ядре, так и в цитоплазме есть протеасома, одна из субъединиц которой участвует в ремоделинге хроматина [48]. Как одна функция может влиять на другую? Давайте разберем несколько примеров.
Например, и у человека, и у мушек Drosophila есть белок MOF (males absent on the first, самцы отсутствуют в первом <поколении>), и самцы мух с мутантным MOF не развиваются. Этот белок навешивает ацетильные метки на гистоны и вовлечен в систему дозовой компенсации у мушек. У самок дрозофилы две Х-хромосомы, а у самцов одна. И чтобы гены с этой хромосомы читались у обоих полов в одинаковом объеме, их транскрипция у самцов удвоена. Один белок — MSL2 (male specific lethal 2, только самцы летальны 2) — синтезируется только у самцов. Он собирает на Х-хромосоме самцов комплекс из белков, в который входит и MOF, окрашивая Х-хроматин в особенный «цвет» (о чем речь шла выше). MOF навешивает на гистоны ацетильные метки, заряженные отрицательно и потому отталкивающиеся друг от друга, что делает хроматин более доступным для белков, осуществляющих транскрипцию. Таким образом, гены, на которых MOF ацетилирует гистоны , читаются лучше [49] (а это и есть дозовая компенсация).
Недавно выяснилось, что MOF находится не только в ядре, но и в митохондриях, где регулирует транскрипцию генов, отвечающих за дыхание [50].
Теперь один пример про неактивный хроматин. Тройное метилирование по девятому лизину гистона (H3K9me3) — самая известная модификация такого хроматина. Чтобы навесить метильные метки на этот лизин, может потребоваться его деацетилировать. С H3K9me3 связывается один из главных белков гетерохроматина — HP1a (heterochromatin protein 1 alpha, гетерохроматиновый белок 1 альфа). Молекулы HP1a на соседних нуклеосомах прочно взаимодействуют между собой, и таким образом сближают нуклеосомы. Таким образом, определенные модификации гистонов (снятие ацетильных и навешивание метильных меток на H3K9) компактизируют хроматин, снижая его активность [51], [52].
Известно, что возле точки начала транскрипции гена на ДНК нуклеосомы расположены определенным образом. В дрожжах было показано, что, если выключить в клетке транскрипцию, то позиции нуклеосом меняются [53]. Как мы помним, именно ремоделеры хроматина двигают нуклеосомы взад-вперед по ДНК. Следовательно, транскрипция влияет на ремоделинг хроматина. Может ли быть обратное? Да, может. Например, немного подвинув нуклеосому, ремоделер хроматина может высвободить специальную последовательность ДНК — сайт связывания транскрипционного фактора (белка, помогающего РНК-полимеразе сесть и начать транскрипцию гена), — который с ней свяжется и запустит транскрипцию [54].
Модельный эксперимент эпигенетики
Сегодня, открыв PubMed, можно получить представление о типичной работе по молекулярной биологии. Там, например, может описываться новая функция какого-то белка. Чтобы установить его функции, обычно делают иммунопреципитацию и масс-спектрометрию (см. методы) и определяют, какие белки взаимодействуют с изучаемым. Чтобы проверить предполагаемую функцию, белок убирают из клетки (или значительно снижают его уровень) и смотрят на фенотип: что изменилось без него?
Представьте: мы изучаем белок А, выявляем список белков, с которыми он взаимодействует, и получаем, что они связаны с транскрипцией. Мы формулируем гипотезу, что наш белок A повышает или понижает уровень транскрипции. Для ее проверки нужно понизить в клетках уровень A и определить количество РНК отдельных генов в этих клетках, а также сравнить его с обычными (контрольными) клетками. Так мы выясним, на транскрипцию каких генов влияет А (и влияет ли).
Можно дальше сделать ChIP-секвенирование и определить, на каких последовательностях в геноме «сидит» А (то есть — построить профиль белка А; см. рис. 4 и 5). Может выясниться, что это происходит на промоторах тех генов, которые он регулирует. Дальше можно поинтересоваться, что общего у «его» промоторов? Например, располагаются ли они в определенных хроматиновых доменах, на определенных хромосомах, на тех же местах, где «сидят» другие белки с известной функцией? Допустим, мы нашли, что промоторы с А располагаются в активном хроматине, а сам он повышает транскрипцию «своих» генов. Но как белку A это удается?
Посмотрим внимательнее на список белков, с которыми взаимодействует А. Допустим, мы нашли среди них Б, который навешивает на хвосты гистонов ацетильные метки (которые, как мы помним, расталкиваясь, «разрыхляют» хроматин и делают его доступнее для белков транскрипции). Можно выдвинуть гипотезу, что наш белок не просто связывается с ДНК на промоторах в активном хроматине, а привлекает туда белок Б, ацетилирующий гистоны и повышающий транскрипцию. Можно понизить уровень А в клетке и сделать ChIP-секвенирование белка Б и навешиваемой им ацетильной метки гистонов. Так можно установить, что количество Б на хроматине тоже понижается, и гистоны под влиянием А теряют ацетильные метки. И так далее. Можно задавать всё новые и новые вопросы и отвечать на них, хотя, конечно не всегда эксперименты сработают и дадут однозначный ответ.
Почему данные могут быть ложными или противоречить друг другу? Иногда потому, что мы неправильно их интерпретируем. Например, если вернуться к списку белков — потенциальных партнеров белка А, — то может оказаться, что идентификация Б была неспецифической: например, из-за высокой концентрации в ядре (а это вполне возможно для белков, связанных с транскрипцией, или ремоделеров хроматина).
Иногда данные, полученные в пробирке, ничего не говорят о процессах в культуре клеток, а они, в свою очередь, кардинально отличаются от того, что происходит в живом организме. Потому что живые клетки — гораздо более сложные системы, чем те, что собраны из молекул в пробирке, а культуры клеток, которые культивируют в лабораториях уже долгие-долгие годы, эволюционировали, изменились и уже не совсем похожи на организмы, из которых их взяли. Как бы то ни было, полученные данные, пусть и противоречивые, всегда учат нас чему-то новому, и их важно публиковать, пусть и не в самых трендовых журналах. К сожалению, не все ученые это делают.
Какие научные работы делают ученые? Или все статьи важны
Пожалуй, все работы можно разделить на два типа. Первые практически не меняют сложившуюся парадигму, лишь добавляя детали в уже существующую картину. Например, работы, которые открывают новые молекулы, взаимодействующие с интересующей нас молекулой А, без связи функции А с совершенно другими функциями взаимодействующих молекул. Или работы, описывающие, как молекула А взаимодействует с молекулой Б, без описания принципиально новых связей между функциями А и Б (или значимо новых элементов строения или взаимодействия А и Б, если это работа по структурной биологии). Такие работы могут быть очень важны, например, для подтверждения результатов больших исследований, где изучаются взаимодействия между собой тысяч молекул. В случае таких масштабных работ очень велик риск ошибок, и каждое новое открытое взаимодействие рассматривается как потенциальное и требующее подтверждения. И статьи, которые детально подтверждают отдельные взаимодействия, очень важны.
Второй тип работ — те, которые вводят новые научные концепции. Они значительно меняют схему происходящего, которую мы можем представить себе исходя только из предшествующих данных. Например, работы, объясняющие связь разных функций молекул, скажем, транскрипции и ремоделинга хроматина. Или работы, описывающие принципиально новые методы, структуру укладки ДНК в ядре, классифицирующие разные типы («цвета») хроматина.
И хотя работы второго типа кажутся более привлекательными, и именно их старается делать большая часть ученых, не стоит недооценивать работы, которые на первый взгляд не отвечают на какой-либо очень важный вопрос. Нельзя угадать, возможно, через много лет выяснится, что какая-то из давно открытых молекул очень важна и ее нужно изучать. И тут-то и пригодятся данные про эту молекулу, открытые давным-давно и плохо цитируемые.
В следующей статье цикла мы рассмотрим, что представляют собой хроматиновые домены и какими они бывают. Но в заключение этого материала идет еще одна — последняя — глава, рассказывающая о методах, которыми исследуют строение ядра клетки и упаковку ДНК в нем.
Методы изучения ДНК и хроматина
Как получить много ДНК и как определить ее последовательность?
Теперь давайте разберем несколько методов, которые ученые используют в своих исследованиях. Увеличение количества (амплификация) интересующего фрагмента ДНК — ключевая химическая реакция в молекулярной биологии. Она называется полимеразной цепной реакцией (ПЦР) [55], [56]. Например, чтобы клонировать ген (вставить его в плазмиду — кольцевую последовательность ДНК, которая может размножаться в бактериях), нужно получить огромное количество копий гена и плазмиды. Много плазмиды можно размножить в бактериях. А что делать с геном? В отличие от плазмиды, линейная последовательность в бактериях удваиваться не будет. Химически синтезировать длинную цепочку ДНК очень дорого. Зато множество копий целевого гена можно «наработать» при помощи ПЦР. Также, например, во многих методах для определения последовательности ДНК — секвенирования — нужно большое количество молекул ДНК, и их, опять-таки, можно «наработать» ПЦР.
Для РНК реакции амплификации не разработано, несмотря на то, что в клетках существует процесс удвоения РНК. Проблема в том, что РНК-зависимая РНК-полимераза, осуществляющая процесс, не сможет работать при высоких температурах, а при низких комплементарные цепи РНК будут не расходиться, а слипаться. Но молекулярным биологам это и не нужно. Много РНК можно наработать транскрипцией с ДНК, а ДНК уже наработать при помощи ПЦР.
Как определить последовательность ДНК? Технологии секвенирования появились в середине прошлого века, сейчас их уже много, и они всё еще совершенствуются.
Рассмотрим один из самых используемых методов: секвенирование на платформе «Иллюмина» [56], [57]. Двухцепочечная ДНК, которую нужно секвенировать, расщепляется на фрагменты, после чего к их концам ДНК-лигазами пришиваются короткие двухцепочечные последовательности — адаптеры. После этого количество фрагментов увеличивают с помощью ПЦР. Раствор с такими фрагментами наносится на платформу, к которой пришиты праймеры, комплементарные адаптерам. На платформе фрагменты ДНК копируются таким образом, что скопировавшиеся фрагменты оказываются пришитыми к платформе.
После этого можно провести секвенирование. Тут тоже не обходится без ПЦР, только с особенностями. К нуклеотидам (как мы помним, это строительные блоки для ДНК) присоединены цветные метки. После пришивания такого меченого нуклеотида к цепочке ДНК-полимераза не способна сама пришить следующий. Это обеспечивает паузу, за время которой можно определить (по цвету флуоресценции), что это за нуклеотид. Определение проводится специальной камерой над платформой, к которой пришиты фрагменты ДНК, служащие матрицей для ПЦР. После этого флуоресцентную метку от нуклеотида отщепляют, делая возможным присоединение следующего нуклеотида; определяют его «цвет»; и так далее, пока ПЦР не закончится.
Так можно узнать последовательности коротких фрагментов — ридов (от англ. read — читать), или прочтений. Их длина в технологии «Иллюмина» не превышает 600 нуклеотидных остатков, а чаще — ≈200–300. Поскольку при фрагментации ДНК (которая происходит в самом начале процедуры) разные молекулы разрываются в разных местах, риды будут перекрываться, и, «выровняв» их на компьютере, можно собрать длинные исходные последовательности ДНК. К сожалению, такая сборка может давать сбои в участках ДНК, состоящих из повторяющихся последовательностей.
Для секвенирования РНК чаще всего начинают с обратной транскрипции — синтеза ДНК с цепи РНК. Фермент, который осуществляет это, называется РНК-зависимой ДНК-полимеразой. И полученную ДНК потом секвенируют.
Совсем недавно появившийся метод секвенирования ДНК и РНК — нанопоровое секвенирование [58], [59]. Его принцип крайне прост по сравнению с секвенированием на платформе «Иллюмина». Представьте себе небольшую камеру, заполненную раствором электролита и разделенную липидной мембраной, содержащей белковую пору. Под напряжением через пору пойдет электрический ток (поток ионов), а если в камере окажется молекула ДНК или РНК, она, подобно червячку, тоже начнет протискиваться через пору, и с прохождением каждого нуклеотидного остатка ток будет меняться, причем — в соответствии с тем, какой именно остаток проходит через пору в данный момент. Длина ридов в нанопоровом секвенировании — много тысяч пар оснований нуклеотидных остатков! Это очень много по сравнению с «Иллюминой». К тому же, такое секвенирование становится всё дешевле, а приборы — всё меньше в размерах. Можно секвенировать ДНК или РНК, просто загрузив ее в прибор немногим больше флешки. Правда, к сожалению, при нанопоровом секвенировании велика вероятность ошибок, что ограничивает его применение.
Методы секвенирования рутинно применяются для определения последовательностей геномов и уровня РНК в клетках, а также являются заключительным этапом таких методов как ChiP-seq, Dnase-seq, ATAC-seq и других.
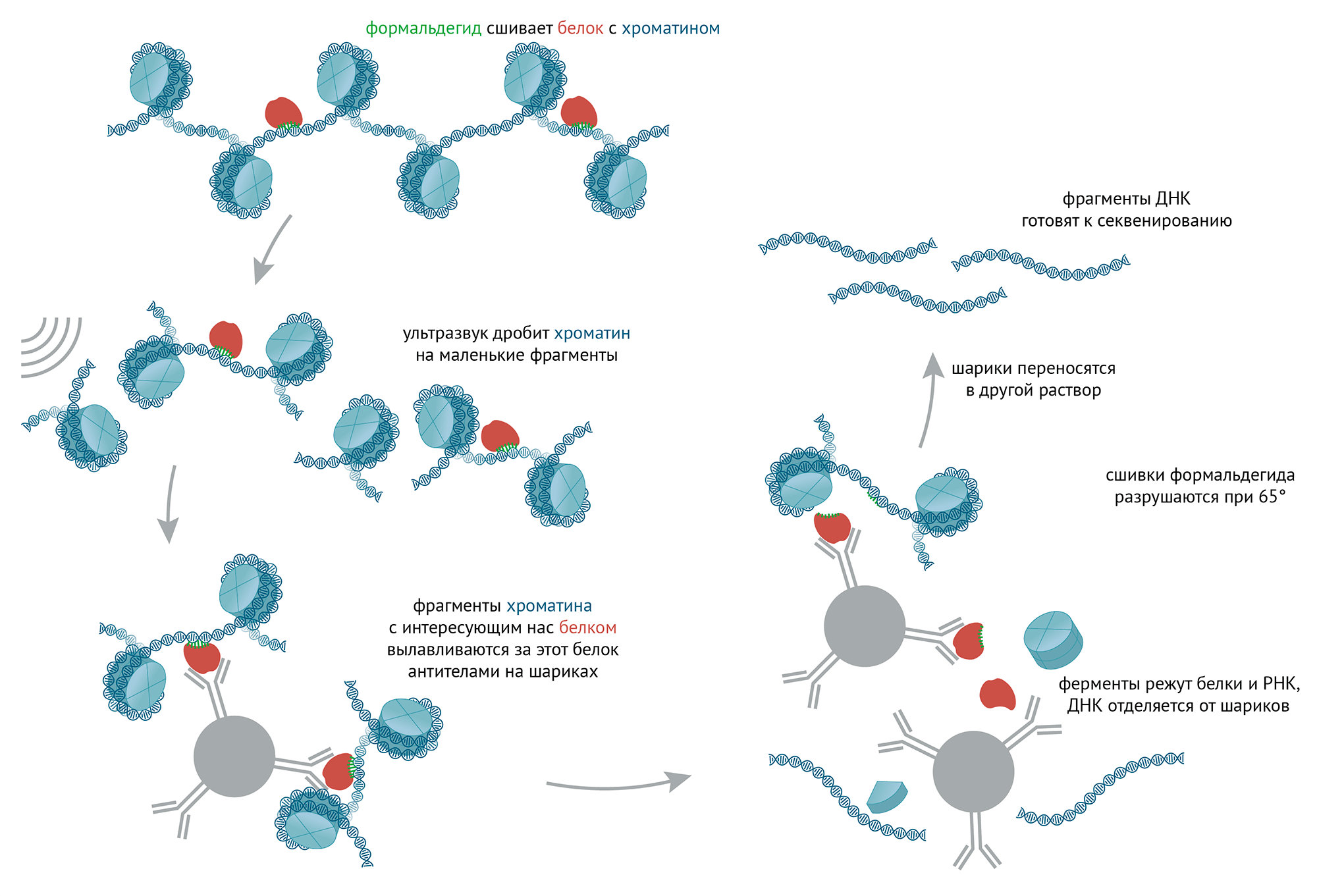
Определение профилей белков на хроматине
Для выявления местоположения белков на ДНК — то есть определения ДНК-профилей (см. рис. 4 и 5) — был разработан метод иммунопреципитации хроматина и секвенирования (ChIP-sequencing, chromatin immunoprecipitation sequencing; рис. 8) [60], [61].
Рисунок 8. Схема ChiP-секвенирования. Клетки фиксируют формальдегидом, сшивающим многие молекулы между собой. В том числе молекулы белков пришиваются к ДНК, если связаны с ней. Потом хроматин дробят на фрагменты (200–300 нуклеотидов) ультразвуком или ферментами эндонуклеазами (действуют на ДНК как молекулярные ножницы [62]). Важно, что при нарезке ДНК молекулы всё еще сшиты между собой. Потом добавляют антитело — белок, который специфично взаимодействует только с нужным нам белком и ни с какими другими. Белки связываются с антителами, а их в свою очередь можно выловить из раствора специальными шариками, «уцепившись» за константные части молекул антител.
Итак, теперь у нас есть комплексы сшитых белков, за которые держатся антитела, за которые в свою очередь держатся шарики, которые можно отделить от раствора. Такие шарики с антителами и молекулярными комплексами отмывают специальными растворами от неспецифически налипших молекул, а потом шарики с антителами и молекулярными комплексами отделяют от раствора в отдельную пробирку. Туда добавляют раствор с ферментами, которые разрушают белки и РНК, и нагревают ее до 65 °C. При этом белки и РНК разрушаются, а формальдегидные сшивки между молекулами от нагрева исчезают. Теперь можно очистить фрагменты ДНК, с которыми взаимодействовал исследуемый белок, и секвенировать их. Результаты секвенирования покажут, с какими именно участками исходной ДНК взаимодействовал исследуемый белок.
Антитела — белки в форме буквы Y, которые вырабатывает наша иммунная система. Aнтитело можно сделать, введя животному в кровь белок, с которым потенциальное антитело должно взаимодействовать. Потом специальные иммунные клетки вырабатывают антитела в крови животного в ответ на инородный белок. Можно изолировать эти иммунные клетки и сделать так, чтобы они производили антитела в условиях лаборатории. Далее антитела легко почистить и использовать в методах молекулярной биологии. Подробнее о применении антител в исследованиях и медицине можно прочесть в спецпроекте «Биомолекулы» «Терапевтические антитела», например в статье «Краткая история открытия и применения антител» [63].
Похожий подход используется и для определения профилей определенных молекул РНК на ДНК [64]. Только для того, чтобы вытащить сшитые молекулярные комплексы из раствора, там используют не антитела, а молекулы РНК, комплементарные изучаемой. Эти РНК, в свою очередь, пришиты к биотину. Модифицированные биотином молекулы РНК можно соединить с шариками, «увешанными» молекулами белка стрептавидина: это прекрасная «удочка» для биотина, ведь взаимодействие этих двух молекул — одно из самых сильных в молекулярной биологии. Называется такой метод «аффинная очистка РНК» (RNA affinity purification, RAP) [65], [66].
Наконец, последний метод, который мы рассмотрим в этом разделе, является альтернативой ChiP-seq’у, но выгодно отличается тем, что для него нам не требуется специфическое антитело под нужный нам белок. Метод называется «идентификация ДНК-аденин-метилирования» (DNA adenine methyltransferase identification, DamID) по имени фермента метилтрансферазы Dam, которая метилирует ДНК по аденинам строго в последовательностях ГАТЦ (рис. 9) [67]. Такой метилирующий ДНК фермент в природе встречается только в бактериях, а в эукариотах ни фермента, ни метилированного аденозина нет. Поэтому можно отличить метилирование, протекающее в клетке в естественных условиях (по цитозинам), от метилирования, которое сделал этот фермент. Ген фермента Dam пришивают к гену нужного нам белка, и этот гибрид доставляют в клетку, где с него считывается гибридная мРНК, в результате чего транслируется гибридный белок. Он метилирует ГАТЦ-последовательности преимущественно в тех местах, где сидит его натуральный аналог. Но ведь и вторая часть гибридного белка — фермент Dam — тоже будет взаимодействовать с какими-то белками хроматина, и следовательно, будет притаскивать гибридный белок на участки ДНК, где натурального белка нет. Поэтому в качестве контроля берут клетки, куда доставляют только ген белка Dam. Белок, получающийся с РНК такого гена, будет метилировать только места ДНК, куда идет он сам. И сравнивая сайты метилирования гибридного белка и одиночного белка Dam, мы сможем отличить сайты связывания гибридного белка, которые специфичны для исследуемого белка, от сайтов, которые специфичны для Dam.
Рисунок 9. Схема Dam-метилирования. Как обнаружить сайты метилирования на очищенной ДНК? Для этого ДНК сначала нарезают рестриктазой DpnI, которая режет только метилированные по аденину ГАТЦ-последовательности. Затем к такой нарезанной ДНК пришивают адаптеры — короткие двухцепочечные последовательности ДНК. После чего нарезанную ДНК с пришитыми адаптерами нарезают второй раз рестриктазой DpnII, которая режет только неметилированные ГАТЦ-последовательности. При этом с двумя адаптерами по обе стороны фрагмента остаются только те кусочки ДНК, которые лежат между двумя соседними метилированными ГАТЦ-последовательностями. И такие фрагменты нарабатывают ПЦР, подбирая праймеры к последовательностям адаптеров.
С помощью методов ChIP-секвенирования и Dam-ID ученые получили множество профилей разных белков на ДНК. В том числе, эти данные классифицировали на типы, или «цвета», хроматина (см. раздел «Линейный хроматин»).
Определение доступности хроматина
Традиционно доступность хроматина определяли, подвергая ядра действию ДНКазы, которая режет только открытые участки хроматина и не пробирается к закрытым. На основе этого фермента разработали высокопроизводительный метод детекции ДНКазо-чувствительности хроматина — DNAse-seq. Сначала ядра обрабатываются ДНКазой. Затем к концам участков, которые она порезала, лигируют биотинилированный адаптер. Потом ядра обрабатывают рестриктазой, и образуются фрагменты с рестриктным сайтом на одном конце и адаптером на другом. К ним пришивают второй адаптер. Затем такие фрагменты с двумя адаптерами отделяют на стрептавидиновые шарики и секвенируют на платформе «Иллюмина» [68].
Позже появился более продвинутый метод ATAC-seq (Assay for Transposase-Accessible Chromatin using sequencing — метод для определения хроматина, доступного транспозазе, используя секвенирование). В нем ядра инкубируют с ферментом транспозазой. Она вносит двухцепочечные разрывы в ДНК в открытых участках хроматина и пришивает адаптеры в местах разрывов. Затем такие вырезанные участки с адаптерами секвенируют [69].
Определение контактов ДНК в пространстве
Как отследить взаимодействующие участки ДНК в ядре? Для ответа на этот вопрос было разработано несколько методов, в основе которых лежит один и тот же принцип (рис. 10) [3], [70], [71].
Рисунок 10. Схема С-методов. Представим себе два двухцепочечных участка ДНК, соединенные каким-нибудь белком. Сначала клетки фиксируют формальдегидом. При этом все молекулы в клетках химически сшиваются. В том числе фрагменты ДНК сшиваются с белками, которые их соединяют. Затем добавляют фермент рестриктазу, он действует как молекулярные ножницы и разрезает ДНК по определенным последовательностям. При этом разрезы вносятся в том числе и в фрагменты ДНК, соединенные с белком. После этого сшитые молекулы с порезанной ДНК разводят в большом объеме и сшивают ДНК заново ферментом лигазой. При этом пара фрагментов ДНК, находящихся близко друг к другу, сшиваются. Потом формальдегидные, но не ДНК-сшивки, химически удаляют, и белки, которые связывали близкие друг к другу участки ДНК, отделяются от уже гибридных участков ДНК. Теперь у нас имеется набор линейных и кольцевых молекул ДНК (в зависимости от того, сшилась одна или обе части фрагментов), включающие в себя фрагменты ДНК, которые находились рядом. В 3С, подобрав правильные праймеры, методом ПЦР проверяют, находятся ли два известных участка ДНК (например, промотор и энхансер) близко друг к другу [72].
Во втором методе из серии — 4С — гибридные молекулы ДНК, полученные по схеме 3С, разрезают рестриктазами и сшивают лигазами еще раз, при этом образовавшиеся после рестриктаз короткие фрагменты ДНК лигируются сами на себя, образуя кольцевые молекулы. Дальше ставят так называемую обратную ПЦР — праймеры подбирают к концам фрагмента, последовательность которого мы знаем, только с тем расчетом, чтобы ДНК синтезировалась не «внутрь», а «наружу», на последовательностях, которые мы не знаем. Таким образом, нарабатываются все фрагменты ДНК, сшившиеся и, следовательно, находившиеся рядом с изучаемым фрагментом ДНК [73], [74].
В методе 5С подбирают коллекцию двух типов праймеров, у которых один конец одинаков, а другой нет. Они покрывают своими 3′- или 5′-фрагментами выбранную часть молекулы ДНК. Можно комплементарно спарить эти праймеры со сшитыми линейными или кольцевыми фрагментами из 3С, и сшить между собой праймеры, которые окажутся рядом. A потом методом ПЦР увеличить количество этих сшивок, подобрав праймеры к 3′- или 5′-концам, одинаковым у всех предыдущих праймеров [75].
И, наконец, самый последний метод — Hi-C — позволяет секвенировать всё подряд и построить карту взаимодействий всех участков со всеми. Это достигается за счет того, что при лигировании добавляются и встраиваются на липкие концы гибридных молекул нуклеотидные остатки, к которым пришит биотин. Как мы помним, биотин очень прочно взаимодействует с белком стрептавидином, это одно из самых сильных взаимодействий в молекулярной биологии. Пользуясь таким сильным взаимодействием, можно выловить часть гибридных фрагментов ДНК, куда встроился нуклеотидный остаток с биотином, на шарики с пришитым на них стрептавидином, и секвенировать гибридные участки [29].
В последнее время получил распространение метод Micro-C, который похож на Hi-C, но в котором вместо рестриктазы используется микрококковая нуклеаза. Этот фермент режет ДНК гораздо чаще рестриктазы, внося разрывы между нуклеосомами, и результаты Micro-C имеют более высокое разрешение [76].
Методы Hi-C и Micro-C произвели настоящую революцию в биологии клеточного ядра. Благодаря им мы знаем, по каким принципам ДНК уложена в ядре, и какие архитектурные элементы и детали есть у генома в пространстве (см. «Хроматин в 3D»).
Иммунопреципитация, биотинилирование окрестности и масс-спектрометрия
Чтобы определить новые белки, вовлеченные в определенный молекулярный каскад (и, следовательно, выполняющие какую-то молекулярную функцию), можно выявить неизвестные белки, взаимодействующие с уже известными, задействованными в этом каскаде. Один из способов сделать это — иммунопреципитация [83].
Для этого разрушают мембраны клеток, а из содержимого выделяют нужные части (например, ядра или митохондрии) и дробят их на отдельные молекулярные комплексы. Затем к материалу добавляют антитела или комплементарную РНК (в зависимости от того, вылавливаем ли мы из раствора белки или РНК), соединенные с шариками, и перемешивают раствор. Через некоторое время нужные молекулы, налипшие на шарики, можно извлечь и отмыть от неспецифически связавшихся молекул. Дальше белки на шариках можно разрезать протеазами на пептиды — специальными молекулярными ножницами для белков. Какие именно пептиды плавают в растворе, можно определить масс-спектрометрией [84], а по этому набору пептидов определить, какие белки антитела вытащили из клетки. Если же нас интересует РНК на шариках, то можно ее отделить от белков и секвенировать.
Интересно, что специальным методом можно выявить не только белки, взаимодействующие с «нашим», но и расположенные неподалеку от него в пространстве! Такой метод появился относительно недавно и получил название «биотинилирование окрестности» (proximity labeling) [85], [86]. Суть его в том, что ген изучаемого белка соединяют с геном специального белка, биотинилирующего всё вокруг себя. Такую конструкцию доставляют в клетку, а дальше происходит следующее: часть этого химерного белка будет располагаться в том же месте клетки, что и нативный белок. А биотинилирующий всё вокруг фрагмент будет притаскиваться в это место в клетке и биотинилировать всё вокруг. Конечно, нужно учитывать, что гибридный белок будет идти и по путям биотинилирующего белка, но для этого ставят и отрицательный контроль: смотрят, что он биотинилирует в тех же клетках сам по себе, не в составе химеры. Потом биотинилированные белки можно связать со стрептавидиновыми шариками, почистить и определить масс-спектрометрией.
Недавно выяснилось, что один из используемых биотинилирующих белков может биотинилировать РНК [87], а также ДНК [88].
Вероятно, метод скоро будет применяться в качестве альтернативы ChIP-seq’у и DamID для белков, которые очень подвижны на ДНК и которые очень сложно исследовать двумя известными методами (например, некоторые ремоделеры хроматина). Дело в том, что биотинилирование одним из известных белков занимает всего одну минуту, а сшивка формальдегидом — минимум 5–10 минут, не говоря уже о часах, которые занимает DamID. Поэтому на профилях таких белков, полученных ChIP-seq’ом и DamID, сложно выделить пики — места, на которых сидит изучаемый белок. А метод с биотинилирующим белком мог бы эту проблему решить и дать пики, полученные одноминутной реакцией.
Данные, полученные иммунопреципитацией и биотинилированием окрестности, отвечают на разные вопросы и дополняют друг друга. В случае иммунопреципитации мы получаем список всех белков/РНК, которые прочно взаимодействуют с нашим белком, а в случае биотинилирования окрестности — список всего, что находится поблизости. Последнее может быть важно, поскольку некоторые важные взаимодействия очень слабые или короткоживущие, и иммунопреципитацией их не поймать.
Оба метода рутинно используются в типичных экспериментах по эпигенетике. Благодаря им были описаны составы многих белковых комплексов и органелл.
- От атомов к древу (рецензия);
- От атомов к древу (отрывок из книги);
- Путешествие внутрь клеточного ядра, или Системная биология хроматина;
- Молекулярная биология;
- Липидный фундамент жизни;
- Физическая водобоязнь;
- Обо всех РНК на свете, больших и малых;
- Инге-Вечтомов С.Г. (2003). Матричный принцип в биологии (прошлое, настоящее, будущее?). «Экологическая генетика». 1 (спецвыпуск), 3–15;
- Модельные организмы: нематода;
- Passarge E. (1979). Emil Heitz and the concept of heterochromatin: longitudinal chromosome differentiation was recognized fifty years ago. Am. J. Hum. Genet. 31 (2), 106–115;
- Karolin Luger, Armin W. Mäder, Robin K. Richmond, David F. Sargent, Timothy J. Richmond. (1997). Crystal structure of the nucleosome core particle at 2.8 Å resolution. Nature. 389, 251-260;
- Вездесущий убиквитин;
- SUMO: японская борьба или уникальная посттрансляционная модификация?;
- Brian D. Strahl, C. David Allis. (2000). The language of covalent histone modifications. Nature. 403, 41-45;
- Steven Henikoff, John M. Greally. (2016). Epigenetics, cellular memory and gene regulation. Current Biology. 26, R644-R648;
- Война и мир: как устроить белковую жизнь?;
- Aydan Bulut-Karslioglu, Valentina Perrera, Manuela Scaranaro, Inti Alberto de la Rosa-Velazquez, Suzanne van de Nobelen, et. al.. (2012). A transcription factor–based mechanism for mouse heterochromatin formation. Nat Struct Mol Biol. 19, 1023-1030;
- Guillaume J. Filion, Joke G. van Bemmel, Ulrich Braunschweig, Wendy Talhout, Jop Kind, et. al.. (2010). Systematic Protein Location Mapping Reveals Five Principal Chromatin Types in Drosophila Cells. Cell. 143, 212-224;
- Peter V. Kharchenko, Artyom A. Alekseyenko, Yuri B. Schwartz, Aki Minoda, Nicole C. Riddle, et. al.. (2011). Comprehensive analysis of the chromatin landscape in Drosophila melanogaster. Nature. 471, 480-485;
- Bas van Steensel. (2011). Chromatin: constructing the big picture. The EMBO Journal. 30, 1885-1895;
- Jason Ernst, Manolis Kellis. (2012). ChromHMM: automating chromatin-state discovery and characterization. Nat Methods. 9, 215-216;
- Feng Yue, The Mouse ENCODE Consortium, Yong Cheng, Alessandra Breschi, Jeff Vierstra, et. al.. (2014). A comparative encyclopedia of DNA elements in the mouse genome. Nature. 515, 355-364;
- Aaron C. Daugherty, Robin W. Yeo, Jason D. Buenrostro, William J. Greenleaf, Anshul Kundaje, Anne Brunet. (2017). Chromatin accessibility dynamics reveal novel functional enhancers in C. elegans. Genome Res.. 27, 2096-2107;
- Yue Liu, Tian Tian, Kang Zhang, Qi You, Hengyu Yan, et. al.. (2018). PCSD: a plant chromatin state database. Nucleic Acids Research. 46, D1157-D1167;
- Horng D. Ou, Sébastien Phan, Thomas J. Deerinck, Andrea Thor, Mark H. Ellisman, Clodagh C. O’Shea. (2017). ChromEMT: Visualizing 3D chromatin structure and compaction in interphase and mitotic cells. Science. 357, eaag0025;
- Организовать геном: запутанная история гипотез и экспериментов;
- Hancock R. and Hughes M.E. (1982). Organization of DNA in the eukaryotic nucleus. Biol. Cell. 44, 201–212;
- James R. Paulson, U.K. Laemmli. (1977). The structure of histone-depleted metaphase chromosomes. Cell. 12, 817-828;
- E. Lieberman-Aiden, N. L. van Berkum, L. Williams, M. Imakaev, T. Ragoczy, et. al.. (2009). Comprehensive Mapping of Long-Range Interactions Reveals Folding Principles of the Human Genome. Science. 326, 289-293;
- Tsung-Han S. Hsieh, Assaf Weiner, Bryan Lajoie, Job Dekker, Nir Friedman, Oliver J. Rando. (2015). Mapping Nucleosome Resolution Chromosome Folding in Yeast by Micro-C. Cell. 162, 108-119;
- Jesse R. Dixon, Siddarth Selvaraj, Feng Yue, Audrey Kim, Yan Li, et. al.. (2012). Topological domains in mammalian genomes identified by analysis of chromatin interactions. Nature. 485, 376-380;
- Suhas S.P. Rao, Miriam H. Huntley, Neva C. Durand, Elena K. Stamenova, Ivan D. Bochkov, et. al.. (2014). A 3D Map of the Human Genome at Kilobase Resolution Reveals Principles of Chromatin Looping. Cell. 159, 1665-1680;
- Kyle P. Eagen. (2018). Principles of Chromosome Architecture Revealed by Hi-C. Trends in Biochemical Sciences. 43, 469-478;
- Мальчик или девочка;
- Мальчики налево, девочки направо… А остальные?;
- Johan H. Gibcus, Kumiko Samejima, Anton Goloborodko, Itaru Samejima, Natalia Naumova, et. al.. (2018). A pathway for mitotic chromosome formation. Science. 359, eaao6135;
- N. Naumova, M. Imakaev, G. Fudenberg, Y. Zhan, B. R. Lajoie, et. al.. (2013). Organization of the Mitotic Chromosome. Science. 342, 948-953;
- Geoffrey Fudenberg, Nezar Abdennur, Maxim Imakaev, Anton Goloborodko, Leonid A. Mirny. (2017). Emerging Evidence of Chromosome Folding by Loop Extrusion. Cold Spring Harb Symp Quant Biol. 82, 45-55;
- Mahipal Ganji, Indra A. Shaltiel, Shveta Bisht, Eugene Kim, Ana Kalichava, et. al.. (2018). Real-time imaging of DNA loop extrusion by condensin. Science. 360, 102-105;
- Ryu J.-K., Rah S.-H., Janissen R., Kerssemakers J.W.J., Dekker C. (2020). Resolving the step size in condensing — driven DNA loop extrusion identifies ATP binding as the step — generating process. bioRxiv;
- Je-Kyung Ryu, Allard J. Katan, Eli O. van der Sluis, Thomas Wisse, Ralph de Groot, et. al.. (2020). The condensin holocomplex cycles dynamically between open and collapsed states. Nat Struct Mol Biol. 27, 1134-1141;
- Yan Li, Judith H. I. Haarhuis, Ángela Sedeño Cacciatore, Roel Oldenkamp, Marjon S. van Ruiten, et. al.. (2020). The structural basis for cohesin–CTCF-anchored loops. Nature. 578, 472-476;
- Wendy A. Bickmore, Bas van Steensel. (2013). Genome Architecture: Domain Organization of Interphase Chromosomes. Cell. 152, 1270-1284;
- Irene Chiolo, Aki Minoda, Serafin U. Colmenares, Aris Polyzos, Sylvain V. Costes, Gary H. Karpen. (2011). Double-Strand Breaks in Heterochromatin Move Outside of a Dynamic HP1a Domain to Complete Recombinational Repair. Cell. 144, 732-744;
- Alistair N. Boettiger, Bogdan Bintu, Jeffrey R. Moffitt, Siyuan Wang, Brian J. Beliveau, et. al.. (2016). Super-resolution imaging reveals distinct chromatin folding for different epigenetic states. Nature. 529, 418-422;
- Tom Sexton, Eitan Yaffe, Ephraim Kenigsberg, Frédéric Bantignies, Benjamin Leblanc, et. al.. (2012). Three-Dimensional Folding and Functional Organization Principles of the Drosophila Genome. Cell. 148, 458-472;
- Stefania del Prete, Pawel Mikulski, Daniel Schubert, Valérie Gaudin. (2015). One, Two, Three: Polycomb Proteins Hit All Dimensions of Gene Regulation. Genes. 6, 520-542;
- Tyler McCann, William Tansey. (2014). Functions of the Proteasome on Chromatin. Biomolecules. 4, 1026-1044;
- Tobias Straub, Peter B. Becker. (2007). Dosage compensation: the beginning and end of generalization. Nat Rev Genet. 8, 47-57;
- Aindrila Chatterjee, Janine Seyfferth, Jacopo Lucci, Ralf Gilsbach, Sebastian Preissl, et. al.. (2016). MOF Acetyl Transferase Regulates Transcription and Respiration in Mitochondria. Cell. 167, 722-738.e23;
- Joel C. Eissenberg, Sarah C.R. Elgin. (2014). HP1a: a structural chromosomal protein regulating transcription. Trends in Genetics. 30, 103-110;
- M. David Stewart, Jiwen Li, Jiemin Wong. (2005). Relationship between Histone H3 Lysine 9 Methylation, Transcription Repression, and Heterochromatin Protein 1 Recruitment. MCB. 25, 2525-2538;
- Singh A.K., Schauer T., Pfaller L., Straub T., Mueller-Planitz F. (2021). The biogenesis and function of nucleosome arrays. bioRxiv;
- Denis Tolkunov, Karl A. Zawadzki, Cara Singer, Nils Elfving, Alexandre V. Morozov, James R. Broach. (2011). Chromatin remodelers clear nucleosomes from intrinsically unfavorable sites to establish nucleosome-depleted regions at promoters. MBoC. 22, 2106-2118;
- 12 методов в картинках: полимеразная цепная реакция;
- Важнейшие методы молекулярной биологии и генной инженерии;
- 12 методов в картинках: секвенирование нуклеиновых кислот;
- D. Stoddart, A. J. Heron, E. Mikhailova, G. Maglia, H. Bayley. (2009). Single-nucleotide discrimination in immobilized DNA oligonucleotides with a biological nanopore. Proceedings of the National Academy of Sciences. 106, 7702-7707;
- Нанопоровое секвенирование: на пороге третьей геномной революции;
- Gordon Robertson, Martin Hirst, Matthew Bainbridge, Misha Bilenky, Yongjun Zhao, et. al.. (2007). Genome-wide profiles of STAT1 DNA association using chromatin immunoprecipitation and massively parallel sequencing. Nat Meth. 4, 651-657;
- Новый метод CETCh-seq может за одну метку поймать много результатов;
- Benjamin L Kidder, Gangqing Hu, Keji Zhao. (2011). ChIP-Seq: technical considerations for obtaining high-quality data. Nat Immunol. 12, 918-922;
- Краткая история открытия и применения антител;
- Как молекулы РНК общаются с хроматином;
- J. M. Engreitz, A. Pandya-Jones, P. McDonel, A. Shishkin, K. Sirokman, et. al.. (2013). The Xist lncRNA Exploits Three-Dimensional Genome Architecture to Spread Across the X Chromosome. Science. 341, 1237973-1237973;
- Загадочное путешествие некодирующей РНК Xist по X-хромосоме;
- Bas van Steensel, Steven Henikoff. (2000). Identification of in vivo DNA targets of chromatin proteins using tethered Dam methyltransferase. Nat Biotechnol. 18, 424-428;
- L. Song, G. E. Crawford. (2010). DNase-seq: A High-Resolution Technique for Mapping Active Gene Regulatory Elements across the Genome from Mammalian Cells. Cold Spring Harbor Protocols. 2010, pdb.prot5384-pdb.prot5384;
- Jason D. Buenrostro, Beijing Wu, Howard Y. Chang, William J. Greenleaf. (2015). ATAC‐seq: A Method for Assaying Chromatin Accessibility Genome‐Wide. Current Protocols in Molecular Biology. 109;
- Новый взгляд на геном: не просто цепочка генов, а трехмерная сеть, интегрирующая функциональные домены ядра;
- Пространственный контакт;
- J. Dekker. (2002). Capturing Chromosome Conformation. Science. 295, 1306-1311;
- Marieke Simonis, Petra Klous, Erik Splinter, Yuri Moshkin, Rob Willemsen, et. al.. (2006). Nuclear organization of active and inactive chromatin domains uncovered by chromosome conformation capture–on-chip (4C). Nat Genet. 38, 1348-1354;
- Zhihu Zhao, Gholamreza Tavoosidana, Mikael Sjölinder, Anita Göndör, Piero Mariano, et. al.. (2006). Circular chromosome conformation capture (4C) uncovers extensive networks of epigenetically regulated intra- and interchromosomal interactions. Nat Genet. 38, 1341-1347;
- J. Dostie, T. A. Richmond, R. A. Arnaout, R. R. Selzer, W. L. Lee, et. al.. (2006). Chromosome Conformation Capture Carbon Copy (5C): A massively parallel solution for mapping interactions between genomic elements. Genome Research. 16, 1299-1309;
- Tsung-Han S. Hsieh, Assaf Weiner, Bryan Lajoie, Job Dekker, Nir Friedman, Oliver J. Rando. (2015). Mapping Nucleosome Resolution Chromosome Folding in Yeast by Micro-C. Cell. 162, 108-119;
- F. Song, P. Chen, D. Sun, M. Wang, L. Dong, et. al.. (2014). Cryo-EM Study of the Chromatin Fiber Reveals a Double Helix Twisted by Tetranucleosomal Units. Science. 344, 376-380;
- Jon-Matthew Belton, Rachel Patton McCord, Johan Harmen Gibcus, Natalia Naumova, Ye Zhan, Job Dekker. (2012). Hi–C: A comprehensive technique to capture the conformation of genomes. Methods. 58, 268-276;
- Alexey A. Gavrilov, Arkadiy K. Golov, Sergey V. Razin. (2013). Actual Ligation Frequencies in the Chromosome Conformation Capture Procedure. PLoS ONE. 8, e60403;
- Takashi Nagano, Yaniv Lubling, Eitan Yaffe, Steven W Wingett, Wendy Dean, et. al.. (2015). Single-cell Hi-C for genome-wide detection of chromatin interactions that occur simultaneously in a single cell. Nat Protoc. 10, 1986-2003;
- Houda Belaghzal, Job Dekker, Johan H. Gibcus. (2017). Hi-C 2.0: An optimized Hi-C procedure for high-resolution genome-wide mapping of chromosome conformation. Methods. 123, 56-65;
- Ulahannan N., Pendleton M., Deshpande A., Schwenk S., Behr J.M., Dai X. et al. (2019). Nanopore sequencing of DNA concatemers reveals higher-order features of chromatin structure. bioRxiv;
- Giuseppina Maccarrone, Juan Jose Bonfiglio, Susana Silberstein, Christoph W. Turck, Daniel Martins-de-Souza. (2017). Characterization of a Protein Interactome by Co-Immunoprecipitation and Shotgun Mass Spectrometry. Multiplex Biomarker Techniques. 223-234;
- 12 методов в картинках: протеомика;
- H.-W. Rhee, P. Zou, N. D. Udeshi, J. D. Martell, V. K. Mootha, et. al.. (2013). Proteomic Mapping of Mitochondria in Living Cells via Spatially Restricted Enzymatic Tagging. Science. 339, 1328-1331;
- Kyle J. Roux, Dae In Kim, Manfred Raida, Brian Burke. (2012). A promiscuous biotin ligase fusion protein identifies proximal and interacting proteins in mammalian cells. Journal of Cell Biology. 196, 801-810;
- Furqan M. Fazal, Shuo Han, Kevin R. Parker, Pornchai Kaewsapsak, Jin Xu, et. al.. (2019). Atlas of Subcellular RNA Localization Revealed by APEX-Seq. Cell. 178, 473-490.e26;
- Joseph R. Tran, Danielle I. Paulson, James J. Moresco, Stephen A. Adam, John R. Yates, et. al.. (2021). An APEX2 proximity ligation method for mapping interactions with the nuclear lamina. Journal of Cell Biology. 220.
Схема генетического кода


Последовательность кодонов в части молекулы мРНК. Каждый кодон состоит из трёх нуклеотидов и соответствует единственной аминокислоте. Рибосома синтезирует белок согласно инструкции, записанной в мРНК, причём соответствие между кодоном и аминокислотой определяется генетическим кодом
Генети́ческий код (англ. Genetic code) — совокупность правил, согласно которым в живых клетках последовательность кодонов (генов и мРНК) переводится в последовательность аминокислот (белков). Собственно перевод (трансляцию) осуществляет рибосома, которая соединяет аминокислоты в цепочку согласно инструкции, записанной в кодонах мРНК. Соответствующие аминокислоты доставляются в рибосому молекулами тРНК. Генетический код всех живых организмов Земли един (имеются лишь незначительные вариации), что свидетельствует о наличии общего предка.
Правила генетического кода определяют, какой аминокислоте соответствует триплет (три подряд идущих нуклеотида) в мРНК. За редкими исключениями[1], каждому кодону соответствует только одна аминокислота. Конкретная аминокислота может кодироваться более чем одним кодоном, есть также кодоны, означающие начало и конец белка. Вариант генетического кода, который используется подавляющим большинством живых организмов, называют стандартным, или каноническим, генетическим кодом. Однако известно несколько десятков исключений из стандартного генетического кода, например, при трансляции в митохондриях используются несколько изменённые правила генетического кода.
Простейшим представлением генетического кода может служить таблица из 64 ячеек, в которой каждая ячейка соответствует одному из 64 возможных кодонов[2].
История изучения[править | править код]
Маршалл Ниренберг, начавший расшифровку генетического кода
Попытки понять, каким образом последовательность ДНК кодирует аминокислотную последовательность белков, начали предприниматься почти сразу же после того, как в 1953 году была установлена структура ДНК (двойная спираль). Георгий Гамов предположил, что кодоны должны состоять из трёх нуклеотидов, чтобы кодонов хватило для всех 20 аминокислот (всего же возможно 64 различных кодона из трёх нуклеотидов: на каждую из трёх позиций можно поставить один из четырёх нуклеотидов)[3].
В 1961 году триплетность генетического кода удалось подтвердить экспериментально. В том же году Маршалл Ниренберг и его коллега Генрих Маттеи[en] использовали бесклеточную систему для трансляции in vitro. В качестве матрицы был взят олигонуклеотид, состоящий из остатков урацила (UUUU…). Пептид, синтезированный с него, содержал только аминокислоту фенилаланин[4]. Так впервые было установлено значение кодона: кодон UUU кодирует фенилаланин. Дальнейшие правила соответствия между кодонами и аминокислотами были установлены в лаборатории Северо Очоа. Было показано, что полиадениновая РНК (ААА…) транслируется в полилизиновый пептид[5], а на матрице полицитозиновой РНК (ССС…) синтезируется пептид, состоящий только из остатков пролина[6]. Значение остальных кодонов было установлено при помощи разнообразных сополимеров в ходе экспериментов, проведённых в лаборатории Хара Гобинда Кораны. Вскоре после этого Роберт Холли установил структуру молекулы тРНК, которая служит посредником при трансляции. В 1968 году Ниренберг, Корана и Холли были удостоены Нобелевской премии по физиологии и медицине[7].
После установления правил генетического кода многие учёные занялись его искусственными преобразованиями[en]. Так, начиная с 2001 года в генетический код были внедрены 40 аминокислот, которые в природе не входят в состав белков. Для каждой аминокислоты создавались свой кодон и соответствующая аминоацил-тРНК-синтетаза. Искусственное расширение генетического кода и создание белков с новыми аминокислотами могут помочь глубже изучить структуру белковых молекул, а также получить искусственные белки с заданными свойствами[8][9]. Х. Мураками и М. Сисидо смогли превратить некоторые кодоны из трёхнуклеотидных в четырёх- и пятинуклеотидные. Стивен Бреннер получил 65-й кодон, который был функционален in vivo[10].
В 2015 году у бактерии Escherichia coli удалось изменить значение всех кодонов UGG с триптофана на тиенопиррол-аланин, не встречающийся в природе[11]. В 2016 году был получен первый полусинтетический организм — бактерия, геном которой содержал два искусственных азотистых основания (X и Y), сохраняющихся при делении[12][13]. В 2017 году исследователи из Южной Кореи заявили о создании мыши с расширенным генетическим кодом, способной синтезировать белки с аминокислотами, не встречающимися в природе[14].
Свойства[править | править код]
Рамки считывания в митохондриальной ДНК человека в области генов MT-ATP8[en] и MT-ATP6[en] (чёрным цветом показан участок между позициями 8525 и 8580). В направлении считывания 5′ → 3′ возможны три рамки считывания, начинающиеся с первой (+1), второй (+2) и третьей позиций (+3). Соответствие между кодонами (кодоны выделены квадратными скобками) определяется митохондриальным генетическим кодом позвоночных[en] в рамке +1 для MT-ATP8 (показана красным) или в рамке +3 для MT-ATP6 (показана синим). Ген MT-ATP8 заканчивается стоп-кодоном TAG (отмечен красной точкой) в рамке +1. Ген MT-ATP6 начинается с кодона ATG (кодирует аминокислоту метионин (М) и выделен синим кружком) в рамке +3
Выделяют следующие свойства генетического кода, также называемые принципами трансляции:
- триплетность — одной аминокислоте соответствует триплет из трех нуклеотидов РНК;
- неперекрываемость — один нуклеотид входит в состав единственного триплета;
- однозначность — одному триплету соответствует единственная аминокислота (за исключением стоп-кодонов);
- вырожденность — многим аминокислотам соответствует несколько триплетов (кроме метионина и триптофана);
- компактность — отсутствие знаков препинания внутри гена;
- наличие стоп-кодонов;
- универсальность — генетический код един для всех живых организмов;
- помехоустойчивость — большая часть однонуклеотидных замен внутри триплетов не приводят к изменению свойств аминокислоты.
Рамка считывания[править | править код]
Гены кодируются в направлении 5’→3′ нуклеотидной последовательности[15]. Рамка считывания определяется самым первым триплетом, с которого начинается трансляция. Последовательность неперекрывающихся кодонов, начинающуюся со старт-кодона и заканчивающуюся стоп-кодоном, называют открытой рамкой считывания. Например, последовательность 5′-AAATGAACG-3′ (см. рис.) при чтении с первого нуклеотида разбивается на кодоны AAA, TGA и ACG. Если чтение начинается со второго нуклеотида, то ей соответствуют кодоны AAT и GAA. Наконец, при чтении с третьего нуклеотида используются кодоны ATG и AAC. Таким образом, любую последовательность можно прочесть в направлении 5′ → 3′ тремя разными способами (с тремя разными рамками считывания), причём в каждом случае последовательность белкового продукта будет отличаться из-за распознавания рибосомой разных кодонов. Если учесть, что ДНК имеет двуцепочечную структуру, то возможны 6 рамок считывания: три на одной цепи и три на другой[16]. Однако считывание генов с ДНК не является случайным. Все другие рамки считывания в пределах одного гена обычно содержат многочисленные стоп-кодоны, чтобы быстро остановить и уменьшить метаболическую стоимость неправильного синтеза[17].
Старт- и стоп-кодоны[править | править код]
Трансляция информации с последовательности мРНК в аминокислотную последовательность начинается с так называемого старт-кодона — как правило, AUG, причём у эукариот он читается как метионин, а у бактерий — как формилметионин. Одного старт-кодона недостаточно для запуска трансляции; для него необходимы факторы инициации трансляции, а также особые элементы в соседних последовательностях, например, последовательность Шайна — Дальгарно у бактерий. У некоторых организмов в роли старт-кодонов используются кодоны GUG, который в норме кодирует валин, и UUG, который в стандартном коде соответствует лейцину[18].
После инициационного кодона трансляция продолжается через последовательное считывание кодонов и присоединение аминокислот друг к другу рибосомой до достижения сигнала к прекращению трансляции — стоп-кодона. Существуют три стоп-кодона, каждый из которых имеет своё название: UAG (янтарь), UGA (опал) и UAA (охра). Стоп-кодоны также называют терминаторными. В клетках нет тРНК, соответствующих стоп-кодонам, поэтому, когда рибосома доходит до стоп-кодона, вместо тРНК с ним взаимодействуют факторы терминации трансляции, которые гидролизуют последнюю тРНК от аминокислотной цепочки, а затем заставляют рибосому диссоциировать[19]. У бактерий в терминации трансляции принимают участие три белковых фактора[en]: RF-1, RF-2 и RF-3: RF-1 узнаёт кодоны UAG и UAA, а RF-2 распознаёт UAA и UGA. Фактор RF-3 выполняет вспомогательную работу. Трёхмерная структура RF-1 и RF-2 напоминает формой и распределением заряда тРНК и, таким образом, представляет собой пример молекулярной мимикрии[en][20]. У эукариот фактор терминации трансляции eRF1 распознаёт все три стоп-кодона. Зависимая от рибосомы ГТФаза eRF3, которую рассматривают как второй фактор терминации трансляции эукариот, помогает eRF1 в высвобождении с рибосомы готового полипептида[21][22][23].
Распределение стоп-кодонов в геноме организма неслучайно и может быть связано с GC-составом генома[24][25]. Например, у штамма E. coli K-12 в геноме имеется 2705 кодонов TAA (63 %), 1257 TGA (29 %) и 326 TAG (8 %) при GC-составе 50,8 %[26]. Масштабное исследование геномов разных видов бактерий показало, что доля кодона TAA положительно коррелирует с GC-составом, а доля TGA — отрицательно. Частота самого редко используемого стоп-кодона, TAG, не связана с GC-составом[27]. Сила стоп-кодонов также неодинакова. Спонтанный обрыв трансляции чаще всего происходит на кодоне UGA, а на UAA — реже всего[23].
Помимо собственно стоп-кодона, важнейшее значение для терминации трансляции имеет его окружение. Наиболее велика роль нуклеотида, расположенного сразу за стоп-кодоном (+4). Вероятно, нуклеотид +4 и другие нуклеотиды, следующие за ним, влияют на терминацию трансляции, обеспечивая сайты связывания факторов терминации трансляции. По этой причине некоторые исследователи предлагают рассматривать четырёхнуклеотидный стоп-сигнал вместо трёхнуклеотидного стоп-кодона. Нуклеотиды, расположенные выше стоп-кодонов, также влияют на трансляцию. Например, для дрожжей было показано, что аденин, располагающийся на 2 позиции выше первого нуклеотида стоп-кодона, стимулирует обрыв трансляции на стоп-кодоне UAG (возможно, и на остальных кодонах)[23].
Иногда стоп-кодоны выступают в роли смысловых. Например, кодон UGA кодирует нестандартную аминокислоту селеноцистеин, если рядом с ним в транскрипте находится так называемый SECIS-элемент[28]. Стоп-кодон UAG может кодировать другую нестандартную аминокислоту — пирролизин. Иногда стоп-кодон распознаётся как смысловой при мутациях, затрагивающих тРНК. Наиболее часто это явление наблюдается у вирусов, но оно также описано у бактерий, дрожжей, дрозофилы и человека, у которых играет регуляторную роль[29][30].
Генетический код и мутации[править | править код]
В ходе репликации ДНК изредка возникают ошибки при синтезе дочерней цепи. Эти ошибки, называемые мутациями, могут повлиять на фенотип организма, особенно если они затрагивают кодирующую область гена. Ошибки происходят с частотой 1 на каждые 10—100 миллионов пар оснований (п. о.), так как ДНК-полимеразы могут эффективно исправлять свои ошибки[31][32].
Под точечными мутациями понимают единичные замены одного азотистого основания. Если новое основание относится к тому же классу, что и исходное (оба пурины или оба пиримидины), то мутацию относят к транзициям. Если происходит замена пурина на пиримидин или пиримидина на пурин, то говорят о трансверсиях. Транзиции встречаются чаще трансверсий[33]. Примерами точечных мутаций являются миссенс- и нонсенс-мутации. Они могут вызывать такие заболевания, как серповидноклеточная анемия и талассемия соответственно[34][35]. Клинически значимые миссенс-мутации приводят к замене аминокислотного остатка на остаток с другими физико-химическими свойствами, а нонсенс-мутации заключаются в появлении преждевременного стоп-кодона[16].
Мутации, при которых нарушается правильная рамка считывания из-за вставок и делеций (в совокупности они называются инделами[en]), содержащих некратное трём число нуклеотидов, называются мутациями сдвига рамки считывания. При этих мутациях белковый продукт получается совершенно иной, чем в диком типе. Как правило, при сдвигах рамки считывания появляются преждевременные стоп-кодоны, которые вызывают образование усечённых белков[36]. Поскольку эти мутации значительно нарушают функцию белка, они довольно редко закрепляются отбором: нередко отсутствие белка приводит к гибели организма ещё до рождения[37]. Мутации сдвига рамки считывания связаны с такими заболеваниями, как болезнь Тея — Сакса[38].
Хотя подавляющее число мутаций вредно или нейтрально[en], некоторые оказываются полезными[39]. Они могут давать организму лучшую приспособленность по сравнению с диким типом к определённым условиям окружающей среды или дают ему возможность размножаться быстрее особей дикого типа. В этом случае мутация будет постепенно распространяться в популяции в ходе нейтрального отбора[40]. Вирусы, геномы которых представлены РНК, мутируют очень быстро[41], что нередко приносит им пользу, потому что иммунная система, эффективно распознающая одни варианты вирусных антигенов, оказывается бессильна против слегка изменённых[42]. В больших популяциях организмов, размножающихся бесполым путём, например, E. coli, одновременно может происходить несколько полезных мутаций. Этот феномен получил название клональной интерференции[en] и вызывает конкуренцию между мутациями[43].
Вырожденность[править | править код]
Способность разных кодонов кодировать одну аминокислоту называется вырожденностью кода. Впервые генетический код назвали вырожденным[en] Ниренберг и Бернфилд. Однако, несмотря на вырожденность, в генетическом коде полностью отсутствует двусмысленность. Например, кодоны GAA и GAG оба кодируют глутамат, но ни один из них не кодирует одновременно ещё какую-то аминокислоту. Кодоны, соответствующие одной аминокислоте, могут различаться по любым позициям, однако чаще всего две первые позиции у таких кодонов совпадают, а различается только последняя. Благодаря этому мутация, затронувшая третью позицию кодона, скорее всего, не скажется на белковом продукте[44].
Эта особенность может быть объяснена гипотезой неоднозначной пары оснований, предложенной Франсисом Криком. Согласно этой гипотезе, третий нуклеотид в кодоне ДНК может быть не полностью комплементарен антикодону тРНК для компенсации несоответствия числа типов тРНК числу кодонов[45][46].
Кодоны близких по физико-химическим свойствам аминокислот также нередко похожи, благодаря чему мутации не приводят к значительным нарушениям белковой структуры. Так, кодоны NUN (N — любой нуклеотид) обычно кодируют гидрофобные аминокислоты. NCN кодируют маленькие аминокислоты с умеренной гидрофобностью, а NAN кодируют гидрофильные аминокислоты среднего размера. Генетический код устроен настолько оптимально с точки зрения гидрофобности, что математический анализ при помощи сингулярного разложения 12 переменных (4 нуклеотида на 3 позиции) даёт значимую корреляцию (0,95) для предсказания гидрофобности аминокислоты по её кодону[47]. На восемь аминокислот мутации по третьим позициям не влияют вообще, а мутации по второй позиции, как правило, приводят к замене на аминокислоту с совершенно другими физико-химическими свойствами. Однако наибольшее влияние на белковый продукт имеют мутации по первым позициям. Так, мутации, приводящие к замене заряженной аминокислоты на аминокислоту с противоположным зарядом, могут затрагивать только первую позицию, а вторую — никогда. Такая замена заряда, вероятнее всего, окажет сильный эффект на структуру белка[48].
Стандартный генетический код[править | править код]
В таблице ниже представлен генетический код, общий для большинства про- и эукариот. В таблице приведены все 64 кодона и указаны соответствующие аминокислоты. Порядок оснований — от 5′- к 3′-концу мРНК. Приведены трёхбуквенные и однобуквенные обозначения аминокислот.
| неполярный | полярный | основный | кислотный | (стоп-кодон) |
| 1-е основание |
2-е основание | 3-е основание |
|||||||
|---|---|---|---|---|---|---|---|---|---|
| U | C | A | G | ||||||
| U | UUU | (Phe/F) Фенилаланин | UCU | (Ser/S) Серин | UAU | (Tyr/Y) Тирозин | UGU | (Cys/C) Цистеин | U |
| UUC | UCC | UAC | UGC | C | |||||
| UUA | (Leu/L) Лейцин | UCA | UAA | Стоп (Охра) | UGA | Стоп (Опал) | A | ||
| UUG | UCG | UAG | Стоп (Янтарь) | UGG | (Trp/W) Триптофан | G | |||
| C | CUU | CCU | (Pro/P) Пролин | CAU | (His/H) Гистидин | CGU | (Arg/R) Аргинин | U | |
| CUC | CCC | CAC | CGC | C | |||||
| CUA | CCA | CAA | (Gln/Q) Глутамин | CGA | A | ||||
| CUG | CCG | CAG | CGG | G | |||||
| A | AUU | (Ile/I) Изолейцин | ACU | (Thr/T) Треонин | AAU | (Asn/N) Аспарагин | AGU | (Ser/S) Серин | U |
| AUC | ACC | AAC | AGC | C | |||||
| AUA | ACA | AAA | (Lys/K) Лизин | AGA | (Arg/R) Аргинин | A | |||
| AUG[A] | (Met/M) Метионин | ACG | AAG | AGG | G | ||||
| G | GUU | (Val/V) Валин | GCU | (Ala/A) Аланин | GAU | (Asp/D) Аспарагиновая кислота | GGU | (Gly/G) Глицин | U |
| GUC | GCC | GAC | GGC | C | |||||
| GUA | GCA | GAA | (Glu/E) Глутаминовая кислота | GGA | A | ||||
| GUG | GCG | GAG | GGG | G |
- A Кодон AUG кодирует метионин и одновременно является сайтом инициации трансляции: первый кодон AUG в кодирующей области мРНК служит началом синтеза белка[49].
| Ala/A | GCU, GCC, GCA, GCG | Leu/L | UUA, UUG, CUU, CUC, CUA, CUG |
|---|---|---|---|
| Arg/R | CGU, CGC, CGA, CGG, AGA, AGG | Lys/K | AAA, AAG |
| Asn/N | AAU, AAC | Met/M | AUG |
| Asp/D | GAU, GAC | Phe/F | UUU, UUC |
| Cys/C | UGU, UGC | Pro/P | CCU, CCC, CCA, CCG |
| Gln/Q | CAA, CAG | Ser/S | UCU, UCC, UCA, UCG, AGU, AGC |
| Glu/E | GAA, GAG | Thr/T | ACU, ACC, ACA, ACG |
| Gly/G | GGU, GGC, GGA, GGG | Trp/W | UGG |
| His/H | CAU, CAC | Tyr/Y | UAU, UAC |
| Ile/I | AUU, AUC, AUA | Val/V | GUU, GUC, GUA, GUG |
| START | AUG | STOP | UAG, UGA, UAA |
Альтернативные генетические коды[править | править код]
Нестандартные аминокислоты[править | править код]
В некоторых белках нестандартные аминокислоты кодируются стоп-кодонами в зависимости от наличия особой сигнальной последовательности в мРНК. Например, стоп-кодон UGA может кодировать селеноцистеин, а UAG — пирролизин. Селеноцистеин и пирролизин рассматривают как 21-ю и 22-ю протеиногенную аминокислоту соответственно. В отличие от селеноцистеина, у пирролизина есть собственная аминоацил-тРНК-синтетаза[50]. Хотя обычно генетический код, используемый клетками одного организма, фиксирован, архея Acetohalobium arabaticum[en] может переключаться с 20-аминокислотного кода на 21-аминокислотный (включая пирролизин) при разных условиях роста[51].
Вариации[править | править код]
Схема митохондриального генетического кода Globobulimina pseudospinescens. Показаны 64 кодона (слева направо), предсказанные отклонения от стандартного генетического кода изображены красным. Красной линией показаны стоп-кодоны. Высота буквы, соответствующей аминокислоте, пропорциональна частоте соответствия этой аминокислоты данному кодону
Существование отклонений от стандартного генетического кода предсказывалось ещё в 1970-х[52]. Первое отклонение было описано в 1979 году в митохондриях человека[53]. Впоследствии было описано ещё несколько альтернативных генетических кодов, слегка отличающихся от стандартного, в том числе альтернативные митохондриальные коды[54].
Например, у бактерий рода Mycoplasma стоп-кодон UGA кодирует триптофан, а у дрожжей из так называемой «CTG-клады» (в том числе патогенного вида Candida albicans) кодон CUG кодирует серин, а не лейцин, как в стандартном генетическом коде[55][56][57]. Поскольку вирусы используют тот же генетический код, что и клетки-хозяева, отклонения от стандартного генетического кода могут нарушить размножение вирусов[58]. Впрочем, некоторые вирусы, например, вирусы рода Totivirus[en], используют тот же альтернативный генетический код, что и организм-хозяин[59].
У бактерий и архей GUG и UUG нередко выступают старт-кодонами[60]. Некоторые отклонения от стандартного генетического кода есть и в ядерном геноме человека: так, в 4 % мРНК фермента малатдегидрогеназы один из стоп-кодонов кодирует триптофан или аргинин[61]. Значение стоп-кодона зависит от его окружения[30]. Отклонения в генетическом коде организма можно обнаружить, если найти в его геноме очень консервативные гены и сравнить их кодоны с соответствующими аминокислотами гомологичных белков близкородственных организмов. По такому принципу работает программа FACIL, которая рассчитывает, с какой частотой каждый кодон соответствует той или иной аминокислоте, а также определяет поддержку стоп-кодона и представляет результат в виде логотипа (LOGO)[62]. Впрочем, несмотря на все перечисленные отличия, генетические коды, используемые всеми организмами, в общих чертах схожи[63].
В таблице ниже перечислены известные на данный момент нестандартные генетические коды[64][65]. Насчитывают 23 нестандартных генетических кода, причём наиболее частым отличием от стандартного генетического кода является превращение стоп-кодона UGA в смысловой, кодирующий триптофан[66].
Предпочтение кодонов[править | править код]
В геномах многих организмов наблюдается так называемое предпочтение кодонов, то есть частота встречаемости всех синонимичных кодонов, соответствующих определённой аминокислоте, не равна и для одних кодонов выше, чем для других[67][68]. Эволюционные основы возникновения предпочтения кодонов неясны. Согласно одной гипотезе, реже встречаются те кодоны, которые наиболее часто мутируют. Другая гипотеза утверждает, что предпочтение кодонов регулируется естественным отбором в пользу тех, которые обеспечивают наибольшую эффективность и точность экспрессии генов[69][70].
Предпочтение кодонов в значительной мере связано с GC-составом генома, и в некоторых случаях по GC-составу можно даже предсказать частоту использования кодонов[71]. С функциональной точки зрения предпочтение кодонов связано с эффективностью и точностью трансляции и, следовательно, уровнем экспрессии гена[72][73].
Происхождение[править | править код]
В настоящее время наиболее общепринятой гипотезой о происхождении жизни на Земле является гипотеза мира РНК. Любая модель возникновения генетического кода использует гипотезу о передаче основных функций от РНК-ферментов (рибозимов) к белковым ферментам. Как и предполагает гипотеза мира РНК, тРНК появились раньше аминоацил-тРНК-синтетаз, поэтому эти ферменты не могли оказать влияние на свойства тРНК[74].
Генетический код последнего универсального общего предка (LUCA) был основан, вероятнее всего, на ДНК, а не РНК[75]. Генетический код состоял из трёхнуклеотидных кодонов, и всего было возможно 64 различных кодона. Поскольку для построения белков использовалось только 20 аминокислот, некоторые аминокислоты кодировались несколькими кодонами[76][77][78][79].
Если бы соответствие между кодонами и аминокислотами было случайным, в природе существовало бы 1,5 × 1084 генетических кодов[80]. Это число получилось в результате расчёта количества способов, которыми можно 21 предмет (20 кодонов, кодирующих аминокислоты, и один стоп-кодон) разложить в 64 корзины, так, чтобы каждый предмет был использован по крайней мере единожды[81]. Однако соответствия кодонов и аминокислот неслучайны[82]. Аминокислоты, которые имеют общий путь биосинтеза, как правило, имеют общую первую позицию кодонов. Этот факт может быть пережитком раннего, более простого генетического кода, который содержал меньше аминокислот, чем современный, и постепенно включил в свой состав все 20 аминокислот[83]. Кодоны аминокислот со схожими физико-химическими свойствами также, как правило, похожи, что смягчает последствия точечных мутаций и нарушений трансляции[84][85].
Поскольку генетический код неслучаен, правдоподобная гипотеза о его возникновении должна объяснять такие свойства стандартного генетического кода, как отсутствие кодонов для D-аминокислот, включение всего лишь 20 аминокислот из возможных 64, ограничение синонимичных замен третьей позицией кодонов, функционирование в качестве стоп-кодонов именно кодонов UAG, UGA и UAA[86]. Существуют три основные гипотезы происхождения генетического кода. Каждая из них представлена множеством моделей, многие модели гибридны[87].
- Замороженная случайность: генетический код возник случайно и в таком виде закрепился. Возможно, древние рибозимы, подобные современным тРНК, имели разное сродство к аминокислотам, причём кодоны из разных частей одного и того же рибозима могли обладать наибольшим сродством к разным аминокислотам. После того, как появились первые функциональные пептиды, любое изменение генетического кода было бы летальным, поэтому он оказался «заморожен»[88].
- Стереохимическое сродство: генетический код определяется высоким сродством каждой аминокислоты с соответствующими ей кодонами и антикодонами. Сродство аминокислоты и антикодона означает, что предковым тРНК соответствовали те аминокислоты, с которыми они связывались с наибольшим сродством. В ходе эволюции соответствие антикодонов и аминокислот заменилось соответствием аминоацил-тРНК-синтетаз и аминокислот[89].
- Оптимальность: генетический код продолжал некоторое время изменяться после своего появления, поэтому современный код обеспечивает максимальную приспособленность и минимизирует последствия мутаций, то есть является лучшим из возможных генетических кодов[87].
Примечания[править | править код]
- ↑ Turanov A. A., Lobanov A. V., Fomenko D. E., Morrison H. G., Sogin M. L., Klobutcher L. A., Hatfield D. L., Gladyshev V. N. Genetic code supports targeted insertion of two amino acids by one codon. (англ.) // Science (New York, N.Y.). — 2009. — 9 January (vol. 323, no. 5911). — P. 259—261. — doi:10.1126/science.1164748. — PMID 19131629. [исправить]
- ↑ Shu J. J. A new integrated symmetrical table for genetic codes. (англ.) // Bio Systems. — 2017. — January (vol. 151). — P. 21—26. — doi:10.1016/j.biosystems.2016.11.004. — PMID 27887904. [исправить]
- ↑ Crick, Francis. [[1] в «Книгах Google» Chapter 8: The genetic code] // What Mad Pursuit: A Personal View of Scientific Discovery (англ.). — Basic Books, 1990. — P. 89—101. — ISBN 978-0-465-09138-6.
- ↑ NIRENBERG MW, MATTHAEI JH. The dependence of cell-free protein synthesis in E. coli upon naturally occurring or synthetic polyribonucleotides. (англ.) // Proceedings Of The National Academy Of Sciences Of The United States Of America. — 1961. — 15 October (vol. 47). — P. 1588—1602. — PMID 14479932. [исправить]
- ↑ GARDNER RS, WAHBA AJ, BASILIO C, MILLER RS, LENGYEL P, SPEYER JF. Synthetic polynucleotides and the amino acid code. VII. (англ.) // Proceedings Of The National Academy Of Sciences Of The United States Of America. — 1962. — 15 December (vol. 48). — P. 2087—2094. — PMID 13946552. [исправить]
- ↑ WAHBA AJ, GARDNER RS, BASILIO C, MILLER RS, SPEYER JF, LENGYEL P. Synthetic polynucleotides and the amino acid code. VIII. (англ.) // Proceedings Of The National Academy Of Sciences Of The United States Of America. — 1963. — 15 January (vol. 49). — P. 116—122. — PMID 13998282. [исправить]
- ↑ The Royal Swedish Academy of Science (1959). The Nobel Prize in Physiology or Medicine 1959. Пресс-релиз. Архивировано из первоисточника 26 декабря 2018. Проверено 2010-02-27. “The Nobel Prize in Physiology or Medicine 1959 was awarded jointly to Severo Ochoa and Arthur Kornberg ‘for their discovery of the mechanisms in the biological synthesis of ribonucleic acid and deoxyribonucleic acid’.”
- ↑ Xie J., Schultz P. G. Adding amino acids to the genetic repertoire. (англ.) // Current Opinion In Chemical Biology. — 2005. — December (vol. 9, no. 6). — P. 548—554. — doi:10.1016/j.cbpa.2005.10.011. — PMID 16260173. [исправить]
- ↑ Wang Q., Parrish A. R., Wang L. Expanding the genetic code for biological studies. (англ.) // Chemistry & Biology. — 2009. — 27 March (vol. 16, no. 3). — P. 323—336. — doi:10.1016/j.chembiol.2009.03.001. — PMID 19318213. [исправить]
- ↑ Simon, Matthew. [[2] в «Книгах Google» Emergent Computation: Emphasizing Bioinformatics] (англ.). — Springer Science & Business Media, 2005. — P. 105—106. — ISBN 978-0-387-22046-8.
- ↑ Hoesl M. G., Oehm S., Durkin P., Darmon E., Peil L., Aerni H. R., Rappsilber J., Rinehart J., Leach D., Söll D., Budisa N. Chemical Evolution of a Bacterial Proteome. (англ.) // Angewandte Chemie (International Ed. In English). — 2015. — 17 August (vol. 54, no. 34). — P. 10030—10034. — doi:10.1002/anie.201502868. — PMID 26136259. [исправить]
- ↑ First stable semisynthetic organism created | KurzweilAI. www.kurzweilai.net (3 февраля 2017). Дата обращения: 9 февраля 2017. Архивировано 10 февраля 2017 года.
- ↑ Zhang Y., Lamb B. M., Feldman A. W., Zhou A. X., Lavergne T., Li L., Romesberg F. E. A semisynthetic organism engineered for the stable expansion of the genetic alphabet. (англ.) // Proceedings Of The National Academy Of Sciences Of The United States Of America. — 2017. — 7 February (vol. 114, no. 6). — P. 1317—1322. — doi:10.1073/pnas.1616443114. — PMID 28115716. [исправить]
- ↑ Han S., Yang A., Lee S., Lee H. W., Park C. B., Park H. S. Expanding the genetic code of Mus musculus. (англ.) // Nature Communications. — 2017. — 21 February (vol. 8). — P. 14568—14568. — doi:10.1038/ncomms14568. — PMID 28220771. [исправить]
- ↑ Cartwright R. A., Graur D. The multiple personalities of Watson and Crick strands. (англ.) // Biology Direct. — 2011. — 8 February (vol. 6). — P. 7. — doi:10.1186/1745-6150-6-7. — PMID 21303550. [исправить]
- ↑ 1 2 King, Robert C.; Mulligan, Pamela; Stansfield, William. [[3] в «Книгах Google» A Dictionary of Genetics] (неопр.). — OUP USA, 2013. — С. 608. — ISBN 978-0-19-976644-4. Архивная копия от 8 апреля 2022 на Wayback Machine
- ↑ Tse H., Cai J. J., Tsoi H. W., Lam E. P., Yuen K. Y. Natural selection retains overrepresented out-of-frame stop codons against frameshift peptides in prokaryotes. (англ.) // BMC Genomics. — 2010. — 9 September (vol. 11). — P. 491. — doi:10.1186/1471-2164-11-491. — PMID 20828396. [исправить]
- ↑ Touriol C., Bornes S., Bonnal S., Audigier S., Prats H., Prats A. C., Vagner S. Generation of protein isoform diversity by alternative initiation of translation at non-AUG codons. (англ.) // Biology Of The Cell. — 2003. — May (vol. 95, no. 3-4). — P. 169—178. — PMID 12867081. [исправить]
- ↑ Maloy S. How nonsense mutations got their names. Microbial Genetics Course. San Diego State University (29 ноября 2003). Дата обращения: 10 марта 2010. Архивировано 16 ноября 2014 года.
- ↑ Альбертс и др., 2013, с. 586.
- ↑ Protacio R. U., Storey A. J., Davidson M. K., Wahls W. P. Nonsense codon suppression in fission yeast due to mutations of tRNA(Ser.11) and translation release factor Sup35 (eRF3). (англ.) // Current Genetics. — 2015. — May (vol. 61, no. 2). — P. 165—173. — doi:10.1007/s00294-014-0465-7. — PMID 25519804. [исправить]
- ↑ Schueren F., Thoms S. Functional Translational Readthrough: A Systems Biology Perspective. (англ.) // PLoS Genetics. — 2016. — August (vol. 12, no. 8). — P. e1006196—1006196. — doi:10.1371/journal.pgen.1006196. — PMID 27490485. [исправить]
- ↑ 1 2 3 Dabrowski Maciej, Bukowy-Bieryllo Zuzanna, Zietkiewicz Ewa. Translational readthrough potential of natural termination codons in eucaryotes – The impact of RNA sequence (англ.) // RNA Biology. — 2015. — 15 July (vol. 12, no. 9). — P. 950—958. — ISSN 1547-6286. — doi:10.1080/15476286.2015.1068497. [исправить]
- ↑ Povolotskaya I. S., Kondrashov F. A., Ledda A., Vlasov P. K. Stop codons in bacteria are not selectively equivalent. (англ.) // Biology Direct. — 2012. — 13 September (vol. 7). — P. 30. — doi:10.1186/1745-6150-7-30. — PMID 22974057. [исправить]
- ↑ Korkmaz G., Holm M., Wiens T., Sanyal S. Comprehensive analysis of stop codon usage in bacteria and its correlation with release factor abundance. (англ.) // The Journal Of Biological Chemistry. — 2014. — 31 October (vol. 289, no. 44). — P. 30334—30342. — doi:10.1074/jbc.M114.606632. — PMID 25217634. [исправить]
- ↑ Escherichia coli str. K-12 substr. MG1655, complete genome [Genbank Accession Number: U00096]. GenBank. NCBI. Дата обращения: 27 января 2013. Архивировано 16 января 2013 года.
- ↑ Wong T. Y., Fernandes S., Sankhon N., Leong P. P., Kuo J., Liu J. K. Role of premature stop codons in bacterial evolution. (англ.) // Journal Of Bacteriology. — 2008. — October (vol. 190, no. 20). — P. 6718—6725. — doi:10.1128/JB.00682-08. — PMID 18708500. [исправить]
- ↑ Papp L. V., Lu J., Holmgren A., Khanna K. K. From selenium to selenoproteins: synthesis, identity, and their role in human health. (англ.) // Antioxidants & Redox Signaling. — 2007. — July (vol. 9, no. 7). — P. 775—806. — doi:10.1089/ars.2007.1528. — PMID 17508906. [исправить]
- ↑ Namy O., Rousset J. P., Napthine S., Brierley I. Reprogrammed genetic decoding in cellular gene expression. (англ.) // Molecular Cell. — 2004. — 30 January (vol. 13, no. 2). — P. 157—168. — PMID 14759362. [исправить]
- ↑ 1 2 Schueren F., Lingner T., George R., Hofhuis J., Dickel C., Gärtner J., Thoms S. Peroxisomal lactate dehydrogenase is generated by translational readthrough in mammals. (англ.) // ELife. — 2014. — 23 September (vol. 3). — P. e03640—03640. — doi:10.7554/eLife.03640. — PMID 25247702. [исправить]
- ↑ Spontaneous mutations // An Introduction to Genetic Analysis (неопр.) / Griffiths, Anthony J. F.; Miller, Jeffrey H.; Suzuki, David T.; Lewontin, Richard C.; Gelbart. — 7th. — New York: W. H. Freeman (англ.) (рус., 2000. — ISBN 978-0-7167-3520-5.
- ↑ Freisinger E., Grollman A. P., Miller H., Kisker C. Lesion (in)tolerance reveals insights into DNA replication fidelity. (англ.) // The EMBO Journal. — 2004. — 7 April (vol. 23, no. 7). — P. 1494—1505. — doi:10.1038/sj.emboj.7600158. — PMID 15057282. [исправить]
- ↑ Кребс, Голдштейн, Килпатрик, 2017, с. 31.
- ↑ Chang J. C., Kan Y. W. beta 0 thalassemia, a nonsense mutation in man. (англ.) // Proceedings Of The National Academy Of Sciences Of The United States Of America. — 1979. — June (vol. 76, no. 6). — P. 2886—2889. — PMID 88735. [исправить]
- ↑ Boillée S., Vande Velde C., Cleveland D. W. ALS: a disease of motor neurons and their nonneuronal neighbors. (англ.) // Neuron. — 2006. — 5 October (vol. 52, no. 1). — P. 39—59. — doi:10.1016/j.neuron.2006.09.018. — PMID 17015226. [исправить]
- ↑ Isbrandt D., Hopwood J. J., von Figura K., Peters C. Two novel frameshift mutations causing premature stop codons in a patient with the severe form of Maroteaux-Lamy syndrome. (англ.) // Human Mutation. — 1996. — Vol. 7, no. 4. — P. 361—363. — doi:10.1002/(SICI)1098-1004(1996)7:4<361::AID-HUMU12>3.0.CO;2-0. — PMID 8723688. [исправить]
- ↑ Crow J. F. How much do we know about spontaneous human mutation rates? (англ.) // Environmental And Molecular Mutagenesis. — 1993. — Vol. 21, no. 2. — P. 122—129. — PMID 8444142. [исправить]
- ↑ Lewis, Ricki. Human Genetics: Concepts and Applications (англ.). — 6th. — Boston, Mass: McGraw-Hill Education, 2005. — P. 227—228. — ISBN 978-0-07-111156-0.
- ↑ Sawyer S. A., Parsch J., Zhang Z., Hartl D. L. Prevalence of positive selection among nearly neutral amino acid replacements in Drosophila. (англ.) // Proceedings Of The National Academy Of Sciences Of The United States Of America. — 2007. — 17 April (vol. 104, no. 16). — P. 6504—6510. — doi:10.1073/pnas.0701572104. — PMID 17409186. [исправить]
- ↑ Bridges K. R. Malaria and the Red Cell (неопр.) // Harvard. — 2002. Архивировано 27 ноября 2011 года. Архивированная копия. Дата обращения: 2 сентября 2018. Архивировано из оригинала 27 ноября 2011 года.
- ↑ Drake J. W., Holland J. J. Mutation rates among RNA viruses. (англ.) // Proceedings Of The National Academy Of Sciences Of The United States Of America. — 1999. — 23 November (vol. 96, no. 24). — P. 13910—13913. — PMID 10570172. [исправить]
- ↑ Holland J., Spindler K., Horodyski F., Grabau E., Nichol S., VandePol S. Rapid evolution of RNA genomes. (англ.) // Science (New York, N.Y.). — 1982. — 26 March (vol. 215, no. 4540). — P. 1577—1585. — PMID 7041255. [исправить]
- ↑ de Visser J. A., Rozen D. E. Clonal interference and the periodic selection of new beneficial mutations in Escherichia coli. (англ.) // Genetics. — 2006. — April (vol. 172, no. 4). — P. 2093—2100. — doi:10.1534/genetics.105.052373. — PMID 16489229. [исправить]
- ↑ Кребс, Голдштейн, Килпатрик, 2017, с. 689—691.
- ↑ Biochemistry (неопр.) / Mathews, Christopher K.; Van Holde, K.E.; Appling, Dean; Anthony-Cahill, Spencer. — 4th. — Toronto: Prentice Hall, 2012. — С. 1181. — ISBN 978-0-13-800464-4.
- ↑ Voet, Donald; Voet, Judith. Biochemistry (неопр.). — 4th. — Hoboken, NJ: John Wiley & Sons, 2011. — С. 1360—1361. — ISBN 9780470570951.
- ↑ Michel-Beyerle, Maria Elisabeth. [[4] в «Книгах Google» Reaction centers of photosynthetic bacteria: Feldafing-II-Meeting] (англ.). — Springer-Verlag, 1990. — ISBN 978-3-540-53420-4. Архивная копия от 8 апреля 2022 на Wayback Machine
- ↑ Fricke M., Gerst R., Ibrahim B., Niepmann M., Marz M. Global importance of RNA secondary structures in protein coding sequences. (англ.) // Bioinformatics. — 2018. — 7 August. — doi:10.1093/bioinformatics/bty678. — PMID 30101307. [исправить]
- ↑ Nakamoto T. Evolution and the universality of the mechanism of initiation of protein synthesis. (англ.) // Gene. — 2009. — 1 March (vol. 432, no. 1-2). — P. 1—6. — doi:10.1016/j.gene.2008.11.001. — PMID 19056476. [исправить]
- ↑ Krzycki J. A. The direct genetic encoding of pyrrolysine. (англ.) // Current Opinion In Microbiology. — 2005. — December (vol. 8, no. 6). — P. 706—712. — doi:10.1016/j.mib.2005.10.009. — PMID 16256420. [исправить]
- ↑ Prat L., Heinemann I. U., Aerni H. R., Rinehart J., O’Donoghue P., Söll D. Carbon source-dependent expansion of the genetic code in bacteria. (англ.) // Proceedings Of The National Academy Of Sciences Of The United States Of America. — 2012. — 18 December (vol. 109, no. 51). — P. 21070—21075. — doi:10.1073/pnas.1218613110. — PMID 23185002. [исправить]
- ↑
Crick F.H.C., Orgel L.E. Directed panspermia (англ.) // Icarus. — 1973. — July (vol. 19, no. 3). — P. 341—346. — ISSN 0019-1035. — doi:10.1016/0019-1035(73)90110-3. [исправить] - ↑
Barrell B. G., Bankier A. T., Drouin J. A different genetic code in human mitochondria. (англ.) // Nature. — 1979. — 8 November (vol. 282, no. 5735). — P. 189—194. — PMID 226894. [исправить] - ↑ Jukes T. H., Osawa S. The genetic code in mitochondria and chloroplasts. (англ.) // Experientia. — 1990. — 1 December (vol. 46, no. 11-12). — P. 1117—1126. — PMID 2253709. [исправить]
- ↑
Fitzpatrick D. A., Logue M. E., Stajich J. E., Butler G. A fungal phylogeny based on 42 complete genomes derived from supertree and combined gene analysis. (англ.) // BMC Evolutionary Biology. — 2006. — 22 November (vol. 6). — P. 99—99. — doi:10.1186/1471-2148-6-99. — PMID 17121679. [исправить] - ↑ Santos M. A., Tuite M. F. The CUG codon is decoded in vivo as serine and not leucine in Candida albicans. (англ.) // Nucleic Acids Research. — 1995. — 11 May (vol. 23, no. 9). — P. 1481—1486. — PMID 7784200. [исправить]
- ↑ Butler G., Rasmussen M. D., Lin M. F., Santos M. A., Sakthikumar S., Munro C. A., Rheinbay E., Grabherr M., Forche A., Reedy J. L., Agrafioti I., Arnaud M. B., Bates S., Brown A. J., Brunke S., Costanzo M. C., Fitzpatrick D. A., de Groot P. W., Harris D., Hoyer L. L., Hube B., Klis F. M., Kodira C., Lennard N., Logue M. E., Martin R., Neiman A. M., Nikolaou E., Quail M. A., Quinn J., Santos M. C., Schmitzberger F. F., Sherlock G., Shah P., Silverstein K. A., Skrzypek M. S., Soll D., Staggs R., Stansfield I., Stumpf M. P., Sudbery P. E., Srikantha T., Zeng Q., Berman J., Berriman M., Heitman J., Gow N. A., Lorenz M. C., Birren B. W., Kellis M., Cuomo C. A. Evolution of pathogenicity and sexual reproduction in eight Candida genomes. (англ.) // Nature. — 2009. — 4 June (vol. 459, no. 7247). — P. 657—662. — doi:10.1038/nature08064. — PMID 19465905. [исправить]
- ↑ Witzany G. Crucial steps to life: From chemical reactions to code using agents. (англ.) // Bio Systems. — 2016. — February (vol. 140). — P. 49—57. — doi:10.1016/j.biosystems.2015.12.007. — PMID 26723230. [исправить]
- ↑ Taylor D. J., Ballinger M. J., Bowman S. M., Bruenn J. A. Virus-host co-evolution under a modified nuclear genetic code. (англ.) // PeerJ. — 2013. — Vol. 1. — P. e50—50. — doi:10.7717/peerj.50. — PMID 23638388. [исправить]
- ↑ Elzanowski A, Ostell J. The Genetic Codes. National Center for Biotechnology Information (NCBI) (7 апреля 2008). Дата обращения: 10 марта 2010. Архивировано 20 августа 2016 года.
- ↑ Hofhuis J., Schueren F., Nötzel C., Lingner T., Gärtner J., Jahn O., Thoms S. The functional readthrough extension of malate dehydrogenase reveals a modification of the genetic code. (англ.) // Open Biology. — 2016. — November (vol. 6, no. 11). — doi:10.1098/rsob.160246. — PMID 27881739. [исправить]
- ↑ Dutilh B. E., Jurgelenaite R., Szklarczyk R., van Hijum S. A., Harhangi H. R., Schmid M., de Wild B., Françoijs K. J., Stunnenberg H. G., Strous M., Jetten M. S., Op den Camp H. J., Huynen M. A. FACIL: Fast and Accurate Genetic Code Inference and Logo. (англ.) // Bioinformatics. — 2011. — 15 July (vol. 27, no. 14). — P. 1929—1933. — doi:10.1093/bioinformatics/btr316. — PMID 21653513. [исправить]
- ↑ Kubyshkin V., Acevedo-Rocha C. G., Budisa N. On universal coding events in protein biogenesis. (англ.) // Bio Systems. — 2018. — February (vol. 164). — P. 16—25. — doi:10.1016/j.biosystems.2017.10.004. — PMID 29030023. [исправить]
- ↑ Elzanowski, Andrzej; Jim Ostell.: The Genetic Codes. National Center for Biotechnology Information (7 июля 2010). Дата обращения: 6 мая 2013. Архивировано 18 мая 2013 года.
- ↑ Watanabe Kimitsuna, Suzuki Tsutomu. Genetic Code and its Variants (англ.) // Encyclopedia of Life Sciences. — 2001. — 19 April. — ISBN 047001590X. — doi:10.1038/npg.els.0000810. [исправить]
- ↑ Koonin E. V., Novozhilov A. S. Origin and Evolution of the Universal Genetic Code. (англ.) // Annual Review Of Genetics. — 2017. — 27 November (vol. 51). — P. 45—62. — doi:10.1146/annurev-genet-120116-024713. — PMID 28853922. [исправить]
- ↑ Hershberg R., Petrov D. A. Selection on codon bias. (англ.) // Annual review of genetics. — 2008. — Vol. 42. — P. 287—299. — doi:10.1146/annurev.genet.42.110807.091442. — PMID 18983258. [исправить]
- ↑ Behura S. K., Severson D. W. Codon usage bias: causative factors, quantification methods and genome-wide patterns: with emphasis on insect genomes. (англ.) // Biological reviews of the Cambridge Philosophical Society. — 2013. — Vol. 88, no. 1. — P. 49—61. — doi:10.1111/j.1469-185X.2012.00242.x. — PMID 22889422. [исправить]
- ↑ Shields D. C., Sharp P. M. Synonymous codon usage in Bacillus subtilis reflects both translational selection and mutational biases. (англ.) // Nucleic acids research. — 1987. — Vol. 15, no. 19. — P. 8023—8040. — PMID 3118331. [исправить]
- ↑ Shields D. C., Sharp P. M., Higgins D. G., Wright F. “Silent” sites in Drosophila genes are not neutral: evidence of selection among synonymous codons. (англ.) // Molecular biology and evolution. — 1988. — Vol. 5, no. 6. — P. 704—716. — PMID 3146682. [исправить]
- ↑ Chen S. L., Lee W., Hottes A. K., Shapiro L., McAdams H. H. Codon usage between genomes is constrained by genome-wide mutational processes. (англ.) // Proceedings of the National Academy of Sciences of the United States of America. — 2004. — Vol. 101, no. 10. — P. 3480—3485. — doi:10.1073/pnas.0307827100. — PMID 14990797. [исправить]
- ↑ Akashi H. Synonymous codon usage in Drosophila melanogaster: natural selection and translational accuracy. (англ.) // Genetics. — 1994. — Vol. 136, no. 3. — P. 927—935. — PMID 8005445. [исправить]
- ↑ Sharp P. M., Bailes E., Grocock R. J., Peden J. F., Sockett R. E. Variation in the strength of selected codon usage bias among bacteria. (англ.) // Nucleic acids research. — 2005. — Vol. 33, no. 4. — P. 1141—1153. — doi:10.1093/nar/gki242. — PMID 15728743. [исправить]
- ↑ Ribas de Pouplana L., Turner R. J., Steer B. A., Schimmel P. Genetic code origins: tRNAs older than their synthetases? (англ.) // Proceedings Of The National Academy Of Sciences Of The United States Of America. — 1998. — 15 September (vol. 95, no. 19). — P. 11295—11300. — PMID 9736730. [исправить]
- ↑ Russell J. Garwood. Patterns In Palaeontology: The first 3 billion years of evolution (англ.) // Palaeontology Online : journal. — 2012. — Vol. 2, no. 11. — P. 1—14. Архивировано 26 июня 2015 года.
- ↑ Wächtershäuser Günter. Towards a Reconstruction of Ancestral Genomes by Gene Cluster Alignment (англ.) // Systematic and Applied Microbiology. — 1998. — December (vol. 21, no. 4). — P. 473—477. — ISSN 0723-2020. — doi:10.1016/S0723-2020(98)80058-1. [исправить]
- ↑ Gregory, Michael What is Life? Clinton College. Архивировано 13 декабря 2007 года.
- ↑ Pace N. R. The universal nature of biochemistry. (англ.) // Proceedings of the National Academy of Sciences of the United States of America. — 2001. — Vol. 98, no. 3. — P. 805—808. — doi:10.1073/pnas.98.3.805. — PMID 11158550. [исправить]
- ↑ Wächtershäuser G. From pre-cells to Eukarya–a tale of two lipids. (англ.) // Molecular microbiology. — 2003. — Vol. 47, no. 1. — P. 13—22. — PMID 12492850. [исправить]
- ↑ Yarus, Michael. [[5] в «Книгах Google» Life from an RNA World: The Ancestor Within] (англ.). — Harvard University Press, 2010. — ISBN 978-0-674-05075-4.
- ↑ Mathematica function for # possible arrangements of items in bins? – Online Technical Discussion Groups—Wolfram Community (англ.). community.wolfram.com. Дата обращения: 3 февраля 2017. Архивировано 5 февраля 2017 года.
- ↑ Freeland S. J., Hurst L. D. The genetic code is one in a million. (англ.) // Journal Of Molecular Evolution. — 1998. — September (vol. 47, no. 3). — P. 238—248. — PMID 9732450. [исправить]
- ↑ Taylor F. J., Coates D. The code within the codons. (англ.) // Bio Systems. — 1989. — Vol. 22, no. 3. — P. 177—187. — PMID 2650752. [исправить]
- ↑ Di Giulio M. The extension reached by the minimization of the polarity distances during the evolution of the genetic code. (англ.) // Journal Of Molecular Evolution. — 1989. — October (vol. 29, no. 4). — P. 288—293. — PMID 2514270. [исправить]
- ↑ Wong J. T. Role of minimization of chemical distances between amino acids in the evolution of the genetic code. (англ.) // Proceedings Of The National Academy Of Sciences Of The United States Of America. — 1980. — February (vol. 77, no. 2). — P. 1083—1086. — PMID 6928661. [исправить]
- ↑ Erives A. A model of proto-anti-codon RNA enzymes requiring L-amino acid homochirality. (англ.) // Journal Of Molecular Evolution. — 2011. — August (vol. 73, no. 1-2). — P. 10—22. — doi:10.1007/s00239-011-9453-4. — PMID 21779963. [исправить]
- ↑ 1 2 Freeland S. J., Knight R. D., Landweber L. F., Hurst L. D. Early fixation of an optimal genetic code. (англ.) // Molecular Biology And Evolution. — 2000. — April (vol. 17, no. 4). — P. 511—518. — doi:10.1093/oxfordjournals.molbev.a026331. — PMID 10742043. [исправить]
- ↑ Crick F. H. The origin of the genetic code. (англ.) // Journal Of Molecular Biology. — 1968. — December (vol. 38, no. 3). — P. 367—379. — PMID 4887876. [исправить]
- ↑ Hopfield J. J. Origin of the genetic code: a testable hypothesis based on tRNA structure, sequence, and kinetic proofreading. (англ.) // Proceedings Of The National Academy Of Sciences Of The United States Of America. — 1978. — September (vol. 75, no. 9). — P. 4334—4338. — PMID 279919. [исправить]
Литература[править | править код]
- Альбертс Б. Молекулярная биология клетки. — М. — Ижевск: НИЦ «Регулярная и хаотическая динамика», Институт компьютерных исследований, 2013. — Т. 1. — С. 586. — 808 с. — ISBN 978-5-4344-0112-8.
- Кребс Дж., Голдштейн Э., Килпатрик С. Гены по Льюину. — М.: Лаборатория знаний, 2017. — 919 с. — ISBN 978-5-906828-24-8.